Hepatitis-B-Virus
| Hepatitis-B-Virus | ||||||||
|---|---|---|---|---|---|---|---|---|

3D-Rekonstruktion | ||||||||
| Systematik | ||||||||
| Taxonomische Merkmale | ||||||||
| ||||||||
| Wissenschaftlicher Name | ||||||||
| Hepatitis B virus | ||||||||
| Kurzbezeichnung | ||||||||
| HBV | ||||||||
| Links | ||||||||
|

Das Hepatitis-B-Virus (HBV) ist ein behülltes DNA-Virus mit überwiegend doppelsträngigem Genom und der Auslöser der Hepatitis B.[2] Das Hepatitis-B-Virus entstammt der Gattung Orthohepadnavirus innerhalb der Familie der Hepadnaviridae.[3]
Erkrankung
[Bearbeiten | Quelltext bearbeiten]HBV vermehrt sich nahezu ausschließlich in Leberzellen. Es ist zwar selbst nicht zytopathogen und verursacht daher keinen zytopathischen Effekt; so verlieren Leberzellen nicht ihre Funktion, auch wenn sie sehr viele Viren herstellen. Jedoch verursacht die Immunreaktion eine zunehmende Schädigung der Hepatozyten. Eine zu starke Reaktion kann eine fulminante Hepatitis zur Folge haben. Zusätzlich zur Hepatitis B verursacht HBV in chronischen Verläufen (bei einer zu schwachen Immunreaktion) durch die fortlaufende Entzündung eine Leberfibrose und dann Leberzirrhose.[4] Als Folge der Zirrhose kann Leberkrebs entstehen. Durch die kontinuierliche Wachstumsstimulation zum Ersetzen der durch das Immunsystem zerstörten infizierten Hepatozyten und durch die Zytokine mit teilweise wachstumsfördernden Eigenschaften und durch die teilweise Insertion in die zelluläre DNA entstehen vermehrt Kopierfehler in der DNA während der Mitose (in der S-Phase), was ebenfalls die Entstehung von Tumoren begünstigt.[5][6][7]
Möglicherweise existiert auch ein Zusammenhang mit anderen Krebsarten wie Pankreaskrebs.[8][9]
Aufbau
[Bearbeiten | Quelltext bearbeiten]
Das Virion besitzt eine Virushülle und ein 28 nm großes, ikosaedrisches Kapsid. Das Kapsid ist aus 180 Kopien des HBV-Kernproteins (auch hepatitis B core antigen HBcAg) aufgebaut und umschließt die virale DNA und eine DNA-abhängige DNA-Polymerase mit einer Reverse-Transkriptase-Aktivität (RT), ähnlich einer retroviralen Polymerase.[10] Die Virushülle enthält als Membranprotein das HBsAg und das Pre-S1-Protein zur Bindung an die Wirtszelle.[5] Mit einem Durchmesser von 42 nm gehört das sphärische HBV (auch Dane particle genannt) eher zu den kleineren Tierviren.[11]
Es existieren auch nicht-infektiöse filamentöse Formen ohne Kapsid von 22 nm Durchmesser als Sphäre bzw. auch als Röhrenform, da HBsAg im Überschuss in der Leber Infizierter gebildet wird und im Blut zirkulieren.[11][4] Diese Virosomen bestehen aus HBsAg in einer Lipiddoppelschicht.[12] Aufgrund der Immunogenität ohne Infektiosität wurden sie bei der ersten Generation von Hepatitis-B-Impfstoffen als sogenannte Plasmaimpfstoffe verwendet.[4] Mittlerweile werden HBV-Impfstoffe gentechnisch erzeugt. Das HBcAg kann bei rekombinanter Expression (ohne HBsAg) ebenfalls Virosomen bilden.[13]
HBV erreicht im Blut eine Viruskonzentration zwischen 104 und 109 Virionen pro Milliliter, die Virosomen aus HBsAg erreichen sogar Partikelkonzentrationen bis zu 1012 Partikel pro Milliliter.[5]
Genom
[Bearbeiten | Quelltext bearbeiten]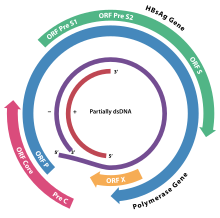
Das Genom des HBV besteht aus zirkulär-geschlossener DNA, jedoch ist die DNA nicht vollständig doppelsträngig und ein Strang durchgängig.[11] Ein Ende des durchgängigen Strangs von circa 3020–3320 Nukleotide bindet die virale DNA-Polymerase, während der kürzere Strang mit 1700 bis 2800 Nukleotiden deutlich kürzer ist.[14]
Der durchgängige (−)-Strang (nicht-codierend) ist revers-komplementär zur viralen mRNA. Die virale DNA ist bereits kurz nach der Infektion im Zellkern nachweisbar. Die teilweise doppelsträngige DNA wird am (+)-Strang vervollständigt und eine RNA-Sequenz am (+)-Strang entfernt. Zuletzt werden überhängende Nukleotide entfernt und der (−)-Strang zum Ring geschlossen.
Die vier Gene des HBV werden als C (core), X, P (polymerase) und S (surface) bezeichnet. Das core-Protein HBcAg wird aus einem Präprotein durch Proteolyse erzeugt. Das Gen S des Proteins HBsAg enthält drei Leseraster-konforme Start-Codons (AUG), wodurch drei Proteine entstehen, pre-S1 (synonym L), pre-S2 (synonym M) und S.[15]
Verschiedene nicht-codierende RNA wurden im Genom des HBV identifiziert. Diese werden als Hepatitis B virus PRE alpha, Hepatitis B virus PRE beta und HBV RNA encapsidation signal epsilon bezeichnet.[16][17]


Proteine
[Bearbeiten | Quelltext bearbeiten]Das Virion des HBV besteht aus vier strukturellen Proteinen, weiterhin kommt das lösliche HBeAg außerhalb des Virions vor.
HBsAg
[Bearbeiten | Quelltext bearbeiten]
HBsAg (englisch HBV surface antigen ‚HBV-Oberflächenantigen‘) ist ein Oberflächenprotein des Virions, das im Zuge der Immunreaktion von Antikörpern als Antigen erkannt wird. Es ist durch eine virologische Diagnostik im Blut von Infizierten nachweisbar und zeigt an, ab wann ein Patient nicht mehr ansteckend ist. In der Histologie wird HBsAg durch eine Shikata-Orceinfärbung nachgewiesen.[18]
Historisch wurde HBsAg zuerst als Australia Antigen bekannt, da es zuerst von Baruch Samuel Blumberg im Blutserum von australischen Aborigines entdeckt wurde.[19] Alfred Prince entdeckte 1968 den Zusammenhang mit einer Hepatitisform. Der erste Hepatitis-B-Virus-Impfstoff (Heptavax) wurde aus dem Blutplasma von HBV-Infizierten gereinigt, aufgrund des Kontaminationsrisikos durch andere Pathogene wurde später auf rekombinante Proteine aus der Bäckerhefe umgestellt.
Das Hepatitis-D-Virus benötigt HBsAg, um pathogen zu werden.[20]
HBcAg
[Bearbeiten | Quelltext bearbeiten]HBcAg (englisch core antigen ‚Antigen des Viruskerns‘) dient der Verpackung der viralen DNA und wird zum Nachweis einer momentanen Infektion verwendet. Es ist das Kapsidprotein des HBV.
HBeAg
[Bearbeiten | Quelltext bearbeiten]HBeAg (englisch extracellular antigen ‚extrazelluläres Antigen‘) ist eine Spleißvariante aus den offenen Leserastern core-ORF und pre-C-ORF. HBeAg wird sezerniert und kommt nicht im Virion vor. HBeAg wird ebenso wie HBcAg zum Nachweis einer momentanen Infektion eingesetzt. Vermutlich dient es der Minderung der Genexpression von Toll-like Rezeptor 2 auf Hepatozyten und Monozyten und somit einer Dämpfung der Immunantwort gegen HBV. HBeAg ist nicht essentiell, d. h. Virionen ohne eine HBeAg-Expression sind infektiös und pathogen.[21]
Hepatitis-B-Virus DNA-Polymerase
[Bearbeiten | Quelltext bearbeiten]Die DNA-Polymerase des HBV dient der Replikation der viralen Genoms. Lamivudine und Penciclovir hemmen bei nichtresistenten HBV-Stämmen die Polymerase.[22][23]
HBx
[Bearbeiten | Quelltext bearbeiten]Das X-Protein HBx ist ein kleiner transkriptioneller Transaktivator, der die zelluläre Transkription von HBV-Genen reguliert.[11] Darüber hinaus ändert er den Zellzyklus und bildet einen Proteinkomplex mit dem zellulären Protein HBX interacting protein (HBXIP), der mit dem Spindelapparat während der Zellteilung interagiert.[24]
Möglicherweise entstammt das X-Protein einer DNA-Glykosylase.[25] HBx erhöht die Ausschüttung von Calciumionen aus den Mitochondrien.[26][27] Weiterhin interagiert HBx mit Damaged DNA Binding Protein 1 (DDB1) und hemmt die CUL4-DDB1-E3-Komplexe und dadurch das Ubiquitin-Proteasom-System.[28] HBx fördert die Zellteilung und fördert dadurch die Entstehung von Tumoren.[29] HBx hemmt die Protein-Arginin-Methyltransferase PRMT1, was die Replikation des HBV verstärkt.[30]
Vorkommen und Genotypen
[Bearbeiten | Quelltext bearbeiten]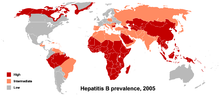
HBV werden in vier Serotypen (adr, adw, ayr, ayw) eingeteilt, je nach den antigenen Epitopen auf ihren Hüllproteinen, genetisch werden sie, je nach ihren Mutationen, 9–10 Genotypen (A–J) zugeordnet. Die einzelnen Genotypen haben eine unterschiedliche geografische Verteilung und werden zur epidemiologischen Bestimmung der Transmission und der viralen Evolution herangezogen. Die unterschiedlichen Genotypen weisen auch Unterschiede in den Krankheitsverläufen, der Häufigkeit von Komplikationen sowie in der Therapie- und der Impfstoffwahl auf.[31][32]
Der Reservoirwirt des HBV ist hauptsächlich der Mensch, daneben kommt HBV auch in Menschenaffen und Gibbons vor.[33] Die mindestens neun bekannten Genotypen des HBV werden A bis I benannt.[34][35] Möglicherweise existiert auch ein Genotyp J, der in auf den Ryūkyū-Inseln in Japan identifiziert wurde.[36] Die unterschiedlichen Genotypen sprechen unterschiedlich auf Therapien an.[37]
Die Genotypen unterscheiden sich mindestens um acht Prozent auf der Genomebene und haben unterschiedliche geographische Häufigkeiten. Der Genotyp F unterscheidet sich von den anderen am stärksten, mit vierzehn Prozent Unterschied. Der Genotyp A ist prävalent in Europa, Afrika und Südasien, einschließlich der Philippinen. Die Genotypen B und C sind vorherrschend in Asien; der Genotyp D tritt im Mittelmeerraum, dem Nahen Osten und Indien auf. Der Genotyp E existiert in Afrika südlich der Sahara. Der Genotyp F (oder H) kommt in Zentral- und Südamerika vor. Der Genotyp G wurde in Frankreich und Deutschland gefunden. Die Genotypen A, D und F dominieren in Brasilien und alle Genotypen kommen mit ethnischen Häufungen in den Vereinigten Staaten vor.
Innerhalb der Genotypen (außer E und G)[35] sind Subtypen beschrieben, die in ihrer DNA-Sequenz untereinander um vier bis acht Prozent abweichen.
- Genotyp A hat zwei Subtypen: Aa (A1) in Afrika/Asien und Ae (A2) in Europa/USA. Letzterer tritt auch bei mittels Hefezellen gewonnenen Impfstoffen auf, der auch vor Infektionen anderer Genotypen schützt.[4]
- Genotyp B besitzt zwei geographisch getrennte Subtypen: Bj/B1 ('j' — Japan) und Ba/B2 ('a' — Asien). Der Genotyp Ba wird weiter in vier clades unterteilt (B2 — B4).
- Genotyp C hat ebenso zwei geographisch getrennte Subtypen: Cs (C1) in Südostasien und Ce (C2) in Ostasien. Der C-Subtyp wird in fünf clades unterteilt, während ein möglicher Sechster (C6) und ein Siebter (C7) bisher nur in einem Isolat in den Philippinen und auf West-Papua gefunden wurden.[38][39] Der Genotyp C1 kommt in Vietnam, Myanmar und Thailand vor. Der Genotyp C2 tritt in Japan, Korea und China auf. Der Genotyp C3 wird in Neukaledonien und Polynesien gefunden. Der Genotyp C4 kommt in Australien vor und C5 auf den Philippinen.
- Genotyp D wird in sieben Subtypen unterteilt (D1 — D7). Er ist in Afrika, Europa, im Mittelmeerraum und Indien verbreitet.[36]
- Genotyp E hat nur einen Subtyp, er tritt in West- und Südafrika auf.[36]
- Genotyp F (dominant in Süd- und Mittelamerika)[36] ist in vier Subtypen unterteilt (F1 — F4). Der Subtyp F1 wird weiter in die clades 1a und 1b getrennt. In Venezuela werden die Subtypen F1, F2 und F3 im Westen und Osten gefunden, während F3 im Süden alleinig auftritt. Der Subtyp Ia kommt in Mittelamerika, der Subtyp III im nördlichen Südamerika und IV im südlichen Südamerika vor. Das clade Ib wird auf beinahe dem ganzen amerikanischen Kontinent (außer dem nördlichen Südamerika) und das clade II wird in Zentral- und Südamerika gefunden.
- Genotyp G hat nur einen Subtyp, er wurde in Frankreich und den USA beobachtet.[36]
- Genotyp H tritt in Mexiko und Zentralamerika auf.[36]
- Genotyp I wurde in Vietnam und Laos identifiziert.[36]
HBV ist ein typischer Vertreter der Gattung der Orthohepadnaviren, die drei weitere Vertreter aufweist: das Ground squirrel hepatitis virus, das Woodchuck hepatitis virus, und das Woolly monkey hepatitis B virus. Die Familie der Hepadnaviridae enthält noch zwei weitere Gattungen, die Avihepadnaviren und eine noch zu Benennende. Die Familie ist noch keiner Ordnung zugeordnet.[40] HBV-ähnliche Viren wurden bereits in allen Altweltaffen gefunden (Orang-Utans, Gibbons, Gorillas und Schimpansen) und auch in Neuweltaffen wie dem Wollaffen nachgewiesen, was einen älteren Ursprung dieses Virus in Primaten nahelegt.
Replikationszyklus
[Bearbeiten | Quelltext bearbeiten]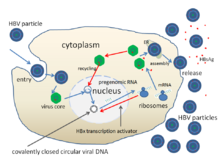
Der Transport von HBV in die Leber erfolgt über Adsorption an Apolipoprotein E in Lipoprotein-Remnants.[41]
Der Replikationszyklus des HBV in Leberzellen:
- Niedrig affine Adsorption (engl. adsorption) an die Zellmembran über Glykosaminoglykane (Heparansulfat) durch HBsAg und Pre-S1-Protein, dann Bindung von Pre-S1 an NTCP (Na+-taurocholate cotransporting polypeptide)[41][42] und anschließende Clathrin-vermittelte Endozytose über Bindung des HBV-NTCP-Komplexes an den EGF-Rezeptor.[41]
- Penetration (engl. penetration oder uncoating) durch eine induzierte Fusion der Virushülle und der Endosomenmembran, wodurch das core mit der DNA des Virus ins Zytosol freigesetzt wird.
- Entpacken (engl. uncoating) und Transport in den Zellkern über Chaperone. Dadurch erfolgt die Freisetzung der DNA aus dem core und anschließend das Vervollständigen des (−)-Strangs und der Ringschluss zur cccDNA, welche als Vorlage für die Transkription der vier viralen mRNA (Pre-S1/L, Pre-S2/M, S und X) und der beiden zur Expression und Genomsynthese verwendeten RNA (C/P und Pre-C) dient. Alle viralen RNA tragen eine Cap-Struktur und einen Poly-A-Schwanz.[5]
- Replikation (engl. replication) im Zytosol, bei der die längste RNA (C/P, länger als das virale DNA-Genom) als Vorlage für die Synthese des viralen DNA-Genoms dient, sowie zur Proteinbiosynthese des Kapsids und der viralen Polymerase. Die RNA-Zwischenstufe erhöht die Mutationsrate zur Erzeugung von Fluchtmutationen. Die cccDNA wird bis zum Zeitpunkt der Synthese der Hüllproteine HBcAg und HBsAg (nach der Replikation) in den Zellkern importiert, erst durch die Hüllproteine erfolgt ein Export der cccDNA aus dem Zellkern. Durch den Kernimport des Genoms entsteht durch die Rückstellung unverpackter viraler Genome im Zellkern die gelegentlich auftretende persistente Infektion.[43][5]
- Zusammenbau (engl. assembly) der neuen Virionen, auf den eine Abschnürung aus der Zelle oder ein Rücktransport zum Zellkern folgt, wo die DNA vom HBcAg getrennt wird und eine erneute Genomsynthese beginnt.[15][44]
- Freisetzung (engl. budding, release) der Tochtervirionen durch Exozytose (nicht-lytisch) bei gleichzeitiger Synthese des DNA-Genoms im Zytoplasma aus der längsten mRNA durch die RT-Aktivität der viralen Polymerase.
Inhibitoren
[Bearbeiten | Quelltext bearbeiten]Synthetic anti-lipopolysaccharide peptides (SALPs) hemmen die Bindung von HBV an Heparansulfat.[41] Myrcludex B, Ezetimibe, Cyclosporin A und -Derivate, Ibersartan, Ritonavir, Rosiglitazon, Zafirlukast, Triax, Sulfasalazin, Troglitazon und Vanitaracin A hemmen die Bindung von HBV an den Rezeptor NTCP.[41] Gefitinib und Erlotinib hemmen die Bindung des HBV-NTCP-Komplexes an EGFR.[41] Pitstop-2 und Dynasore hemmen die Clathrin-vermittelte Endozytose des HBV-NTCP-EGFR-Komplexes.[41] Lamivudine und Penciclovir hemmen bei nichtresistenten HBV-Stämmen die Polymerase.[22][23]
Umweltstabilität
[Bearbeiten | Quelltext bearbeiten]Als behülltes Virus ist das Hepatitis-B-Virus empfindlich gegenüber chemischen oder physikalischen Desinfektionsverfahren, wird jedoch aufgrund des hohen Proteinanteils und eines niedrigen Lipidanteils in der Virushülle im Vergleich zu anderen behüllten Viren durch Alkohole oder Tenside nur relativ langsam inaktiviert. Die Inaktivierung von HBV durch Desinfektionsverfahren kann mit dem MAD-Test bestimmt werden.[45]
Meldepflicht
[Bearbeiten | Quelltext bearbeiten]In Deutschland ist jeder direkte oder indirekte Nachweis vom Hepatitis-B-Virus namentlich meldepflichtig nach § 7 des Infektionsschutzgesetzes.
In der Schweiz ist der positive laboranalytische Befund zu einem Hepatitis-B-Virus meldepflichtig und zwar nach dem Epidemiengesetz (EpG) in Verbindung mit der Epidemienverordnung und Anhang 3 der Verordnung des EDI über die Meldung von Beobachtungen übertragbarer Krankheiten des Menschen.
Geschichte
[Bearbeiten | Quelltext bearbeiten]Die genaue Bestimmung der frühen Evolution des HBV ist problematisch. Die Aufspaltung der Hepadnaviren in Ortho- und Avihepadnaviren erfolgte vor ungefähr 125.000 Jahren (95 % Intervall 78,297–313,500).[25] Sowohl Avi- als auch Orthohepadnaviren begannen etwa vor 25.000 Jahren, sich stärker zu verändern.[25] Zu dieser Zeit erfolgte die Aufspaltung in die acht Genotype. Humane Hepadnaviren trennten sich etwa vor 7.000 (95 % Intervall: 5,287–9,270) bis 10.000 Jahren (95 % Intervall: 6,305–16,681). Den Avihepadnaviren fehlt das X-Protein, jedoch sind noch Reste im Entenhepadnavirus zu finden.[46] Die Rate nicht-synonymer Mutationen des HBV wurde auf etwa 2×10−5 Aminosäuresubstitutionen pro Stelle pro Jahr geschätzt.[47] Die mittlere Häufigkeit von Nukleotidsubstitutionen pro Stelle pro Jahr wurde mit etwa 7.9 × 10−5 beziffert. Eine andere Schätzung vermutet den letzten gemeinsamen Vorfahren der humanen HBV-Stämme vor 1.500 Jahren und eine Trennung von den Avihepadnaviren vor 6.000 Jahren bei einer Mutationsrate von etwa 10−6 Substitutionen pro Stelle pro Jahr.[48]
Die schriftliche Erwähnung des Hepatitis-B-Virus lässt sich bis ins 5. Jahrhundert v. Chr. zurückverfolgen und HBV wird sogar in babylonischen Tontafeln erwähnt. Später beschrieb Hippokrates von Kos eine Gelbsucht-Epidemie bei seinen Patienten, die durch eine Gelbfärbung der Haut und des Weißen der Augen gekennzeichnet war. Die Gelbsucht ist ein klinisches Anzeichen für eine Hepatitis-B-Virusinfektion.[49][50] Aufgrund der langen Zeitspanne (in Wochen) zwischen der Exposition gegenüber dem Erreger und dem Auftreten der Krankheit wurde die Gelbsucht jedoch erst im 20. Jahrhundert als Infektionskrankheit anerkannt.[51] Die ersten dokumentierten Fälle von Hepatitis-B-Virusinfektionen traten 1883 auf, nachdem ein Pockenimpfstoff, der menschliche Lymphe enthielt, einer Gruppe von Menschen verabreicht worden war.[51] Der Pockenimpfstoff wurde Werftarbeitern in Deutschland verabreicht, und die Arbeiter entwickelten später Hepatitis-Symptome.[51] Die Serumhepatitis, die heute als Hepatitis B bekannt ist, wurde häufig nach der Verwendung von kontaminierten Nadeln und Spritzen beobachtet. Diese kontaminierten Nadeln und Spritzen wurden nicht ordnungsgemäß gereinigt und/oder bei Patienten wiederverwendet.[52] 1943 wurde die Übertragung des Hepatitis-B-Virus über Blut noch deutlicher, als Paul Beeson Gelbsucht bei Patienten beschrieb, die gerade Bluttransfusionen erhalten hatten. Eine weitere Gelbsucht-Epidemie wurde 1942 bei Soldaten beobachtet, nachdem sie einen Gelbfieber-Impfstoff erhalten hatten.[52] Die Unterscheidung zwischen Hepatitis-A-Virus und Hepatitis-B-Virus wurde erst 1947 getroffen, als sie durch zahlreiche Studien an menschlichen Freiwilligen als zwei verschiedene filtrierbare Erreger anerkannt wurden.[52]
1965 wurde das „australische Antigen“ entdeckt und als das Oberflächenantigen des Hepatitis-B-Virus HBsAg identifiziert. Dies war einer der ersten Durchbrüche in den Bemühungen, die Pathologie der viralen Hepatitis zu verstehen, die bei HBV-Infizierten eine Gelbsucht auslöst. Sie ermöglichte es den Industrieländern, asymptomatische Träger des Hepatitis-B-Virus zuverlässig zu diagnostizieren, und die Entdeckung bot den Mitarbeitern in Gesundheitsberufen die Möglichkeit, Blut vor der Verabreichung von Bluttransfusionen auf Hep B zu untersuchen.[52][51]
Heute lässt sich eine Infektion mit dem Hepatitis-B-Virus leicht vermeiden, indem man sich mit einem der Hepatitis-B-Impfstoffe impfen lässt. Der aus Plasma gewonnene HepB-Impfstoff wurde 1981 zugelassen und 1986 durch den rekombinanten HepB-Impfstoff ersetzt. Engerix B wurde 1989 und Heplisav-B 2017 zugelassen.[53][54][51] Alle diese Impfstoffe bieten Schutz gegen HBV.
Literatur
[Bearbeiten | Quelltext bearbeiten]- S. Modrow, Dietrich Falke, U. Truyen: Molekulare Virologie. 2. Auflage. Spektrum, Heidelberg 2003, ISBN 3-8274-1086-X.
- D. M. Knipe, Peter M. Howley (Hrsg.): Fields Virology. 5. Auflage. Philadelphia 2007, ISBN 978-0-7817-6060-7.
Weblinks
[Bearbeiten | Quelltext bearbeiten]- Hepatitis B und D, RKI, 20. Juni 2016
- Yingpu Yu, Maximilian A. Kass, Mengyin Zhang, Noor Youssef, Catherine A. Freije, Kelly P. Brock, Lauren C. Aguado, Leon L. Seifert, Sanjana Venkittu, Xupeng Hong, Amir Shlomai, Ype P. de Jong, Debora S. Marks, Charles M. Rice, William M. Schneider: Deep mutational scanning of hepatitis B virus reveals a mechanism for cis-preferential reverse transcription. In: Cell, Band 187, Nr. 11, 8. Mai 2024, S. 2735-2745.e12,; doi:10.1016/j.cell.2024.04.008 (englisch). Dazu:
- Scientists May Have Discovered an Achilles Heel for Hepatitis B. Auf: SciTechDaily vom 28. Mai 2024.
Einzelnachweise
[Bearbeiten | Quelltext bearbeiten]- ↑ a b c d e ICTV: ICTV Taxonomy history: Hepatitis B virus, EC 51, Berlin, Germany, July 2019; Email ratification March 2020 (MSL #35)
- ↑ M. M. Hassan, D. Li, A. S. El-Deeb, R. A. Wolff, M. L. Bondy, M. Davila, J. L. Abbruzzese: Association between hepatitis B virus and pancreatic cancer. In: Journal of clinical oncology : official journal of the American Society of Clinical Oncology. Band 26, Nummer 28, Oktober 2008, S. 4557–4562. ISSN 1527-7755, doi:10.1200/JCO.2008.17.3526, PMID 18824707, PMC 2562875 (freier Volltext).
- ↑ Richard Hunt: Hepatitis viruses. ( vom 19. Juni 2014 im Internet Archive) University of Southern California, Department of Pathology and Microbiology
- ↑ a b c d Wolfgang Jilg: Hepatitis B. In: Heinz Spiess, Ulrich Heininger, Wolfgang Jilg (Hrsg.): Impfkompendium. 8. Auflage. Georg Thieme Verlag, 2015, ISBN 978-3-13-498908-3, S. 185 ff., doi:10.1055/b-0035-127585.
- ↑ a b c d e D. M. Knipe, Peter M. Howley (Hrsg.): Fields Virology. 5. Auflage. Philadelphia 2007, ISBN 978-0-7817-6060-7.
- ↑ F. V. Chisari, M. Isogawa, S. F. Wieland: Pathogenesis of hepatitis B virus infection. In: Pathologie-biologie. Band 58, Nummer 4, August 2010, S. 258–266, ISSN 1768-3114. doi:10.1016/j.patbio.2009.11.001. PMID 20116937. PMC 2888709 (freier Volltext).
- ↑ F. Guerrieri, L. Belloni, N. Pediconi, M. Levrero: Molecular mechanisms of HBV-associated hepatocarcinogenesis. In: Seminars in liver disease. Band 33, Nummer 2, Mai 2013, S. 147–156, ISSN 1098-8971. doi:10.1055/s-0033-1345721. PMID 23749671.
- ↑ Gina Gheorghe et al.: Risk Factors for Pancreatic Cancer: Emerging Role of Viral Hepatitis. In: Journal of Personalized Medicine. Band 12, Nr. 1, 10. Januar 2022, S. 83, doi:10.3390/jpm12010083, PMID 35055398, PMC 8780367 (freier Volltext).
- ↑ Felix Broecker, Karin Moelling: The Roles of the Virome in Cancer. In: Microorganisms. Band 9, Nr. 12, 8. Dezember 2021, S. 2538, doi:10.3390/microorganisms9122538, PMID 34946139, PMC 8706120 (freier Volltext).
- ↑ S. Locarnini: Molecular virology of hepatitis B virus. In: Seminars in liver disease. Band 24 Suppl 1, 2004, S. 3–10. ISSN 0272-8087, doi:10.1055/s-2004-828672, PMID 15192795.
- ↑ a b c d Pierre Van Damme et al.: Hepatitis B Vaccines. In: Stanley A. Plotkin et al. (Hrsg.): Plotkin’s Vaccines. 7. Auflage. Elsevier, Philadelphia 2017, ISBN 978-0-323-35761-6, S. 342 ff., doi:10.1016/B978-0-323-35761-6.00025-0.
- ↑ C. R. Howard: The biology of hepadnaviruses. In: The Journal of general virology. Band 67 ( Pt 7), Juli 1986, S. 1215–1235. ISSN 0022-1317, PMID 3014045.
- ↑ W. Fiers, M. De Filette, K. El Bakkouri, B. Schepens, K. Roose, M. Schotsaert, A. Birkett, X. Saelens: M2e-based universal influenza A vaccine. In: Vaccine. 27(45), 2009, S. 6280–6283. PMID 19840661.
- ↑ A. Kay, F. Zoulim: Hepatitis B virus genetic variability and evolution. In: Virus research. Band 127, Nummer 2, August 2007, S. 164–176 ISSN 0168-1702, doi:10.1016/j.virusres.2007.02.021, PMID 17383765.
- ↑ a b J. Beck, M. Nassal: Hepatitis B virus replication. In: World journal of gastroenterology : WJG. Band 13, Nummer 1, Januar 2007, S. 48–64. ISSN 1007-9327, PMID 17206754, PMC 4065876 (freier Volltext).
- ↑ G. J. Smith, J. E. Donello, R. Lück, G. Steger, T. J. Hope: The hepatitis B virus post-transcriptional regulatory element contains two conserved RNA stem-loops which are required for function. In: Nucleic acids research. Band 26, Nummer 21, November 1998, S. 4818–4827. ISSN 0305-1048, PMID 9776740, PMC 147918 (freier Volltext).
- ↑ S. Flodell, J. Schleucher, J. Cromsigt, H. Ippel, K. Kidd-Ljunggren, S. Wijmenga: The apical stem-loop of the hepatitis B virus encapsidation signal folds into a stable tri-loop with two underlying pyrimidine bulges. In: Nucleic acids research. Band 30, Nummer 21, November 2002, S. 4803–4811. ISSN 1362-4962, PMID 12409471, PMC 135823 (freier Volltext).
- ↑ P. Guarascio, F. Yentis, U. Cevikbas, B. Portmann, R. Williams: Value of copper-associated protein in diagnostic assessment of liver biopsy. In: Journal of clinical pathology. Band 36, Nummer 1, Januar 1983, S. 18–23. ISSN 0021-9746, PMID 6185545, PMC 498098 (freier Volltext).
- ↑ B. S. Blumberg, H. J. Alter, S. Visnich: A "New" antigen in leukemia sera. In: JAMA. Band 191, Februar 1965, S. 541–546. ISSN 0098-7484, PMID 14239025.
- ↑ N. Chai, H. E. Chang, E. Nicolas, Z. Han, M. Jarnik, J. Taylor: Properties of subviral particles of hepatitis B virus. In: Journal of virology. Band 82, Nummer 16, August 2008, S. 7812–7817. ISSN 1098-5514, doi:10.1128/JVI.00561-08, PMID 18524834, PMC 2519590 (freier Volltext).
- ↑ Mandell, Douglas, and Bennett's Principles and Practice of Infectious Diseases. 7. Auflage. Band 2, New York 2010, S. 2062.
- ↑ a b S. K. Ono-Nita, N. Kato, Y. Shiratori, T. Masaki, K. H. Lan, F. J. Carrilho, M. Omata: YMDD motif in hepatitis B virus DNA polymerase influences on replication and lamivudine resistance: A study by in vitro full-length viral DNA transfection. In: Hepatology (Baltimore, Md.). Band 29, Nummer 3, März 1999, S. 939–945. ISSN 0270-9139, doi:10.1002/hep.510290340. PMID 10051501.
- ↑ a b T. Shaw, S. S. Mok, S. A. Locarnini: Inhibition of hepatitis B virus DNA polymerase by enantiomers of penciclovir triphosphate and metabolic basis for selective inhibition of HBV replication by penciclovir. In: Hepatology (Baltimore, Md.). Band 24, Nummer 5, November 1996, S. 996–1002. ISSN 0270-9139, doi:10.1002/hep.510240504, PMID 8903366.
- ↑ Y. Wen, V. S. Golubkov, A. Y. Strongin, W. Jiang, J. C. Reed: Interaction of hepatitis B viral oncoprotein with cellular target HBXIP dysregulates centrosome dynamics and mitotic spindle formation. In: The Journal of biological chemistry. Band 283, Nummer 5, Februar 2008, S. 2793–2803. ISSN 0021-9258, doi:10.1074/jbc.M708419200. PMID 18032378.
- ↑ a b c F. J. van Hemert, M. A. van de Klundert, V. V. Lukashov, N. A. Kootstra, B. Berkhout, H. L. Zaaijer: Protein X of hepatitis B virus: origin and structure similarity with the central domain of DNA glycosylase. In: PloS one. Band 6, Nummer 8, 2011, S. e23392 ISSN 1932-6203, doi:10.1371/journal.pone.0023392. PMID 21850270, PMC 3153941 (freier Volltext).
- ↑ S. L. McClain, A. J. Clippinger, R. Lizzano, M. J. Bouchard: Hepatitis B virus replication is associated with an HBx-dependent mitochondrion-regulated increase in cytosolic calcium levels. In: Journal of virology. Band 81, Nummer 21, November 2007, S. 12061–12065. ISSN 0022-538X, doi:10.1128/JVI.00740-07. PMID 17699583, PMC 2168786 (freier Volltext).
- ↑ M. J. Bouchard, R. J. Puro, L. Wang, R. J. Schneider: Activation and inhibition of cellular calcium and tyrosine kinase signaling pathways identify targets of the HBx protein involved in hepatitis B virus replication. In: Journal of virology. Band 77, Nummer 14, Juli 2003, S. 7713–7719. ISSN 0022-538X, PMID 12829810, PMC 161925 (freier Volltext).
- ↑ T. Li, E. I. Robert, P. C. van Breugel, M. Strubin, N. Zheng: A promiscuous alpha-helical motif anchors viral hijackers and substrate receptors to the CUL4-DDB1 ubiquitin ligase machinery. In: Nature structural & molecular biology. Band 17, Nummer 1, Januar 2010, S. 105–111. ISSN 1545-9985, doi:10.1038/nsmb.1719, PMID 19966799, PMC 2823288 (freier Volltext).
- ↑ M. C. Kew: Hepatitis B virus x protein in the pathogenesis of hepatitis B virus-induced hepatocellular carcinoma. In: Journal of Gastroenterology and Hepatology. Band 26 Suppl 1, Januar 2011, S. 144–152. ISSN 1440-1746, doi:10.1111/j.1440-1746.2010.06546.x, PMID 21199526.
- ↑ S. Benhenda, A. Ducroux, L. Rivière, B. Sobhian, M. D. Ward, S. Dion, O. Hantz, U. Protzer, M. L. Michel, M. Benkirane, O. J. Semmes, M. A. Buendia, C. Neuveut: Methyltransferase PRMT1 is a binding partner of HBx and a negative regulator of hepatitis B virus transcription. In: Journal of virology. Band 87, Nummer 8, April 2013, S. 4360–4371. ISSN 1098-5514, doi:10.1128/JVI.02574-12, PMID 23388725, PMC 3624337 (freier Volltext).
- ↑ A. Kramvis, M. Kew, G. François: Hepatitis B virus genotypes. In: Vaccine. Band 23, Nummer 19, März 2005, S. 2409–2423. ISSN 0264-410X, doi:10.1016/j.vaccine.2004.10.045, PMID 15752827.
- ↑ L. O. Magnius, H. Norder: Subtypes, genotypes and molecular epidemiology of the hepatitis B virus as reflected by sequence variability of the S-gene. In: Intervirology. Band 38, Nummer 1–2, 1995, S. 24–34. ISSN 0300-5526, PMID 8666521
- ↑ P. Sa-Nguanmoo, P. Rianthavorn, S. Amornsawadwattana, Y. Poovorawan: Hepatitis B virus infection in non-human primates. In: Acta virologica. Band 53, Nummer 2, 2009, S. 73–82, ISSN 0001-723X. PMID 19537907.
- ↑ Hepatitis B und D. In: RKI. 20. Mai 2016, abgerufen am 26. April 2022.
- ↑ a b Luicer Anne Olubayo Ingasia et al.: Genotype E: The neglected genotype of hepatitis B virus. In: World Journal of Hepatology. Band 13, Nr. 12, 27. Dezember 2021, S. 1875–1891, doi:10.4254/wjh.v13.i12.1875, PMID 35069995, PMC 8727212 (freier Volltext).
- ↑ a b c d e f g Zhongping Liu et al.: Distribution of hepatitis B virus genotypes and subgenotypes: A meta-analysis. In: Medicine. Band 100, Nr. 50, 17. Dezember 2021, S. e27941, doi:10.1097/MD.0000000000027941, PMID 34918643, PMC 8678021 (freier Volltext).
- ↑ M. A. Mahtab, S. Rahman, M. Khan, F. Karim: Hepatitis B virus genotypes: an overview. In: Hepatobiliary & pancreatic diseases international : HBPD INT. Band 7, Nummer 5, Oktober 2008, S. 457–464. ISSN 1499-3872, PMID 18842489.
- ↑ L. Cavinta, J. Sun, A. May, J. Yin, M. von Meltzer, M. Radtke, N. G. Barzaga, G. Cao, S. Schaefer: A new isolate of hepatitis B virus from the Philippines possibly representing a new subgenotype C6. In: Journal of medical virology. Band 81, Nummer 6, Juni 2009, S. 983–987. ISSN 1096-9071, doi:10.1002/jmv.21475. PMID 19382274.
- ↑ M. I. Lusida, V. E. Nugrahaputra, Soetjipto, R. Handajani, M. Nagano-Fujii, M. Sasayama, T. Utsumi, H. Hotta: Novel subgenotypes of hepatitis B virus genotypes C and D in Papua, Indonesia. In: Journal of clinical microbiology. Band 46, Nummer 7, Juli 2008, S. 2160–2166. ISSN 1098-660X, doi:10.1128/JCM.01681-07, PMID 18463220, PMC 2446895 (freier Volltext).
- ↑ Mason, W.S., u. a.: 00.030. Hepadnaviridae. In: ICTVdB Index of Viruses. International Committee on Taxonomy of Viruses, 8. Juli 2008, abgerufen am 13. März 2009.
- ↑ a b c d e f g J. M. Wettengel, B. J. Burwitz: Innovative HBV Animal Models Based on the Entry Receptor NTCP. In: Viruses. Band 12, Nummer 8, Juli 2020, S. , doi:10.3390/v12080828, PMID 32751581, PMC 7472226 (freier Volltext).
- ↑ H. Yan, G. Zhong, G. Xu, W. He, Z. Jing, Z. Gao, Y. Huang, Y. Qi, B. Peng, H. Wang, L. Fu, M. Song, P. Chen, W. Gao, B. Ren, Y. Sun, T. Cai, X. Feng, J. Sui, W. Li: Sodium taurocholate cotransporting polypeptide is a functional receptor for human hepatitis B and D virus. In: eLife. Band 1, November 2012, S. e00049, doi:10.7554/eLife.00049, PMID 23150796, PMC 3485615 (freier Volltext).
- ↑ M. Levrero, T. Pollicino, J. Petersen, L. Belloni, G. Raimondo, M. Dandri: Control of cccDNA function in hepatitis B virus infection. In: Journal of hepatology. Band 51, Nummer 3, September 2009, S. 581–592, ISSN 1600-0641, doi:10.1016/j.jhep.2009.05.022, PMID 19616338.
- ↑ V. Bruss: Hepatitis B virus morphogenesis. In: World journal of gastroenterology : WJG. Band 13, Nummer 1, Januar 2007, S. 65–73. ISSN 1007-9327, PMID 17206755, PMC 4065877 (freier Volltext).
- ↑ O. Thraenhart, E. K. Kuwert, N. Scheiermann, R. Dermietzel, D. Paar, D. Maruhn, A. Alberti, H. J. Richter, J. Hotz: Comparison of the morphological alteration and disintegration test (MADT) and the chimpanzee infectivity test for determination of hepatitis B virucidal activity of chemical disinfectants. In: Zentralblatt für Bakteriologie, Mikrobiologie und Hygiene. 1. Abt. Originale B, Hygiene. Band 176, Nummer 5–6, 1982, S. 472–484, ISSN 0174-3015, PMID 7158127.
- ↑ B. Lin, D. A. Anderson: A vestigial X open reading frame in duck hepatitis B virus. In: Intervirology. Band 43, Nummer 3, 2000, S. 185–190 ISSN 0300-5526, doi:10.1159/000025037, PMID 11044813.
- ↑ C. Osiowy, E. Giles, Y. Tanaka, M. Mizokami, G. Y. Minuk: Molecular evolution of hepatitis B virus over 25 years. In: Journal of virology. Band 80, Nummer 21, November 2006, S. 10307–10314. ISSN 0022-538X, doi:10.1128/JVI.00996-06, PMID 17041211, PMC 1641782 (freier Volltext).
- ↑ Y. Zhou, E. C. Holmes: Bayesian estimates of the evolutionary rate and age of hepatitis B virus. In: J Mol Evol. 65(2), 2007, S. 197–205.
- ↑ T. Jake Liang: Hepatitis B: The virus and disease. In: Hepatology. 49. Jahrgang, S5, Mai 2009, S. S13–S21, doi:10.1002/hep.22881, PMID 19399811, PMC 2809016 (freier Volltext) – (englisch).
- ↑ VA.gov | Veterans Affairs. In: www.hepatitis.va.gov. Abgerufen am 4. Dezember 2022 (englisch).
- ↑ a b c d e Pinkbook: Hepatitis B | CDC. In: www.cdc.gov. 22. September 2022, abgerufen am 3. Dezember 2022 (amerikanisches Englisch).
- ↑ a b c d Timothy M. Block, Harvey J. Alter, W. Thomas London, Mike Bray: A historical perspective on the discovery and elucidation of the hepatitis B virus. In: Antiviral Research. 131. Jahrgang, 1. Juli 2016, ISSN 0166-3542, S. 109–123, doi:10.1016/j.antiviral.2016.04.012, PMID 27107897 (englisch, sciencedirect.com).
- ↑ Center for Biologics Evaluation and Research: ENGERIX-B. In: FDA. 3. Oktober 2019 (englisch, fda.gov).
- ↑ Immunization Schedules for Heplisav-B Vaccine | CDC. In: www.cdc.gov. 8. Juni 2022, abgerufen am 4. Dezember 2022 (amerikanisches Englisch).