Zellkern

1. Nucleolus (Kernkörperchen)
2. Zellkern (Nukleus)
3. Ribosomen
4. Vesikel
5. Raues (Granuläres) ER (Ergastoplasma)
6. Golgi-Apparat
7. Cytoskelett
8. Glattes (Agranuläres) ER
9. Mitochondrien
10. Lysosom
11. Cytoplasma (mit Cytosol und Cytoskelett)
12. Peroxisomen
13. Zentriolen
14. Zellmembran
Ein Zellkern oder Nukleus (lateinisch nucleus „Kern“) ist ein im Cytoplasma gelegenes, meist rundlich geformtes Organell der eukaryotischen Zelle, welches das Erbgut enthält. Mit nukleär oder karyo (altgriechisch κάρυον káryon „Kern“) wird ein Bezug auf den Zellkern ausgedrückt, das nukleäre Genom heißt (im Gegensatz zu dem in peripheren Organellen) beispielsweise auch Karyom. Die Wissenschaft vom Zellkern wird auch Karyologie genannt.
Beschreibung
[Bearbeiten | Quelltext bearbeiten]Der Zellkern ist das Hauptmerkmal zur Unterscheidung zwischen Eukaryoten (Lebewesen mit abgegrenztem Zellkern) und Prokaryoten (Lebewesen ohne abgegrenzten Zellkern, also Bakterien und Archaeen). Er enthält den größten Teil des genetischen Materials der eukaryotischen Zellen in Form von mehreren Chromosomen (Kern-DNA oder nukleäre DNA). Weitere Gene finden sich in den Mitochondrien und eher ausnahmsweise in Hydrogenosomen sowie bei Algen und Landpflanzen auch in Chloroplasten und anderen Plastiden. Die meisten Zellen enthalten genau einen Kern. Es gibt jedoch auch Ausnahmen (Syncytium); beispielsweise enthalten Myotuben, die durch Verschmelzung von Myoblasten entstehen, mehrere Kerne (polynukleäre Zellen). Der Kern selbst ist durch eine Kernhülle oder Kernmembran, die zum Schutz des Erbguts sowie der Regulierung des Stofftransports zwischen Nucleoplasma und Cytoplasma dient, vom sie umgebenden Cytoplasma abgetrennt. In Embryonen der Fruchtfliege teilen sich Kerne sehr schnell, ohne dass zunächst trennende Zellmembranen entstehen. Reife Erythrozyten der Säuger enthalten keinen Kern mehr, er wird während der Reifung abgestoßen.
Wichtige Vorgänge, die innerhalb des Zellkerns ablaufen, sind DNA-Replikation (die Duplizierung des in Form von DNA vorliegenden genetischen Materials) und Transkription (das Erstellen einer mRNA-Kopie eines gegebenen DNA-Abschnitts, der oft, aber nicht immer, einem Gen entspricht).
-
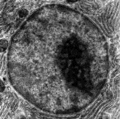
Elektronenmikroskopische Aufnahme des Zellkerns
-
Zellkern aus Medicago truncatula
-
Zwiebel (Allium cepa) Zellkern, 400×, jodgefärbt
Aufbau
[Bearbeiten | Quelltext bearbeiten]
2. Nucleolus
3. Karyoplasma
4. Chromatin 4a. Heterochromatin 4b. Euchromatin
5. Ribosomen
6. Kernporen
Der Zellkern, welcher bei Säugern typischerweise einen Durchmesser von 5 bis 16 µm hat, ist das im Mikroskop am leichtesten zu erkennende Organell der Zelle. Er wird durch die Kernhülle, bestehend aus zwei biologischen Membranen, der inneren und äußeren Kernmembran, begrenzt, welche die sogenannte perinukleäre Zisterne (Breite 10–15 nm, gefestigt von Mikrofilamenten – Dicke 2 bis 3 nm), umschließen. Die Gesamtdicke der Kernhülle beträgt etwa 35 nm. Die äußere Kernmembran geht fließend in das raue Endoplasmatische Retikulum (rER) über und hat wie dieses auch Ribosomen auf ihrer Oberfläche. Die innere Kernmembran grenzt an einen 20–100 nm breiten „Filz“, der Kernlamina (Lamina fibrosa nuclei), die aus Laminen, einer Gruppe von Intermediärfilamenten, besteht, welche den Zellkern stützt und die innere Membran vom Chromatin des Zellkerns trennt.
Zellkerne können je nach Zelltyp sehr unterschiedlich aussehen. Meistens sind sie kugelförmig oder oval. In einigen Zellen sehen sie eher geweihförmig aus. Manchmal kann der Zellkern in knotenartige Abschnitte untergliedert sein, so beim rosenkranzförmigen Zellkern der Trompetentierchen. Auch die Granulocyten der Säuger enthalten gelappte Kerne.
Durch die in der Kernhülle enthaltenen Kernporen, die ca. 25 % der Oberfläche bedecken, findet der aktive Stoffaustausch (z. B. rRNA oder mRNA) zwischen dem Kern und dem Zellplasma, gesteuert von einem Kernporenkomplex, statt. Regulatorische Proteine gelangen aus dem Cytoplasma in den Zellkern, Transkriptionsprodukte wie die mRNA werden zur Proteinsynthese, die an den Ribosomen des Cytoplasmas stattfindet, aus dem Kern in das Plasma exportiert. Die Flüssigkeit im Kern wird auch als Karyoplasma bezeichnet. Zellkerne können durch Anfärben der DNA lichtmikroskopisch hervorgehoben werden, z. B. durch die Feulgen-Färbung, durch Giemsa oder durch Fluoreszenzfarbstoffe wie DAPI.
Das Vorhandensein einer Kernmatrix wurde erstmals in den 1970er Jahren vorgeschlagen. Ihre Existenz ist jedoch weiterhin umstritten.
Das im Zellkern vorhandene Erbgut der Zelle befindet sich in den Chromosomen, d. h. mehreren zu Chromatin verpackten DNA-Fäden, die neben der DNA auch Proteine wie Histone enthalten. Neben den Histonen kommen auch andere Kernproteine, wie z. B. DNA-Polymerasen und RNA-Polymerasen, weitere Transkriptionsfaktoren sowie Ribonukleinsäuren im Kern vor.

Nukleärkörper
[Bearbeiten | Quelltext bearbeiten]Lichtmikroskopisch fallen in vielen Zellkernen ein oder mehrere rundliche Gebilde auf, die Kernkörperchen oder Nucleoli. Sie enthalten die Gene für ribosomale RNA (rRNA). Hier werden die Untereinheiten der Ribosomen gebildet, welche durch die Kernporen ins Cytoplasma gelangen. Nucleoli enthalten im Vergleich zum Rest des Kerns nur geringe Konzentrationen von DNA, stattdessen mehr RNA. Andere „Körperchen“ des Zellkerns lassen sich nur durch spezielle Färbetechniken darstellen, etwa durch Antikörperfärbung. Die Funktion dieser Körperchen ist meistens noch unbekannt. Hierunter fallen etwa „Speckles“ (Ansammlungen von Faktoren, die für Splicing benötigt werden), Cajal-Körper oder PML-Körper. Die räumliche Trennung der unterschiedlichen Komponenten bestimmter Kernkörperchen könnte die molekularen Wechselwirkungen in der extrem überfüllten Umgebung im Kern effizienter ermöglichen.[1]
| Vermutete Funktion[2][3][4] | Zentrale Komponenten | Typische Größe (μm) | Typische Anzahl | |
|---|---|---|---|---|
| Nucleolus | Herstellung von Ribosomen durch Transkription und Verarbeitung von rRNA und dem Aufbau ribosomaler Untereinheiten. Spielt eine Rolle bei der Modifikation und Herstellung anderer nukleärer RNAs und RNPs. Reguliert den Verlauf des Zellzyklus durch Sequestrierung und Modifizierung vieler Proteine. | RNA pol I | 3–8[2]
0,5–8[4] |
1–4 |
| Nuclear speckles (ICG) | Lagerung, Zusammenbau und Modifikation von Spleißfaktoren. | Prä-mRNA-Spleißfaktoren[2], SRSF2, SRSF1, Malat1[4] | 2–3[2]
0,5–1,8[4] |
20–50 |
| Nuclear stress bodies | Regulierung von Transkription und Spleißen unter Stress.
Enthält Satelliten III non-coding RNAs. Präzise Funktion noch nicht festgelegt. |
HSF1, HAP | 1–2[2]
0,3–3,0[4] |
2–6[2]
2–10[4] |
| Histone locus body (HLB) | Synthese von Histon-Genen | NPAT, FLASH,U7 snRNP | 0,2–1,2[2][4] | 2–4[2][4] |
| Cajal-Körper/Gems | Herstellung, Reifung und Recycling von snRNPs. Spielt auch eine Rolle beim Zusammenbau der Telomerase und der Regulierung der Telomerasenverlängerung. | Coilin, SMN | 0,2–1,5[2]
0,1–2,0[4] |
0–10 |
| PML-Körper | Regulierung der Genomstabilität, DNA-Reparatur, Kontrolle der Transkription, Virenabwehr | PML Protein | 0,1–1,0[2]
0,3–1,0[4] |
10–30 |
| Paraspeckles | mRNA-Regulation, RNA-Bearbeitung | NEAT1/MENε/βncRNAs PSP1, p54nrb/NONO | 0,2–1[2]
0,5[4] |
2–20[2]
10–20[4] |
| Perinukleares Kompartiment | Posttranskriptionelle Regulation einer Teilmenge von pol III RNAs in Tumor-Zellen. | PTB, CUGBP | 0,2–1,0 | 1–2[2]
1–4[4] |
| Polycomb (PcG)-Body | Polycomb Group (PcG)-Komplexe sind an der Genrepression durch epigenetische Modifikation des Chromatin und durch die Regulierung der Kernorganisation ihrer Zielgene beteiligt.[5] | Bmi1, Pc2[4] | 0,3–1,0[3] | 12–16[3] |
| OPT Domäne | Angereichert mit Transkriptionsfaktoren Oct1 und PTF. Teilweise Kolokalisation mit Transkriptionsstellen. Vorwiegend in der späten G1-Phase nachgewiesen. Zerfällt bei Transkriptionshemmung. | 1,0–1,5 | 1–3 | |
| Sam68 Kernkörperchen | Konzentriert Sam68 und Sam68-ähnliche Proteine SLM-1 und SLM-2. Zerfällt bei Transkriptionshemmung. Höchstwahrscheinlich angereichert mit RNA. | 0,6–1,0 | 1–5 | |
| Cleavage body | Angereichert mit den Spaltungsfaktoren CstF 64 kDa und CPSF 100 kDa und dem DEAD Boxprotein DDX1. Wird überwiegend in der S-Phase erkannt und ist nicht von der Transkriptionshemmung betroffen. | 0,2–1,0 | 1–4 | |
| Clastosomes | Diese Körper konzentrieren Proteinsubstrate für den proteasomalen Abbau. Vorwiegend bei stimulierter Aktivität des Proteasoms erkannt. Es zerfällt bei proteasomaler Hemmung.[3] Bildet sich als Reaktion auf Reize, die die proteasomabhängige Proteolyse aktivieren. | 19S, 20S Proteasome[4] | 0,2–1,2 | 0–3 |
| SUMO body | Angereichert mit SUMO-1 und SUMO-konjugierendem Enzym Ubc9. Konzentriert die Transkriptionsfaktoren pCREB, CBP, c-Jun. | 1–3 | 1–3 | |
| PIKA | unbekannt[6] (polymorphic interphase karyosomal association) |
Anordnung der Chromosomen
[Bearbeiten | Quelltext bearbeiten]
Chromosomen nehmen während der Interphase abgegrenzte Bereiche im Zellkern ein, die Chromosomenterritorien. Deren Existenz wurde zuerst von Carl Rabl (1885) und Theodor Boveri (1909) vorgeschlagen, der direkte Nachweis gelang erst 1985 mit Hilfe der Fluoreszenz-in-situ-Hybridisierung.[7][8]
Die Verteilung des Chromatins und somit der Chromosomen innerhalb des Zellkerns erscheint auf den ersten Blick zufällig: Die Anordnung der Chromosomen zueinander wechselt von Kern zu Kern, Nachbarn in einem können im nächsten weit auseinanderliegen. Seit den 1990er Jahren konnten jedoch einige Ordnungsprinzipien gefunden werden. Die DNA-Replikation erfolgt während der S-Phase nicht gleichmäßig, sondern an manchen Stellen der Chromosomen früher, an anderen später. Frühe oder späte Replikation sind dabei Eigenschaften, die für alle Abschnitte der Chromosomen in einem gegebenen Zelltyp konstant sind. Es stellte sich heraus, dass sich früh replizierte Bereiche vorwiegend im Inneren des Kerns befinden, während spät replizierte Bereiche vorwiegend an der Kernhülle und um die Nucleoli herum lokalisiert sind.[9] Für die Anordnung der Chromosomenterritorien im Zellkern wurde beobachtet, dass Chromosomen mit hoher Gendichte bevorzugt in der Mitte des Kerns liegen, während Chromosomen mit niedriger Gendichte häufiger an der Peripherie zu finden sind. Für manche Zelltypen wurde auch beschrieben, dass kleine Chromosomen eher in der Mitte liegen, während große außen sind.[10] Beide Motive sind dabei miteinander vereinbar.
Kernteilung
[Bearbeiten | Quelltext bearbeiten]Bei der Mitose und der Meiose, den bei eukaryotischen Zellen vorkommenden Arten der Kernteilung, verschwindet der Zellkern zeitweilig, weil die Kernhülle während des Teilungsvorgangs aufgelöst wird. Während Chromosomen in der Interphase keine lichtmikroskopisch sichtbaren Abgrenzungen ausbilden, kondensieren sie für die Kernteilung zu den kompakten Metaphase-Chromosomen. In dieser Transportform wird das Erbgut auf die Tochterzellen verteilt. Nach der Teilung bilden sich die Kernhüllen um die Chromosomen der Tochterzellen wieder aus und die Chromosomen dekondensieren wieder.
Forschungsgeschichte
[Bearbeiten | Quelltext bearbeiten]

Unten: Falschfarbendarstellung aller Chromosomenterritorien, die in dieser Fokusebene sichtbar sind, nach Computer-Klassifikation.
Der Zellkern ist das zuerst entdeckte Organell der Zelle. Die älteste erhaltene Zeichnung geht zurück auf den frühen Mikroskopiker Antoni van Leeuwenhoek (1632–1723). Dieser untersuchte rote Blutkörperchen des Lachses und beschrieb darin ein „Lumen“, den Zellkern.[11] Im Gegensatz zu den roten Blutkörperchen der Säugetiere haben jene der anderen Wirbeltiere Zellkerne. Eine weitere Erwähnung erfolgte 1804 durch Franz Andreas Bauer.[12] 1831 wurde der Zellkern vom schottischen Botaniker Robert Brown in einem Vortrag vor der Linnéschen Gesellschaft in London als „areola“ beschrieben.[13] Mögliche Bedeutungen erwähnte er nicht. Eine solche wurde zuerst 1838 von Matthias Schleiden vorgeschlagen, nämlich dass er eine Rolle bei der Bildung der Zelle spielt. Daher führte Schleiden den Namen „Cytoblast“ (Zellenbildner) ein. Er meinte beobachtet zu haben, dass sich neue Zellen an diesen Cytoblasten bildeten. Franz Julius Ferdinand Meyen widersprach entschieden der Ansicht, dass „der Zellenkern die Zelle selbst erzeuge“. Er hatte schon zuvor beschrieben, dass sich Zellen durch Teilung vermehren. Allerdings war Meyen auch der Ansicht, dass viele Zellen keine Zellkerne hätten. Überwunden wurde die Vorstellung einer Neubildung von Zellen erst durch die Arbeiten von Robert Remak (1852) und durch Rudolf Virchow (Omnis cellula e cellula, 1855), welcher die neue Lehre von der ausschließlichen Bildung von Zellen aus Zellen offensiv vertrat. Die Funktion des Zellkerns blieb weiter ungeklärt.
Christian Gottfried Ehrenberg hatte 1838 erstmals die Teilung eines Zellkerns beobachtet und auf dessen Rolle aufmerksam gemacht.[14] Bei Orchideen entdeckte 1842 Robert Brown den Zellkern.[15]
Von 1876 bis 1878 veröffentlichte Oscar Hertwig mehrere Studien über die Befruchtungsvorgänge des Seeigel-Eies, aus denen hervorging, dass der Zellkern des Spermiums in das Ei eindringt und dort mit dem Zellkern des Eies verschmilzt. Damit wurde zum ersten Mal behauptet, dass sich ein Individuum aus einer (einzelnen) kernhaltigen Zelle entwickele. Dies widersprach der von Ernst Haeckel vertretenen Ansicht, dass während der Embryonalentwicklung die gesamte Stammesgeschichte wiederholt würde, insbesondere auch die Entstehung der ersten kernhaltigen Zelle aus einer „Monerula“, einer strukturlosen Masse aus Urschleim. Die Notwendigkeit des Spermienkerns zur Befruchtung wurde daher noch lange Zeit kontrovers diskutiert. Hertwig bestätigte jedoch seine Befunde an anderen Tiergruppen, z. B. Amphibien und Mollusken. Eduard Strasburger kam an Pflanzen zum gleichen Ergebnis (1884). Dies ebnete den Weg, dem Kern eine wichtige Bedeutung bei der Vererbung zuzuweisen. Bereits 1873 hatte August Weismann die Gleichwertigkeit der mütterlichen und väterlichen Keimzellen für die Vererbung postuliert. Die Rolle des Kerns hierbei wurde erst später offenbar, nachdem die Mitose beschrieben wurde und Anfang des 20. Jahrhunderts die Mendel’schen Regeln wiederentdeckt wurden: Die Chromosomentheorie der Vererbung wurde entwickelt (siehe dort und Chromosom).
1874 isolierte Friedrich Miescher aus Zellkernen eine Substanz, die er als Nuclein bezeichnete (siehe Entdeckungsgeschichte im Artikel DNA). Nur wenige Jahre später kamen weitere Bestandteile wie Histone und Adenin (Albrecht Kossel) hinzu.[16] 1996 wurde mit der Backhefe (Saccharomyces cerevisiae), die zu den Pilzen gehört, das erste Genom eines Eukaryonten veröffentlicht. Es ist erst seit dem 21. Jahrhundert möglich, das Genom, das Transkriptom oder durch Massenspektrometrie auch das Proteom einer Zelle zu einem bestimmten Zeitpunkt zu bestimmen, um sich so ein umfassendes Bild dieses Systems zu machen.
Siehe auch
[Bearbeiten | Quelltext bearbeiten]| Übergeordnet |
|---|
| Organell |
| Untergeordnet |
| Kernhülle Kernlamina Kernmatrix Karyoplasma Proteinkomplexe |
| Gene Ontology |
| QuickGO |
Literatur
[Bearbeiten | Quelltext bearbeiten]- T. Cremer: Von der Zellenlehre zur Chromosomentheorie. Springer Verlag, Berlin u. a., 1985, ISBN 3-540-13987-7 (online) (zur Geschichte; PDF; 6,1 MB)
Weblinks
[Bearbeiten | Quelltext bearbeiten]- Literatur von und über Zellkern im Katalog der Deutschen Nationalbibliothek
- Elektronenmikroskopische Bilder von Zellkernen verschiedener Zelltypen
Einzelnachweise
[Bearbeiten | Quelltext bearbeiten]- ↑ Shinichi Nakagawa, Tomohiro Yamazaki, Tetsuro Hirose: Molecular dissection of nuclear paraspeckles: towards understanding the emerging world of the RNP milieu. In: Open Biology. Band 8, Nr. 10, Oktober 2018, ISSN 2046-2441, S. 180150, doi:10.1098/rsob.180150, PMID 30355755, PMC 6223218 (freier Volltext).
- ↑ a b c d e f g h i j k l m Miroslav Dundr, Tom Misteli: Biogenesis of Nuclear Bodies. In: Cold Spring Harbor Perspectives in Biology. Band 2, Nr. 12, Dezember 2010, ISSN 1943-0264, doi:10.1101/cshperspect.a000711, PMID 21068152, PMC 2982170 (freier Volltext).
- ↑ a b c d Maria Carmo-Fonseca, Maria T. Berciano, Miguel Lafarga: Orphan Nuclear Bodies. In: Cold Spring Harbor Perspectives in Biology. Band 2, Nr. 9, September 2010, ISSN 1943-0264, doi:10.1101/cshperspect.a000703, PMID 20610547, PMC 2926751 (freier Volltext).
- ↑ a b c d e f g h i j k l m n o Yuntao S. Mao, Bin Zhang, David L. Spector: Biogenesis and function of nuclear bodies. In: Trends in Genetics. Band 27, Nr. 8, August 2011, S. 295–306, doi:10.1016/j.tig.2011.05.006, PMID 21680045, PMC 3144265 (freier Volltext) – (elsevier.com [abgerufen am 2. Mai 2019]).
- ↑ Carmelo Ferrai, Inês Jesus de Castro, Liron Lavitas, Mita Chotalia, Ana Pombo: Gene Positioning. In: Cold Spring Harbor Perspectives in Biology. Band 2, Nr. 6, Juni 2010, ISSN 1943-0264, doi:10.1101/cshperspect.a000588, PMID 20484389, PMC 2869523 (freier Volltext).
- ↑ Compartmentalization within the nucleus: discovery of a novel subnuclear region. In: The Journal of Cell Biology. Band 115, Nr. 4, 2. November 1991, ISSN 0021-9525, S. 919–931, PMID 1955462, PMC 2289954 (freier Volltext).
- ↑ Schardin et al.: Specific staining of human chromosomes in Chinese hamster x man hybrid cell lines demonstrates interphase chromosome territories. Hum. Genet. 71: 281, 1985. PMID 2416668.
- ↑ Manuelidis: Individual interphase chromosome domains revealed by in situ hybridization. Hum. Genet. 71: 288, 1985. PMID 3908288.
- ↑ O'Keefeet al.: Dynamic organization of DNA replication in mammalian cell nuclei: spatially and temporally defined replication of chromosome-specific alpha-satellite DNA sequences. J. Cell Biol. 116: 1095, 1992. Zusammenfassung und Volltext beim Journal of Cell Biology (englisch).
- ↑ Cremer and Cremer: Chromosome territories, nuclear architecture and gene regulation in mammalian cells. Nat Rev Genet 2: 292, 2001. PMID 11283701.
- ↑ Antoni van Leeuwenhoek Opera Omnia, seu Arcana Naturae ope exactissimorum Microscopiorum detecta, experimentis variis comprobata, Epistolis ad varios illustres viros. J. Arnold et Delphis, A. Beman, Leiden 1719–1730. Zitiert nach Dieter Gerlach: Geschichte der Mikroskopie. Verlag Harry Deutsch, Frankfurt am Main 2009, ISBN 978-3-8171-1781-9.
- ↑ H. Harris: The Birth of the Cell. Yale University Press, New Haven 1999.
- ↑ Robert Brown: On the Organs and Mode of Fecundation of Orchidex and Asclepiadea. In: Miscellaneous Botanical Works. Band I, 1866, S. 511–514.
- ↑ Bärbel Häcker: Chromosomen. In: Werner E. Gerabek, Bernhard D. Haage, Gundolf Keil, Wolfgang Wegner (Hrsg.): Enzyklopädie Medizingeschichte. De Gruyter, Berlin / New York 2005, ISBN 3-11-015714-4, S. 261–262.
- ↑ Paul Diepgen, Heinz Goerke: Aschoff/Diepgen/Goerke: Kurze Übersichtstabelle zur Geschichte der Medizin. 7., neubearbeitete Auflage. Springer, Berlin/Göttingen/Heidelberg 1960, S. 35.
- ↑ W. Waldeyer: Ueber Karyokinese und ihre Beziehungen zu den Befruchtungsvorgängen. In: Archiv für mikroskopische Anatomie. Band 32, Nr. 1, 1888, S. 1–122, doi:10.1007/BF02956988 (PDF).