Alphaorpheovirus
| Alphaorpheovirus | ||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|

| ||||||||||||||||
| Systematik | ||||||||||||||||
| ||||||||||||||||
| Taxonomische Merkmale | ||||||||||||||||
| ||||||||||||||||
| Wissenschaftlicher Name | ||||||||||||||||
| Alphaorpheovirus | ||||||||||||||||
| Links | ||||||||||||||||
|
Alphaorpheovirus (ursprünglich vorgeschlagen als Orpheovirus) ist die Bezeichnung einer am 30. April 2024 vom International Committee on Taxonomy of Viruses (ICTV) offiziell anerkannten Gattung innerhalb der gleichzeitig neu errichteten Familie Orpheoviridae (Ordnung Pimascovirales) von Riesenviren des Phylums Negarnaviricota (ursprünglich bezeichnet als Nucleocytoplasmic large DNA viruses, NCLDV).
Mit diesem Stand besteht die Gattung aus einer einzigen Spezies, Alphaorpheovirus massiliense mit dem Referenzstamm Orpheovirus IHUMI-LCC2 alias Orpheovirus brasiliensis (OBRV).[2]
Wie alle diese Riesenviren haben die Viren der Gattung Alphaorpheovirus und damit alle Orpheoviridae ein Genom aus einer doppelsträngigen DNA (dsDNA). Die Erstbeschreibung eines Vertreters der Gattung wurde Anfang 2018 von Julien Andreani et al. veröffentlicht. Sie konnten das Virus Orpheovirus IHUMI-LCC2 (Spezies Alphaorpheovirus massiliense) aus Proben von Rattenkot unter Verwendung Amöben der Spezies Vermamoeba vermiformis (Tubulinea: Euamoebida/Tubulinida) isolieren (La Ciotat, Südfrankreich, westlich von Marseille).[3][4][5][6]
Aufbau
[Bearbeiten | Quelltext bearbeiten]

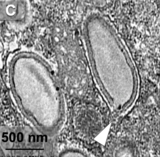
Die Viruspartikel (Virionen) sind wie die der Familien Pithoviridae und Cedratviridae von eiförmiger (ovoidaler) Gestalt und besitzen mindestens eine dicke Membran mit einer Öffnung (Pore, Ostiole) mit 70 bis 80 nm Durchmesser an der Spitze (Apex); eine oberflächliche Ähnlichkeit besteht darin auch zu den (nicht direkt verwandten) Pandoraviren. Durch die Pore wird die DNA auf die Wirtszelle übertragen. Bei den Pandoraviren besteht die Hülle (Tegument) jedoch aus drei Schichten von jeweils etwa 20 bis 25 nm. Die Viruspartikel der Gattung Alphaorpheovirus zeigten eine dunkle dichte äußere Schicht und sind zudem spärlich bedeckt mit kurzen Fibrillen. Unter dieser befindet sich ein Zwischenraum mittlerer Dichte, der nach innen begrenzt wird durch eine sehr dichte Membran (englisch hyperdense membrane). Die Länge der Virionen reicht von 900 bis 1.100 nm, einige Virionen könnten sogar bis 1.300 nm lang sein. Ihr Durchmesser beträgt etwa 500 nm.[3][6]
Genom
[Bearbeiten | Quelltext bearbeiten]Orpheovirus IHUMI-LCC2 besitzt ein ringförmiges Genom von 1.473.573 Basenpaaren mit einem GC-Gehalt von 25 %. Das Genom hat verschiedene Bereiche mit Wiederholungssequenzen (englisch repeats): 57 palindromische Sequenzen, sowie eine im Vergleich zu anderen Riesenviren sehr hohe Zahl von 1.527 Tandem-Repeats und 832 Kandidaten für invertierte Sequenzen (englisch inverted sequences). Mindestens 57,5 % der Gene von Orpheovirus IHUMI-LCC2 sind offene Leserahmen (englisch open reading frames, ORFs). Von 509 kodierten Proteinen gab es in 148 Fällen (12,3 %) beste Übereinstimmung mit Genen anderer Viren, in 176 Fällen mit eukaryotischen Genen (14,7 %) und in 183 Fällen (15,3 %) mit prokaryotischen Genen (von Bakterien oder Archaeen). Unter den Viren gab es beste Übereinstimmung mit Pithovirus sibericum (Spezies Alphapithovirus sibericum), Pithovirus massiliensis (Spezies Alphapithovirus massiliense), Vertretern der Gattung Alphacedratvirus (alle Ordnung Pimascovirales), sowie Vertretern der Gattung Mimivirus und der Unterfamilie Klosneuvirinae (alle Familie Mimiviridae). Es wurden keine Gene für tRNA (Transfer-RNA) gefunden.[3]
Interessanterweise wurden Gen-Homologe mit dem Sputnik-Virophagen und dem Zamilon-Virophagen, beide Gattung Sputnikvirus, gefunden.[3]
Vermehrungszyklus
[Bearbeiten | Quelltext bearbeiten]

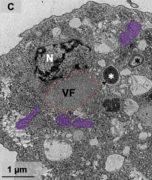
Der Eintritt der Virionen in den Wirt geschieht durch Phagocytose, wobei es den Virionen gelingt, dem darauf normalerweise folgenden phagosomalen Prozess (Verdauungsprozess) zu entkommen. Danach wird die Virus-DNA durch die ostiolartige Spitze ins Zytoplasma entlassen. Die für Riesenviren typischen Virusfabriken sind 14 bis 16 Stunden nach der Infektion gut zu erkennen. Das Bild ist in diesem frühen Stadium der Virussynthese ähnlich wie bei den Pithoviridae und den Cedratviridae. Die Wirtszellen sind dann 20 Stunden nach der Infektion mit neuen Virionen dicht gefüllt. Die Virionen scheinen durch Zellausbrüche (englisch bursts) oder Exocytose auszutreten und sind dann auch außerhalb der Amöbenzelle nachweisbar. Nach weiteren 4 bis 18 Stunden erfolgt vollständiger Zellausbruch.[3] Dieser relativ langsame Vermehrungszyklus ist typisch für den im Labor benutzten Wirt Vermamoeba vermiformis und wurde auch bei der Infektion dieser Spezies mit anderen Viren beobachtet, anders als wenn diese Amöben der Gattung Acanthamoeba infizieren,[3] beispielsweise bei „Faustovirus“[7] oder „Pacmanvirus“.[8] Es könnte also sein, dass sich „Orpheovirus“ in seinem natürlichen Wirt schneller vermehrt.
-
VF: Virusfabrik
-
N: Zellkern
Systematik
[Bearbeiten | Quelltext bearbeiten]Innere Systematik
[Bearbeiten | Quelltext bearbeiten]Die Familie Orpheoviridae gliedert sich mit Stand 30. April 2024 nach ICTV wie folgt:[9][10][11]
Familie Orpheoviridae
- Gattung Alphaorpheovirus
- Spezies Alphaorpheovirus massiliense
- Orpheovirus IHUMI-LCC2 alias Orpheovirus brasiliensis (OBRV)[2]
- Spezies Alphaorpheovirus massiliense

Die phylogenetische Analyse des Genoms von Orpheovirus IHUMI-LCC2 zeigt eine Verwandtschaft mit der Familie Pithoviridae. Einige spezifische Merkmale des Genoms zeugen jedoch von einer unterschiedlichen Entwicklung von Orpheovirus IHUMI-LCC2 im Vergleich zu den Pithoviridae wie auch zu den Cedratviridae. Andreani et al. als Autoren der Erstveröffentlichung 2018 schlugen daher vor, für Orpheovirus IHUMI-LCC2 eine eigene Familie Orpheoviridae einzurichten, eng verwandt mit den damals ebenfalls vorgeschlagenen Pithoviridae und Cedratviridae.[3][13]
Außerdem hatten Andreani et al. (2018) herausgefunden, dass ein zunächst den Rickettsiales-Bakterien zugeordneter aus Metagenomanalysen identifizierter Kandidat offenbar ein dem Orpheovirus IHUMI-LCC2 nahe verwandtes Riesenvirus ist („Misannotatedvirus“ alias „misidentified virus“) – eine Verwechslung, wie sie auch am Anfang der Entdeckungsgeschichte von Mimivirus geschah. Zudem fanden die Autoren noch Hinweise auf ein zweites Virus (mine drainage), dass basal in der Klade aus Pithoviridae, Cedatviridae und Orpeoviridae steht.<name="Andreani2018-04" />[12]
Als nächster Verwandter von Orpheovirus IHUMI-LCC2 gilt das noch ICTV-unbestätigte Sissivirus S55.[12]
Für die diese Funde einschließende Klade einer erweiterten Familie der Pithoviridae schlagen Schulz et al. folgende Systematik innerhalb der Pimascovirales vor[14] – ergänzt nach Julien Andreani et al. (2018) und Clara Rolland et al. (2019):
| „Pithovirus-like viruses“[15] („Pithoviridae s. l.“[16] ) |
| ||||||||||||||||||||||||||||||||||||
Anmerkungen:
- Für die Gattungsnamen wurden die seit dem 30. April 2024 durch das ICTV festgesetzten Bezeichnungen (mit dem Präfix Alpha-) eingesetzt.
- Das ICTV hat am 30. April 2024 für die Cedratviren eine eigene Familie Cedratviridae geschaffen (anders als in Andreani et al. 2018 vorgeschlagen)
- Das Kladogramm ist in Übereinstimmung mit der Systematik bei CNRS (2018)[13] und Rolland et al. (2019) Fig. 2a. – Fig. 2b zeigt dort ein etwas anderes Bild:[12]
Etymologie
[Bearbeiten | Quelltext bearbeiten]Der Name ist augenscheinlich abgeleitet von Orpheus, Sänger und Dichter in der griechische Mythologie mit Bezug zur Unterwelt, womöglich eine Anspielung auf die Herkunft des Virus. Der Präfix Alpha- wurde vom ICTV vergeben, um Mitglieder der Familie und der Gattung leicht unterscheiden zu können (Alphaorpheoviren bilden eine Teilmenge der Orpheoviren).[10]
Das Art-Epitheton von Alphaorpheovirus massiliense verweist auf den Sitz des Instituts IHU Méditerranée infection in Marseille, an dem das in La Ciotat geprobte Orpheovirus IHUMI-LCC2 identifiziert wurde.
Weblinks
[Bearbeiten | Quelltext bearbeiten]- Graziele Oliveira, Bernard La Scola, Jônatas Abrahão: Giant virus vs amoeba: fight for supremacy. In: Virol J, 16, 126, 4. November 2019; doi:10.1186/s12985-019-1244-3, researchgate.net (PDF)
- Khalil Geballa-Koukoulas, Bernard La Scola, Guillaume Blanc, Julien Andreani: Diversity of Giant Viruses Infecting Vermamoeba vermiformis. In: Frontiers in Microbiology, Band 13, 22. April 2022, S. 808499; doi:10.3389/fmicb.2022.808499 , PMID 35602053, PMC 9116030 (freier Volltext), PDF.
Einzelnachweise
[Bearbeiten | Quelltext bearbeiten]- ↑ a b c d e ICTV Master Species List 2019.v1. New MSL including all taxa updates since the 2018b release, March 2020 (MSL #35). ICTV.
- ↑ a b Gabriel Augusto Pires de Souza, Victória Fulgêncio Queiroz, Maurício Teixeira Lima, Erik Vinicius de Sousa Reis, Luiz Felipe Leomil Coelho, Jônatas Santos Abrahão: Virus goes viral: an educational kit for virology classes. In: Virology Journal, Band 17, Nr. 13, 31. Januar 2020, doi:10.1186/s12985-020-1291-9
- ↑ a b c d e f g h Julien Andreani, Jacques Y. B. Khalil, Emeline Baptiste, Issam Hasni, Caroline Michelle, Didier Raoult, Anthony Levasseur, Bernard La Scola: Orpheovirus IHUMI-LCC2: A New Virus among the Giant Viruses. In: Frontiers in Microbiology. Band 8, 22. Januar 2018, ISSN 1664-302X, doi:10.3389/fmicb.2017.02643 (englisch).
- ↑ NCBI Taxonomy Browser: Orpheovirus IHUMI-LCC2 (species).
- ↑ a b c Julien Andreani, Jonathan Verneau, Didier Raoult, Anthony Levasseurn Bernard La Scola: Deciphering viral presences: two novel partial giant viruses detected in marine metagenome and in a mine drainage metagenome. In: Virology Journal, Band 15, Nr. 66, 10. April 2018; doi:10.1186/s12985-018-0976-9 (englisch).
- ↑ a b Khalil Geballa-Koukoulas, Bernard La Scola, Guillaume Blanc, Julien Andreani: Diversity of Giant Viruses Infecting Vermamoeba vermiformis. In: Frontiers in Microbiology, Band 13, 22. April 2022, S. 808499; doi:10.3389/fmicb.2022.808499 , PMID 35602053, PMC 9116030 (freier Volltext), PDF (englisch).
- ↑ Dorine Gaëlle Reteno, Samia Benamar, Jacques Bou Khalil, Julien Andreani, Nicholas Armstrong, Thomas Klose, Michael Rossmann, Philippe Colson, Didier Raoult, Bernard La Scola: Faustovirus, an asfarvirus-related new lineage of giant viruses infecting amoebae. In: Journal of Virology, Band 89, Nr. 3, Juli 2015, S. 6585–94; doi:10.1128/JVI.00115-15, PMID 25878099, PMC 4468488 (freier Volltext) (englisch).
- ↑ Julien Andreani, Jacques Yaacoub Bou Khalil, Madhumati Sevvana, Samia Benamar, Fabrizio Di Pinto, Idir Bitam, Philippe Colson, Thomas Klose, Michael G. Rossmann, Didier Raoult, Bernard La Scola: Pacmanvirus, a New Giant Icosahedral Virus at the Crossroads between Asfarviridae and Faustoviruses. In: J Virol., v.91(14); 15. Juli 2017; PMID 28446673, PMC 5487549 (freier Volltext)
- ↑ ICTV: Taxonomy Browser.
- ↑ a b Victória Fulgêncio Queiroz, Rodrigo Araújo Lima Rodrigues, Jônatas Santos Abrahão: Create 3 new families, 3 genera, and 7 new species within the order Pimascovirales (phylum Nucleocytoviricota). Proposal 2023.011F, Juni 2023 (englisch).
- ↑ ICTV: Virus Metadata Resource (VMR).
- ↑ a b c d e f Clara Rolland, Julien Andreani, Amina Cherif Louazani, Sarah Aherfi, Rania Francis, Rodrigo Rodrigues, Ludmila Santos Silva, Dehia Sahmi, Said Mougari, Nisrine Chelkha, Meriem Bekliz, Lorena Silva, Felipe Assis, Fábio Dornas, Jacques Yaacoub Bou Khalil, Isabelle Pagnier, Christelle Desnues, Anthony Levasseur, Philippe Colson, Jônatas Abrahão, Bernard La Scola: Discovery and Further Studies on Giant Viruses at the IHU Mediterranee Infection That Modified the Perception of the Virosphere. In: Viruses, 11(4), März/April 2019, pii: E312, doi:10.3390/v11040312, PMC 6520786 (freier Volltext), PMID 30935049 (englisch).
- ↑ a b List of the main “giant” viruses known as of today. (PDF; 334 kB) Centre national de la recherche scientifique, Université Aix Marseille, 18. April 2018.
- ↑
Frederik Schulz, Lauren Alteio, Danielle Goudeau, Elizabeth M. Ryan, Feiqiao B. Yu, Rex R. Malmstrom, Jeffrey Blanchard, Tanja Woyke: Hidden diversity of soil giant viruses. In: Nature Communications, Band 9, 19. November 2018, Nr. 4881; doi:10.1038/s41467-018-07335-2 (englisch). Dazu:
- Jan Osterkamp: Virologie: Riesenviren sind weiter verbreitet als gedacht. Auf: Spektrum.de, 20. November 2018.
- ↑ Julien Guglielmini, Anthony C. Woo, Mart Krupovic, Patrick Forterre, Morgan Gaia: Diversification of giant and large eukaryotic dsDNnA viruses predated the origin of modern eukaryotes. In: PNAS, Band 116, Nr. 39, 10./24. September 2019, S. 19585–19592; doi:10.1073/pnas.1912006116, PMID 31506349 (englisch). Siehe insbes. Fig. 2.
- ↑ Eugene V. Koonin, Natalya Yutin: Multiple evolutionary origins of giant viruses. In: F1000 Research, 22. November 2018; doi:10.12688/f1000research.16248.1, version 1 (englisch).