Benutzer:Markus Prokott/Microhyla letovirus 1
Hier entsteht ein Artikel zur Virusart Microhyla letovirus 1.
Der Artikel enthält entweder die Beschreibung der Viren-Unterfamilie Letovirinae als ausführlichen Abschnitt anstatt eines eigenen Artikels über die Unterfamilie, oder aber eine Kurzdarstellung der Unterfamilie mit Verweis auf deren Hauptartikel °Letovirinae. Oder der Unterfamilien-Artikel ersetzt durch einen seiner Abschnitte diesen Artikel ganz.
| Microhyla letovirus 1 | ||||
|---|---|---|---|---|

Nidoviren-typische RNA-Verschachtelung | ||||
| Systematik | ||||
| Taxonomische Merkmale | ||||
| ||||
| Wissenschaftlicher Name | ||||
| Microhyla letovirus 1[1][2] | ||||
| Kurzbezeichnung | ||||
| MLeV[1][3] | ||||
| Links | ||||
|
Microhyla letovirus 1 (ICTV-Name; Abkürzung: MLeV; weitere Namen unten)[1][4] ist eine Virusart der Gattung °Alphaletovirus und deren Typusart.[2]
Es gehört zur Familie Coronaviridae und dort zur Unterfamilie der Letoviren (Letovirinae).[2] Als bisher einziges Virus dieser Unterfamilie repräsentiert es eine Schwestergruppe der Orthocoronaviren (d. h. der „eigentlichen Coronaviren“). Das Virus wurde bisher nur als virusartige RNA-Sequenz im Transkriptom des Engmaulfrosches Microhyla fissipes[5] (Gattung Microhyla) beschrieben und liegt dort als Coronavirus-artiges, partielles Nidovirus-Genom vor (vgl. a. Nidovirales genome (engl.)).[1]
Entdeckung
[Bearbeiten | Quelltext bearbeiten]

Ungefähre Entsprechung im Artikel:[6]
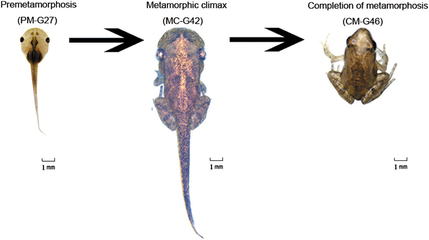
| (a) | ← | premetamorphosis (PM) |
| (c) | ← | metamorphic climax (MC) |
| (e) | ← | completion of metamorphosis (CM) |


2016 machten Zhao et al.[5] Untersuchungen an Fröschen der Art Microhyla fissipes (Gattung Microhyla). Dabei wurden die Transkriptome der Frösche in drei Schlüsselstadien der Frosch-Metamorphose:
- Prämetamorphose (englisch premetamorphosis, PM, Gosner-Stadium 27),
- Höhepunkt der Metamorphose (englisch metamorphic climax, MC, Gosner-Stadium 42) und
- Vollendung der Metamorphose (englisch completion of metamorphosis, CM, Gosner-Stadium 46)
(siehe auch Grafik 1) entschlüsselt, so dass man deren unterschiedliche Genaktivität, d. h. Genregulation und Genexpression (siehe auch Genexpressionsanalyse), erkennen und so die Bedeutung der dadurch jeweils produzierten Proteome, d. h. Protein-Gesamtheiten (hierbei vor allem der Schilddrüsenhormone), für die Froschentwicklung deuten konnte.[5]
Das Ergebnis waren eine Menge Frosch-Transkriptome, die in öffentlichen Datenbanken verfügbar gemacht wurden.[5] Diese Datenbanken wollten sich Bukhari et al. 2018[1] für die Erforschung der Nidoviren-Evolution zunutze machen und suchten systematisch nach noch unbekannten RNA-Virus-Genomen. Dabei konzentrierten sie sich auf die Suche nach Genomsequenzen, die den Viren der Ordnung Nidovirales (Nidoviren) ähnlich waren.[7]
Unter den von Zhao et al. 2016 gewonnenen RNA-Sequenzierungen von Fröschen entdeckten sie ein Coronavirus-artiges, 22,3 kb großes, partielles Genom, das potenziell einem Nidovirus entsprach. Dieses untersuchten sie im Folgenden eingehend zusammen mit einem weiteren 35,9 kb großen, potenziellen Nidovirus-Genom, das in kalifornischen Seehasen (Gattung Aplysia) gefunden worden war und später °Aplysia abyssovirus 1 genannt wurde. (Letzteres besaß das bis dahin zweitgröße bekannte Nidoviren-Genom, nach dem 41,1 kb großen Genom des °Planidovirus 1; PSCNV.[8][9])[7]
Die Ergebnisse zeigten zwei bis dato unbekannte Nidoviren, die in die dann vorhandene Familie Coronaviridae und die neugeschaffene Familie °Abyssoviridae eingeordnet wurden.[7]
Fehler in der Literatur
[Bearbeiten | Quelltext bearbeiten]Ein in der Revista da Sociedade Brasileira de Medicina Tropical (Vol. 53) im Februar 2020 unter dem Titel „Synanthropic rodents as virus reservoirs and transmitters“[10] veröffentlichter Artikel gibt die o. g. Arbeit von Bukhari et al. (2018) falsch wieder: Es wird beschrieben, dass die Untergattung Milecovirus (einschließlich Microhyla letovirus 1) bisher nur in Fröschen und einem Seehasen (engl. “frogs and a sea hare”) gefunden wurde. Offensichtlich ist hier aber das zusammen mit Microhyla letovirus 1 untersuchte Aplysia abyssovirus 1 gemeint, das tatsächlich im kalifornischen Seehasen gefunden wurde. Microhyla letovirus 1 wurde lediglich in der Froschart Microhyla fissipes gefunden.[10][1]
Merkmale
[Bearbeiten | Quelltext bearbeiten]Überblick
[Bearbeiten | Quelltext bearbeiten]Nidoviren
[Bearbeiten | Quelltext bearbeiten]Das Nidoviren-Genom enthält typischerweise eine ganze Reihe sog. open reading frames (ORFs). Diese ORFs können grob in drei Typen eingeteilt werden: ORF1a, ORF1b und die Gruppe der 3'ORFs.[11]:3
3'ORFs
[Bearbeiten | Quelltext bearbeiten]Die 3'ORFs liegen ineinander bzw. aufeinander am einen Ende des Genoms – das sog. 3'-Ende. Aus den 3'ORFs werden die sog. Strukturproteine der Nidoviren erzeugt.
Das 3'-Ende des Gesamtgenoms bildet auch das gemeinsame 3'-Ende aller dieser 3'ORF, daher der Name. Unterscheiden tun sie sich in ihrem jeweiligen anderen Ende (ihrem sog. 5'-Ende), sodass je zwei 3'ORFs einen gemeinsamen, überlappenden Anfangsteil haben.[11]:Abs. 5+6
Diese eigenständigen RNA-Rahmen werden nicht direkt in Proteine übersetzt, wie es grundsätzlich für positiv-strängige RNA möglich wäre. Sondern sie werden zunächst in einen Satz einzelner RNA-Stränge überschrieben, die dann in Proteine übersetzt werden. Aus jeder einzelnen sg mRNA wird jeweils ein einzelnes, individuelles Strukturprotein produziert.[12]:Abs. 2
Solche RNA-Einzelstränge nennt man „sub-genomic messenger RNAs“, oder kurz „sg mRNAs“. „Messenger RNAs“ bzw. deutsch „Boten-RNAs“, weil sie, wie bei Boten-RNAs in Zellen üblich, aus einem Genom erzeugt und dann zu Ribosomen transportiert werden, um daraus Proteine zu produzieren. „Sub-genomic“ bzw. deutsch „subgenomisch“, weil hier nicht das ganze Genom auf einmal in einen RNA-Strang kopiert wird, wie das bei RNA-Viren häufig ist, sonden nur ein Genomteil (Teilgenom) genutzt wird. Die ineinanderliegenden Zonen des Genoms, aus denen diese sg mRNAs erstellt werden, werden als „nested set“ (dt. „verschachtelter Satz“) sub-genomischer RNAs bezeichnet. Genfolgen (oder ein einzelnes Gen), die wie hier nicht nur Teil eines Genabschnittes sind, aus dem Kopien erzeugt werden, sondern, die von mehreren, sich überschneidenden Genabschnitten geteilt werden, nennt man „polycistronisch“, d.h. es werden aus ihnen positiv-Abbilder erstellt, die Teil von Nukleinsäureketten sind, die sich zwar in Terilen gleichen, insgesamt aber verschieden sind. Solche polycistronischen Abfolgen können dabei an beliebiger Stelle in einem Leseabschnitt viorkommen. Liegen mehrerer solcher polycistronischen Abfolgen innenander und teilen sich ein Ende, so heißen sie koterminal, in diesenm Falle 3'-koterminal, da es sich um das 3'-Ende handelt. Insgesamt handelt es sich also um 3'-koterminale subgenomische RNAs, die aus einem Satz ineinanderliegender Genabschnitte als Positiv-Abbilder erstellt werden, wobei viele der beteiligten Gene in diesen Abschnitten polycistronisch genutzt werden.[13][?!]
ORF1a
[Bearbeiten | Quelltext bearbeiten]Ein kleines Coronavirus ⸙ das wollte sich nicht fügen. ⸙ Da traf es auf ein Letovirus, ⸙ nun wohnen sie auf Rügen. ⸿ Genome sind zwar wunderschön, doch Gnome sind viel schöner. ⸿ Bla, bla, bla, es war einmal vor hundert Ja', ⸙ bag, bag, bag, an einem klirrend kalten Tag, ⸙ zah, zah, zah, war alles wunderbar, ⸙ buh, buh, buh, bis der Letovirus kam hinzu, ⸙ Brot, Brot, Brot, jetzt sind sie alle tot.
ORF1a / ORF1b
[Bearbeiten | Quelltext bearbeiten]Ein kleines Coronavirus ⸙ das wollte sich nicht fügen. ⸙ Da traf es auf ein Letovirus, ⸙ nun wohnen sie auf Rügen. ⸿ Genome sind zwar wunderschön, doch Gnome sind viel schöner. ⸿ Bla, bla, bla, es war einmal vor hundert Ja', ⸙ bag, bag, bag, an einem klirrend kalten Tag, ⸙ zah, zah, zah, war alles wunderbar, ⸙ buh, buh, buh, bis der Letovirus kam hinzu, ⸙ Brot, Brot, Brot, jetzt sind sie alle tot.
Coronaviren
[Bearbeiten | Quelltext bearbeiten]
Der letzte ICTV-Report aus dem Jahr 2011 gibt einen Überblick über die Eigenschaften der Viren[14] der Familie Coronaviridae. Damals gehörten noch die bazilliformen Viren der Gattungen Bafinivirus (Stäbchenform) und Torovirus (ringförmig gebogene Stäbchenform) dazu, während die Letoviren noch nicht bekannt waren. Unter Berücksichtigung dieser offensichtlichen und weiterer Änderungen ergibt sich für die Eigenschaften der Coronaviren, einschließlich der Letoviren, folgender Überblick:
- Genom
- Charakter: positive-sense single-stranded RNA (svw. „positiv-einzelsrängige RNS“)
- Gestalt: 1 lineares Segment
- Größe: 25–32 kb[15] (Stand: 2016)
- Virion
- Behüllt: Ja
- Gestalt: kugelig pleomorph bis rotationselliptisch[15] (Stand: 2016)
- Größe: 120–160 nm
- Wirt: Wirbeltiere[14]
Die Wirtsarten beschränken sich auf Wirbeltiere,[14] die rein terrestrisch leben oder ein obligates aquatisches Larvenstadium haben (Stand 2018).[16]:S. 168, Sp. 2 Zu Letzteren gehört auch die Froschart Microhyla fissipes im hier behandelten Falle.
Letoviren
[Bearbeiten | Quelltext bearbeiten]Von den Letoviren als Gruppe ist noch nicht viel bekannt, da sie bisher nur durch eine Art (die hier Beschriebene) vertreten werden.


Microhyla letovirus 1
[Bearbeiten | Quelltext bearbeiten]Die hier beschriebene einzige bekannte Art der Letoviren besitzt die gleichen grundlegenden Charakteristika wie klassische Coronaviren, d. h. wie die Viren der heutigen Unterfamilie Orthocoronavirinae.
- Genom
- Charakter: positive-sense single-stranded RNA
- Gestalt: 1 lineares Segment
- Größe: 23,8 kb–26,3 kb (= 22,3 kb + [1,5–4,0] kb)
- Virion
- Behüllt: Ja
- Gestalt: (vermutlich) pleomorph kugelförmig
- Größe: n. A.
- Wirt: Microhyla fissipes
Bei ihrer Entdeckung unterschied sich Microhyla letovirus 1 nur in ihrer Genetik von den bis dahin bekannten sog. „echten Coronaviren“ („true coronaviruses“[17], d. h. die Viren der damaligen Unterfamilie Coronavirinae), und nicht wesentlich in anderen Bereichen wie Gestalt oder Wirtsspektrum. Außerdem bildete sie in computergestützten genetischen Vergleichsberechnungen eine statistisch stark unterstützte monophyletische Gruppe (Klade) unmittelbar benachbart zu den damaligen echten Coronaviren, aber sehr deutlich entfernt zu allen anderen, damals auch noch als Coronaviren bezeichneten Viren.
Die anderen damaligen Coronaviren, damals „Toroviren“, heute „Tobaniviren“ genannt, wichen in ihrer Form von den klassischen Coronaviren ab. Toroviren waren mehr oder weniger bazilliform, Coronaviren und die neue Art waren mehr oder weniger kugelförmig. Auch bestanden größere Unterschiede im Virionen- und im Genomaufbau (Hüllenproteine bzw. Proteindomänen) der beiden Gruppen.
Es fehlte jedoch ein Teil des Genoms von Microhyla letovirus 1,[18][19] sodass im fehlenden Abschnitt keine Aussage über die paarweise Aminosäurenübereinstimmung zwischen den Gensequenzen der neuen Art und denen der anderen Coronaviren gemacht werden konnte. Dieser fehlende Abschnitt (bestehend aus den sog. non-structural proteins, nsps:[20] nsp1, nsp2 und größtenteils nsp3) beinhaltete aber nur den Teil des Genoms von Nidoviren, der am veränderlichsten in Bezug auf Art, Aufbau und Verteilung der Proteindomänen, selbst innerhalb einer Gattung, ist, und so kaum zu einer Abgrenzung von Viren beiträgt.[16] Andererseits deutete alles darauf hin, dass der vorhandene Teil des Genoms einen vollständigen Satz aller konservierten Coronavirus-artigen Proteine kodiert.[21] Diese beiden Umstände – Unerheblichkeit des fehlenden Bereichs, vollständige Produktion konservierter Proteine im vorhandenen Bereich –, zusammen mit der Tatsache, dass innerhalb der damaligen echten Coronaviren die Sequenzgleichheit gering, die Domänenkonservierung aber hoch war, führte die Forscher dazu, die neue Art den echten Coronaviren zuzuordnen.[22]
Genomstruktur
[Bearbeiten | Quelltext bearbeiten]Partielles, Coronavirus-artiges Genom beginnend bei SARS-CoV-nsp3 bis zum Ende (2.8, Anfang, S. 165, unten). Anfangsteile typisch zu unübersetzter Region und ca. Coronavirus-nsp1–3 mit geschätzter Größe (2.8, Anfang, S. 166, mitte-links und oben-rechts). Vollständiger Satz konservierter Coronavirus-artiger Proteine von nsp3-Abschnittsende bis Gesamtgenom-Ende (2.9, Anfang, S. 168, mitte-links). Region bis zum SARS-CoV-nsp4-Abschnitt hochvariable (S. 168, unten-links).
Ein kleines Coronavirus ⸙ das wollte sich nicht fügen. ⸙ Da traf es auf ein Letovirus, ⸙ nun wohnen sie auf Rügen. ⸿ Genome sind zwar wunderschön, doch Gnome sind viel schöner. ⸿ Bla, bla, bla, es war einmal vor hundert Ja', ⸙ bag, bag, bag, an einem klirrend kalten Tag, ⸙ zah, zah, zah, war alles wunderbar, ⸙ buh, buh, buh, bis der Letovirus kam hinzu, ⸙ Brot, Brot, Brot, jetzt sind sie alle tot.
Benennung
[Bearbeiten | Quelltext bearbeiten]Etymologie
[Bearbeiten | Quelltext bearbeiten]

Der Name wurde in der Erstbeschreibung von Bukhari et al. (2018)[1] aufgrund der Entdeckung in Microhyla-fissipes-Fröschen gewählt:[22]
Nach Ovids Metamorphosen verwandelte Leto, die Tochter der Titanen Koios und Phoibe, auf der Flucht vor der Göttin Hera einige Hirten in Lykien in Frösche. Sie bestrafte sie so, weil sie den Schlamm am Grunde eines Teiches aufgewühlt hatten, um es Leto nicht zu gestatten, aus ihm zu trinken.[22]
Der Namensteil Microhyla geht offensichtlich auf die Wirtsspezies Microhyla fissipes und deren Gattung Microhyla zurück und setzt sich aus dem Vorsatz „Micro-“ (griechisch μικρός mikrós „klein“) und dem Namen der verwandten Frosch-Gattung Hyla zusammen. Letzterer geht wiederum auf Hylas, einen Geliebten des Helden Herakles, zurück. (Kommentar im Quellcode!)
Namen
[Bearbeiten | Quelltext bearbeiten]Für diese Virusart gibt es den offiziell vom ICTV zugelassenen, eindeutigen Namen. Daneben werden in der Literatur unterschiedliche weitere Namen und Abkürzungen gebraucht. Die folgenden Namen sind im Gebrauch:
- Microhyla letovirus 1 (offizieller ICTV-Name)[1][2]
- MLeV[1]
- Microhyla fissipes corona-like virus[24]
- Microhyla alphaletovirus 1[24]
Systematik
[Bearbeiten | Quelltext bearbeiten]Microhyla letovirus 1 ist aktuell die einzige Art in der Untergattung °Milecovirus, dies die einzige Untergattung in der Gattung °Alphaletovirus, dies die einzige Gattung in der Unterfamilie Letovirinae, dies neben Orthocoronavirinae eine von zwei Unterfamilien in der Familie Coronaviridae in der Ordnung Nidovirales.
- Bereich Riboviria (RNA-Viren)
- Reich Orthornavirae
- Stamm Pisuviricota
- Klasse Pisoniviricetes
- Ordnung Nidovirales
- Unterordnung Cornidovirineae
- Familie Coronaviridae
- Unteramilie Orthocoronavirinae (Sammelbezeichnungen für Viren hierin: sowohl Orthocoronaviren, als auch, wie zuvor, „echte“, „eigentliche“ etc. Coronaviren, als auch immer noch schlicht (die) Coronaviren), ehem. Unteramilie Coronavirinae,[25] ehem. Gattung Coronavirus, viele verschiedene Viren[17]
- …
Unteramilie Torovirinae→ Familie Tobaniviridae- Unteramilie Letovirinae
- Gattung Alphaletovirus
- Untergattung Milecovirus
- vorgeschl. Art Microhyla letovirus 1 (Typusart), bei Microhyla fissipes (Art der kleinen Baumfrösche) (*)
- Untergattung Milecovirus
- Gattung Alphaletovirus
- Unteramilie Orthocoronavirinae (Sammelbezeichnungen für Viren hierin: sowohl Orthocoronaviren, als auch, wie zuvor, „echte“, „eigentliche“ etc. Coronaviren, als auch immer noch schlicht (die) Coronaviren), ehem. Unteramilie Coronavirinae,[25] ehem. Gattung Coronavirus, viele verschiedene Viren[17]
- Familie Coronaviridae
- Unterordnung Cornidovirineae
- Ordnung Nidovirales
- Klasse Pisoniviricetes
- Stamm Pisuviricota
- Reich Orthornavirae
Microhyla letovirus 1
[Bearbeiten | Quelltext bearbeiten]In Verbindung mit der geringen Sequenzgleichheit und der gleichzeitig hohen Proteindomänen-Konservierung innerhalb der klassischen Coronaviren (Orthocoronavirinae) und mithilfe von Computeranalysen, deutete man die mögliche Art „Microhyla letovirus 1“ als Mitglied einer möglichen Schwestergruppe aller Coronaviren. Damals umfassten sowohl die Familie Coronaviridae als auch deren einzige Unterfamilie Coronavirinae alle bekannten Coronaviren. Die Forschungsgruppe Bukhari et al. (2018)[1] sah diese neue Schwestergruppe als neue Gattung innerhalb der damaligen Unterfamilie Coronavirinae (heute Orthocoronavirinae). Es wurde daher zunächst Ende 2018 eine neue Gattung „Alphaletovirus“ vorgeschlagen, neben den Gattungen Alpha- bis Deltacoronavirus (vgl. Gattungen in Orthocoronavirinae) der damaligen Unterfamilie Coronavirinae.[22]
Vom ICTV wurde stattdessen 2018 eine neue Unterfamilie Letovirinae, der Letoviren, innerhalb der Familie Coronaviridae geschaffen und als zweite neben die bis dahin alleinstehende Unterfamilie Coronavirinae gestellt. Letztere wurde parallel dazu in Orthocoronavirinae (svw. „eigentliche Coronaviren“) unbenannt. Microhyla letovirus 1 wurde als Typusart der Gattung Alphaletovirus gemeinsam mit dieser ratifiziert und innerhalb Letovirinae eingeordnet. Die Untergattung Milecovirus schließlich machte die o. g. Struktur der Unterfamilie Letovirinae vollständig.[3] Microhyla letovirus 1 ist bisher die einzige Art innerhalb Letovirinae geblieben.
Ein möglicher naher Verwandter von Microhyla letovirus 1 ist das bisher unklassifizierte Nidovirus „°Pacific salmon nidovirus“[26] (PsNV). Es ist wahrscheinlich, dass es innerhalb der Familie Coronaviridae zu einer eigenen Gattung parallel zu Alphaletovirus gehören wird. Ob innerhalb derselben Unterfamilie Letovirinae, oder jenseits davon ist noch unklar.[24]
Milecovirus
[Bearbeiten | Quelltext bearbeiten]Milecovirus ist eine Virusuntergattung. Sie ist die einzige Untergattung in der Virengattung Alphaletovirus. Sie enthält auch die Typusart dieser Gattung, die hier beschriebene Virusart Microhyla letovirus 1. Der Name ist ein Kofferwort (nach ICTV-Code ein sog. siglum (engl. / lat.)) bezugnehmend auf Microhyla letovirus-like coronavirus und ähnliche Formulierungen.
Damit entspricht der Name einem Schema vieler anderen Untergattungen innerhalb der beiden Virenfamilien Coronaviridae und Tobaniviridae innerhalb der Ordnung der Nidoviren, die explizit so sprechend benannt wurden, um der starken vergangenen und zukünftigen Zunahme an Neuentdeckungen und -erforschungen von Viren (aufgrund technischen Fortschrittes) einen stabilen Rahmen für die systematische Einordnung zu bieten. Bereits bei Erarbeitung dieser taxonomischen Struktur bestand ein beträchtlicher Rückstau an nicht ausreichend bestimmten Viren innerhalb dieser beiden Familien. Sorgen des ICTV, ob man diese Viren gemäß ihrer molekularen und biologischen Eigenschaften möglichst einfach und rationell in die vorhandene Struktur einordnen können würde, ohne diese ändern zu müssen, und somit die zukünftige Stabilität dieses taxonomischen Rahmens gewährleistet sei, wollte man durch diesen und andere hilfreiche Aspekte der taxonomischen Struktur abhelfen.[27]
Alphaletovirus
[Bearbeiten | Quelltext bearbeiten]Alphaletovirus ist eine Virusgattung. Sie ist die einzige Gattung in der Virenunterfamilie Letovirinae. Wie für Gattungen üblich, wurde sie im Rahmen der Entdeckung und Festlegung ihrer Typusart gebildet. Typusart ist die hier beschriebene Art Microhyla letovirus 1.
Letovirinae
[Bearbeiten | Quelltext bearbeiten]Letovirinae ist eine der beiden Unterfamilien in der Virusfamilie Coronaviridae.
Sie wurde im Zuge der Entdeckung der hier beschriebenen Virusart Microhyla letovirus 1 geschaffen und nach dieser benannt. Sie enthält bisher nur diese Virusart und grenzt diese besondere Coronavirus-Art von den „eigentlichen Coronaviren“ der Unterfamilie Orthocoronavirinae ab.[1]
Höhere Taxa
[Bearbeiten | Quelltext bearbeiten]Familie(, Unterordnung?) und Ordnung.
Prävalenz
[Bearbeiten | Quelltext bearbeiten]

Das Virus zeigte eine sehr starke Altersabhängigkeit bei seiner mengenmäßigen Auffindbarkeit in Microhyla fissipes.[18][6]
In den in der ursprünglichen Frosch-Studie (Zhao et al., 2016[5]) untersuchten Fröschen zeigte sich zwischen den drei untersuchten Metamorphose-Stadien eine starke Verminderung der feststellbaren Detektion von Microhyla letovirus 1: Eine 7-fache Verminderung von Stadium PM nach Stadium MC und eine nochmals 14-fache Verminderung von Stadium MC nach Stadium CM. (Für Abkürzungen siehe Abschnitt „Entdeckung“.) Erkundigungen der zweiten Forschungsgruppe (Bukhari et al., 2018[1]) ergaben, dass weitere, später untersuchte, vollständig ausgewachsene Microhyla fissipes mithilfe von MLeV-spezifischer PCR getestet und alle Frösche MLeV-negativ befunden worden waren. Microhyla letovirus 1 also beim adulten Tier gänzlich verschwunden war.[18][6]
Siehe auch
[Bearbeiten | Quelltext bearbeiten]Weblinks
[Bearbeiten | Quelltext bearbeiten]- Geraldine (Jay) Mulley, eine der Erstbeschreiberinnen, an der University of Reading, UK (irrelevant?)
- Microhyla fissipes auf AmphibiaWeb
Quellen
[Bearbeiten | Quelltext bearbeiten]Literatur
[Bearbeiten | Quelltext bearbeiten]- Khulud Bukhari, Geraldine Mulley, Anastasia A. Gulyaeva, Lanying Zhao, Guocheng Shu, Jianping Jiang, Benjamin W. Neuman: Description and initial characterization of metatranscriptomic nidovirus-like genomes from the proposed new family Abyssoviridae, and from a sister group to the Coronavirinae, the proposed genus Alphaletovirus. In: Virology. Band 524, Ausgabe November 2018, S. 160–171. Elsevier, 7. September 2018, doi:10.1016/j.virol.2018.08.010, PMID 30199753, PMC 7112036 (freier Volltext) – (englisch, Volltext [PDF; 3,3 MB; abgerufen am 18. Mai 2020] „Coronavirinae“: heute „Orthocoronavirinae“).
Einzelnachweise
[Bearbeiten | Quelltext bearbeiten]- Letovirus, Hauptwerk
[1] [28] [29] [30] [31] [32] [33] [34] [19] [6] [7] [35] [36] [37] [22] [38] [39] [40] [41] [18] [21] [16]
- Letovirus, Zusätzliches
- Coronaviren
- Fledermaus-Coronaviren
- Nidoviren
- PsNV
- Planidovirus
[9] [46] [47] [11] [48] [49] [50] [51] [52] [53] [54] [55] [56] [57] [58] [59] [60] [61] [62] [63] [64] [65] [66] [67] [68] [69] [70] [71] [72] [73] [74] [75] [76] [77] [78] [79] [80] [81] [82] [83] [84] [85] [86] [87] [88] [89] [90] [91] [92] [93] [94] [95] [96] [97] [98]
- ICTV-Referenzen
[99] [2] [100] [101] [3] [8] [102] [103] [104] [27] [105] [25] [17]
- ICTV-FAQs
- ICTV 9th Report
[107] [108] [109] [110] [111] [112] [113] [114] [14] [115]
- Weitere
- ↑ a b c d e f g h i j k l m Khulud Bukhari, Geraldine Mulley, Anastasia A. Gulyaeva, Lanying Zhao, Guocheng Shu, Jianping Jiang, Benjamin W. Neuman: Description and initial characterization of metatranscriptomic nidovirus-like genomes from the proposed new family Abyssoviridae, and from a sister group to the Coronavirinae, the proposed genus Alphaletovirus. In: Virology. Band 524, Ausgabe November 2018, S. 160–171. Elsevier, 7. September 2018, doi:10.1016/j.virol.2018.08.010, PMID 30199753, PMC 7112036 (freier Volltext) – (englisch, Volltext [PDF; 3,3 MB; abgerufen am 18. Mai 2020] „Coronavirinae“: heute „Orthocoronavirinae“).
- ↑ a b c d e f g h i j k l m n o p ICTV: ICTV Master Species List 2019.v1. MSL #35, März 2020
- ↑ a b c Datenbankeintrag und relevantes Proposal:
- Eintrag: ICTV Taxonomy history: Microhyla letovirus 1. In: ICTV-Homepage. International Committee on Taxonomy of Viruses (ICTV), abgerufen am 7. Mai 2020 (englisch).
- Proposal 2017.013S zu ICTV-Revision № 2018a: Reorganization of the family Coronaviridae into two families, Coronaviridae (including the current subfamily Coronavirinae and the new subfamily Letovirinae) and the new family Tobaniviridae (accommodating the current subfamily Torovirinae and three other subfamilies), revision of the genus rank structure and introduction of a new subgenus rank. (Eine von mehreren PDF-Dateien in ZIP-Ordner; 4,49 MB) 2017.013S. In: ICTV-Homepage. International Committee on Taxonomy of Viruses (ICTV), abgerufen am 7. Mai 2020 (englisch).
- ↑ a b How to write virus and species names? In: ICTV-Homepage. International Committee on Taxonomy of Viruses (ICTV), 6. April 2020, abgerufen am 7. Mai 2020 (englisch).
- ↑ a b c d e f Lanying Zhao, Lusha Liu, Shouhong Wang, Hongyuan Wang, Jianping Jiang: Transcriptome profiles of metamorphosis in the ornamented pygmy frog Microhyla fissipes clarify the functions of thyroid hormone receptors in metamorphosis. In: Scientific Reports. Band 6, Artikelnr. 27310. Springer-Verlag, 2. Juni 2016, ISSN 2045-2322, doi:10.1038/srep27310, PMID 27254593, PMC 4890586 (freier Volltext) – (englisch, Volltext [PDF; 3,3 MB; abgerufen am 18. Mai 2020]).
- ↑ a b c d Khulud Bukhari, Geraldine Mulley, Anastasia A. Gulyaeva, Lanying Zhao, Guocheng Shu, Jianping Jiang, Benjamin W. Neuman: Description and initial characterization of metatranscriptomic nidovirus-like genomes from the proposed new family Abyssoviridae, and from a sister group to the Coronavirinae, the proposed genus Alphaletovirus. In: Virology. Band 524, Ausgabe November 2018, S. 160–171. Elsevier, 7. September 2018, Fig. 8. Coding Capacity and prevalence of MLeV, Teilbild B, S. 167, doi:10.1016/j.virol.2018.08.010, PMID 30199753, PMC 7112036 (freier Volltext) – (englisch, Volltext [PDF; 3,3 MB; abgerufen am 18. Mai 2020] „Coronavirinae“: heute „Orthocoronavirinae“).
- ↑ a b c d Khulud Bukhari, Geraldine Mulley, Anastasia A. Gulyaeva, Lanying Zhao, Guocheng Shu, Jianping Jiang, Benjamin W. Neuman: Description and initial characterization of metatranscriptomic nidovirus-like genomes from the proposed new family Abyssoviridae, and from a sister group to the Coronavirinae, the proposed genus Alphaletovirus. In: Virology. Band 524, Ausgabe November 2018, S. 160–171. Elsevier, 7. September 2018, Abstract, S. 160, doi:10.1016/j.virol.2018.08.010, PMID 30199753, PMC 7112036 (freier Volltext) – (englisch, Volltext [PDF; 3,3 MB; abgerufen am 18. Mai 2020] „Coronavirinae“: heute „Orthocoronavirinae“).
- ↑ a b Datenbankeintrag und relevantes Proposal:
- Eintrag: ICTV Taxonomy history: Planidovirus 1. In: ICTV-Homepage. International Committee on Taxonomy of Viruses (ICTV), abgerufen am 22. Mai 2020 (englisch).
- Proposal 2017.013S zu ICTV-Revision № 2018a: Reorganization of the family Coronaviridae into two families, Coronaviridae (including the current subfamily Coronavirinae and the new subfamily Letovirinae) and the new family Tobaniviridae (accommodating the current subfamily Torovirinae and three other subfamilies), revision of the genus rank structure and introduction of a new subgenus rank. (Eine DOCX-Datei in ZIP-Ordner (mit mehreren Dateien); 4,49 MiB) 2017.013S. In: ICTV-Homepage. International Committee on Taxonomy of Viruses (ICTV), abgerufen am 7. Mai 2020 (englisch).
- ↑ a b Amir Saberi, Anastasia A. Gulyaeva, John L. Brubacher, Phillip A. Newmark, Alexander E. Gorbalenya: A planarian nidovirus expands the limits of RNA genome size. In: PLOS Pathogens. Band 14, Ausgabe 11, Artikel e1007314, 1. November 2018, doi:10.1371/journal.ppat.1007314, PMID 30383829, PMC 6211748 (freier Volltext) – (englisch, Volltext [PDF; 4,2 MB; abgerufen am 1. Juni 2020]).
- ↑ a b c Mara Lucia Gravinatti, Carla Meneguin Barbosa, Rodrigo Martins Soares, Fábio Gregori: Synanthropic rodents as virus reservoirs and transmitters. Version 1. In: Revista da Sociedade Brasileira de Medicina Tropical. Band 53. Sociedade Brasileira de Medicina Tropical (SBMT), 7. Februar 2020, 20190486, doi:10.1590/0037-8682-0486-2019 (englisch, Volltext [PDF; 997 kB; abgerufen am 21. Mai 2020] Falschzitation einer Veröffentlichung von Bukhari et al.).
- ↑ a b c Amir Saberi, Anastasia A. Gulyaeva, John L. Brubacher, Phillip A. Newmark, Alexander E. Gorbalenya: A planarian nidovirus expands the limits of RNA genome size. In: PLOS Pathogens. Band 14, Ausgabe 11, Artikel e1007314, 1. November 2018, Kapitel „Introduction“, S. 2–5, doi:10.1371/journal.ppat.1007314, PMID 30383829, PMC 6211748 (freier Volltext) – (englisch, Volltext [PDF; 4,2 MB; abgerufen am 1. Juni 2020]).
- ↑ a b Fehr A.R., Perlman S.: Coronaviruses: An Overview of Their Replication and Pathogenesis (= Maier H., Bickerton E., Britton P. [Hrsg.]: Coronaviruses. Methods in Molecular Biology. Band 1282, S. 1–23). Humana Press, New York 2015, ISBN 978-1-4939-2438-7, doi:10.1007/978-1-4939-2438-7_1 (englisch).
- ↑ a b c d Autoren des neunten ICTV-Reports: Virus Taxonomy – Classification and Nomenclature of Viruses. Online-Ausgabe. Einleitungskapitel: „Virus Properties“, vor allem Tabellenzeile Nidovirales – Coronaviridae. In: ICTV Reports. International Committee on Taxonomy of Viruses, 2011, abgerufen am 12. Juni 2020 (englisch, parallel archiviert am 2. April 2019 auf web.archive.org.).
- ↑ a b c B.W. Neuman, M.J. Buchmeier: Coronaviruses. Nur Kapitel 1: Supramolecular Architecture of the Coronavirus Particle. In: Advances in Virus Research. 1. Auflage. Band 96. Elsevier Inc., 2016, ISBN 978-0-12-804736-1, ISSN 0065-3527, Abschnitt 2: Virion Structure and Durability, S. 4+5, doi:10.1016/bs.aivir.2016.08.005 (englisch, Volltext [PDF; 1,3 MB; abgerufen am 15. Juni 2020]).
- ↑ a b c Khulud Bukhari, Geraldine Mulley, Anastasia A. Gulyaeva, Lanying Zhao, Guocheng Shu, Jianping Jiang, Benjamin W. Neuman: Description and initial characterization of metatranscriptomic nidovirus-like genomes from the proposed new family Abyssoviridae, and from a sister group to the Coronavirinae, the proposed genus Alphaletovirus. In: Virology. Band 524, Ausgabe November 2018, S. 160–171. Elsevier, 7. September 2018, 3. Discussion and conclusions, S. 168 f., doi:10.1016/j.virol.2018.08.010, PMID 30199753, PMC 7112036 (freier Volltext) – (englisch, Volltext [PDF; 3,3 MB; abgerufen am 18. Mai 2020] „Coronavirinae“: heute „Orthocoronavirinae“).
- ↑ a b c Raoul J. de Groot, John Ziebuhr, Leo L. Poon, Patrick C.Woo, Pierre Talbot, Peter J.M. Rottier, Kathryn V. Holmes, Ralph Baric, Stanley Perlman, Luis Enjuanes, Alexander E. Gorbalenya: Revision of the family Coronaviridae. Proposal. In: Virus Taxonomy. History. Revision 2009, June. International Committee on Taxonomy of Viruses (ICTV), 2008, Proposal-Code 2008.085-126V (englisch, ictvonline.org [PDF; 175 kB; abgerufen am 5. Mai 2020]).
- ↑ a b c d Khulud Bukhari, Geraldine Mulley, Anastasia A. Gulyaeva, Lanying Zhao, Guocheng Shu, Jianping Jiang, Benjamin W. Neuman: Description and initial characterization of metatranscriptomic nidovirus-like genomes from the proposed new family Abyssoviridae, and from a sister group to the Coronavirinae, the proposed genus Alphaletovirus. In: Virology. Band 524, Ausgabe November 2018, S. 160–171. Elsevier, 7. September 2018, 2.8. MLeV genome, S. 165 ff., doi:10.1016/j.virol.2018.08.010, PMID 30199753, PMC 7112036 (freier Volltext) – (englisch, Volltext [PDF; 3,3 MB; abgerufen am 18. Mai 2020] „Coronavirinae“: heute „Orthocoronavirinae“).
- ↑ a b Khulud Bukhari, Geraldine Mulley, Anastasia A. Gulyaeva, Lanying Zhao, Guocheng Shu, Jianping Jiang, Benjamin W. Neuman: Description and initial characterization of metatranscriptomic nidovirus-like genomes from the proposed new family Abyssoviridae, and from a sister group to the Coronavirinae, the proposed genus Alphaletovirus. In: Virology. Band 524, Ausgabe November 2018, S. 160–171. Elsevier, 7. September 2018, Fig. 8. Coding Capacity and prevalence of MLeV, Teilbild A, S. 167, doi:10.1016/j.virol.2018.08.010, PMID 30199753, PMC 7112036 (freier Volltext) – (englisch, Volltext [PDF; 3,3 MB; abgerufen am 18. Mai 2020] „Coronavirinae“: heute „Orthocoronavirinae“).
- ↑ a b Natacha S. Ogando, Francois Ferron, Etienne Decroly, Bruno Canard, Clara C. Posthuma and Eric J. Snijder: The Curious Case of the Nidovirus Exoribonuclease: Its Role in RNA Synthesis and Replication Fidelity. In: Aartjan Te Velthuis (Hrsg.): Frontiers in Microbiology. Band 10, 7. August 2010, 1813, doi:10.3389/fmicb.2019.01813, PMID 31440227, PMC 6693484 (freier Volltext) – (englisch, frontiersin.org [PDF; 6,8 MB; abgerufen am 1. Juni 2020]).
- ↑ a b Khulud Bukhari, Geraldine Mulley, Anastasia A. Gulyaeva, Lanying Zhao, Guocheng Shu, Jianping Jiang, Benjamin W. Neuman: Description and initial characterization of metatranscriptomic nidovirus-like genomes from the proposed new family Abyssoviridae, and from a sister group to the Coronavirinae, the proposed genus Alphaletovirus. In: Virology. Band 524, Ausgabe November 2018, S. 160–171. Elsevier, 7. September 2018, 2.9. MLeV protein bioinformatics, S. 168, doi:10.1016/j.virol.2018.08.010, PMID 30199753, PMC 7112036 (freier Volltext) – (englisch, Volltext [PDF; 3,3 MB; abgerufen am 18. Mai 2020] „Coronavirinae“: heute „Orthocoronavirinae“).
- ↑ a b c d e Khulud Bukhari, Geraldine Mulley, Anastasia A. Gulyaeva, Lanying Zhao, Guocheng Shu, Jianping Jiang, Benjamin W. Neuman: Description and initial characterization of metatranscriptomic nidovirus-like genomes from the proposed new family Abyssoviridae, and from a sister group to the Coronavirinae, the proposed genus Alphaletovirus. In: Virology. Band 524, Ausgabe November 2018, S. 160–171. Elsevier, 7. September 2018, 2.3. Naming and etymology, S. 161, doi:10.1016/j.virol.2018.08.010, PMID 30199753, PMC 7112036 (freier Volltext) – (englisch, Volltext [PDF; 3,3 MB; abgerufen am 18. Mai 2020] „Coronavirinae“: heute „Orthocoronavirinae“).
- ↑ a b Erstellung und Bearbeitung der Bildunterschrift auf de.wikipedia.org:
- 2013-04-13 07:24 (UTC) . . Ute Erb (http://www.duden.de/suchen/dudenonline/als%20n%C3%A4chstes)
- 2012-03-06 21:45 (UTC) . . 84.176.183.30 (k (Bildunterschrift))
- 2007-11-03 01:53 (UTC) . . PodracerHH (+ Pic)
- ↑ a b c d Gideon J Mordecai, Kristina M Miller, Emiliano Di Cicco, Angela D Schulze, Karia H Kaukinen, Tobi J Ming, Shaorong Li, Amy Tabata, Amy Teffer, David A Patterson, Hugh W Ferguson, Curtis A Suttle: Endangered wild salmon infected by newly discovered viruses. Version 1. In: eLife. eLife Sciences Publications Ltd., 3. September 2019, 47615, doi:10.7554/eLife.47615 (englisch, Volltext [PDF; 2,7 MB; abgerufen am 21. Mai 2020]): “The novel nidovirus, named Pacific salmon nidovirus (PsNV), is most closely related to the recently described Microhyla alphaletovirus 1 […].”
- ↑ a b J. Ziebuhr, R.S. Baric, S. Baker, R.J. de Groot, C. Drosten, A. Gulyaeva, B.L. Haagmans, B.W. Neuman, S. Perlman, L.L.M. Poon, I. Sola, A.E. Gorbalenya: Reorganization of the family Coronaviridae into two families, Coronaviridae (including the current subfamily Coronavirinae and the new subfamily Letovirinae) and the new family Tobaniviridae (accommodating the current subfamily Torovirinae and three other subfamilies), revision of the genus rank structure and introduction of a new subgenus rank. Proposal. In: Virus Taxonomy. History. Revision 2018a, Juli. International Committee on Taxonomy of Viruses (ICTV), 18. Februar 2017, Proposal-Code 2017.013S (englisch, ictvonline.org [ZIP; 5,1 MB; abgerufen am 7. Mai 2020]).
- ↑ a b NCBI Taxonomy browser: Pacific salmon nidovirus. In: NCBI-Homepage. National Center for Biotechnology Information (NCBI), abgerufen am 21. Mai 2020 (englisch).
- ↑ a b ICTV-Proposal 2018a: 2017.012_015S.A.v1.Nidovirales. (Verschiedene Dokument-Dateien in ZIP-Ordner; 4,49 MiB) In: ICTV-Homepage. International Committee on Taxonomy of Viruses (ICTV), abgerufen am 7. Mai 2020 (englisch).
- ↑ Khulud Bukhari, Geraldine Mulley, Anastasia A. Gulyaeva, Lanying Zhao, Guocheng Shu, Jianping Jiang, Benjamin W. Neuman: Description and initial characterization of metatranscriptomic nidovirus-like genomes from the proposed new family Abyssoviridae, and from a sister group to the Coronavirinae, the proposed genus Alphaletovirus. In: Virology. Band 524, Ausgabe November 2018, S. 160–171. Elsevier, 7. September 2018, Fig. 3. Coding capacity, depth of coverage and bioinformatics of AAbV, S. 163, doi:10.1016/j.virol.2018.08.010, PMID 30199753, PMC 7112036 (freier Volltext) – (englisch, Volltext [PDF; 3,3 MB; abgerufen am 18. Mai 2020] „Coronavirinae“: heute „Orthocoronavirinae“).
- ↑ Khulud Bukhari, Geraldine Mulley, Anastasia A. Gulyaeva, Lanying Zhao, Guocheng Shu, Jianping Jiang, Benjamin W. Neuman: Description and initial characterization of metatranscriptomic nidovirus-like genomes from the proposed new family Abyssoviridae, and from a sister group to the Coronavirinae, the proposed genus Alphaletovirus. In: Virology. Band 524, Ausgabe November 2018, S. 160–171. Elsevier, 7. September 2018, Fig. 3. Coding capacity, depth of coverage and bioinformatics of AAbV, Teilbild A, S. 163, doi:10.1016/j.virol.2018.08.010, PMID 30199753, PMC 7112036 (freier Volltext) – (englisch, Volltext [PDF; 3,3 MB; abgerufen am 18. Mai 2020] „Coronavirinae“: heute „Orthocoronavirinae“).
- ↑ Khulud Bukhari, Geraldine Mulley, Anastasia A. Gulyaeva, Lanying Zhao, Guocheng Shu, Jianping Jiang, Benjamin W. Neuman: Description and initial characterization of metatranscriptomic nidovirus-like genomes from the proposed new family Abyssoviridae, and from a sister group to the Coronavirinae, the proposed genus Alphaletovirus. In: Virology. Band 524, Ausgabe November 2018, S. 160–171. Elsevier, 7. September 2018, Fig. 4. Comparison of predicted domain-level organization in polyprotein 1a of new viruses to previously described nidoviruses, S. 164, doi:10.1016/j.virol.2018.08.010, PMID 30199753, PMC 7112036 (freier Volltext) – (englisch, Volltext [PDF; 3,3 MB; abgerufen am 18. Mai 2020] „Coronavirinae“: heute „Orthocoronavirinae“).
- ↑ Khulud Bukhari, Geraldine Mulley, Anastasia A. Gulyaeva, Lanying Zhao, Guocheng Shu, Jianping Jiang, Benjamin W. Neuman: Description and initial characterization of metatranscriptomic nidovirus-like genomes from the proposed new family Abyssoviridae, and from a sister group to the Coronavirinae, the proposed genus Alphaletovirus. In: Virology. Band 524, Ausgabe November 2018, S. 160–171. Elsevier, 7. September 2018, Fig. 5. Comparison of predicted domain-level organization in polyprotein 1b of new viruses to previously described nidoviruses, S. 165, doi:10.1016/j.virol.2018.08.010, PMID 30199753, PMC 7112036 (freier Volltext) – (englisch, Volltext [PDF; 3,3 MB; abgerufen am 18. Mai 2020] „Coronavirinae“: heute „Orthocoronavirinae“).
- ↑ Khulud Bukhari, Geraldine Mulley, Anastasia A. Gulyaeva, Lanying Zhao, Guocheng Shu, Jianping Jiang, Benjamin W. Neuman: Description and initial characterization of metatranscriptomic nidovirus-like genomes from the proposed new family Abyssoviridae, and from a sister group to the Coronavirinae, the proposed genus Alphaletovirus. In: Virology. Band 524, Ausgabe November 2018, S. 160–171. Elsevier, 7. September 2018, Fig. 5. Comparison of predicted domain-level organization in polyprotein 1b of new viruses to previously described nidoviruses, Teilbild A, S. 165, doi:10.1016/j.virol.2018.08.010, PMID 30199753, PMC 7112036 (freier Volltext) – (englisch, Volltext [PDF; 3,3 MB; abgerufen am 18. Mai 2020] „Coronavirinae“: heute „Orthocoronavirinae“).
- ↑ Khulud Bukhari, Geraldine Mulley, Anastasia A. Gulyaeva, Lanying Zhao, Guocheng Shu, Jianping Jiang, Benjamin W. Neuman: Description and initial characterization of metatranscriptomic nidovirus-like genomes from the proposed new family Abyssoviridae, and from a sister group to the Coronavirinae, the proposed genus Alphaletovirus. In: Virology. Band 524, Ausgabe November 2018, S. 160–171. Elsevier, 7. September 2018, Fig. 5. Comparison of predicted domain-level organization in polyprotein 1b of new viruses to previously described nidoviruses, Teilbild B, S. 165, doi:10.1016/j.virol.2018.08.010, PMID 30199753, PMC 7112036 (freier Volltext) – (englisch, Volltext [PDF; 3,3 MB; abgerufen am 18. Mai 2020] „Coronavirinae“: heute „Orthocoronavirinae“).
- ↑ Khulud Bukhari, Geraldine Mulley, Anastasia A. Gulyaeva, Lanying Zhao, Guocheng Shu, Jianping Jiang, Benjamin W. Neuman: Description and initial characterization of metatranscriptomic nidovirus-like genomes from the proposed new family Abyssoviridae, and from a sister group to the Coronavirinae, the proposed genus Alphaletovirus. In: Virology. Band 524, Ausgabe November 2018, S. 160–171. Elsevier, 7. September 2018, Fig. 8. Coding Capacity and prevalence of MLeV, S. 167, doi:10.1016/j.virol.2018.08.010, PMID 30199753, PMC 7112036 (freier Volltext) – (englisch, Volltext [PDF; 3,3 MB; abgerufen am 18. Mai 2020] „Coronavirinae“: heute „Orthocoronavirinae“).
- ↑ Khulud Bukhari, Geraldine Mulley, Anastasia A. Gulyaeva, Lanying Zhao, Guocheng Shu, Jianping Jiang, Benjamin W. Neuman: Description and initial characterization of metatranscriptomic nidovirus-like genomes from the proposed new family Abyssoviridae, and from a sister group to the Coronavirinae, the proposed genus Alphaletovirus. In: Virology. Band 524, Ausgabe November 2018, S. 160–171. Elsevier, 7. September 2018, 1. Introduction, S. 160, doi:10.1016/j.virol.2018.08.010, PMID 30199753, PMC 7112036 (freier Volltext) – (englisch, Volltext [PDF; 3,3 MB; abgerufen am 18. Mai 2020] „Coronavirinae“: heute „Orthocoronavirinae“).
- ↑ Khulud Bukhari, Geraldine Mulley, Anastasia A. Gulyaeva, Lanying Zhao, Guocheng Shu, Jianping Jiang, Benjamin W. Neuman: Description and initial characterization of metatranscriptomic nidovirus-like genomes from the proposed new family Abyssoviridae, and from a sister group to the Coronavirinae, the proposed genus Alphaletovirus. In: Virology. Band 524, Ausgabe November 2018, S. 160–171. Elsevier, 7. September 2018, 2.1. Virus discovery, S. 161, doi:10.1016/j.virol.2018.08.010, PMID 30199753, PMC 7112036 (freier Volltext) – (englisch, Volltext [PDF; 3,3 MB; abgerufen am 18. Mai 2020] „Coronavirinae“: heute „Orthocoronavirinae“).
- ↑ Khulud Bukhari, Geraldine Mulley, Anastasia A. Gulyaeva, Lanying Zhao, Guocheng Shu, Jianping Jiang, Benjamin W. Neuman: Description and initial characterization of metatranscriptomic nidovirus-like genomes from the proposed new family Abyssoviridae, and from a sister group to the Coronavirinae, the proposed genus Alphaletovirus. In: Virology. Band 524, Ausgabe November 2018, S. 160–171. Elsevier, 7. September 2018, 2.2. Phylogenetic analysis, S. 161, doi:10.1016/j.virol.2018.08.010, PMID 30199753, PMC 7112036 (freier Volltext) – (englisch, Volltext [PDF; 3,3 MB; abgerufen am 18. Mai 2020] „Coronavirinae“: heute „Orthocoronavirinae“).
- ↑ Khulud Bukhari, Geraldine Mulley, Anastasia A. Gulyaeva, Lanying Zhao, Guocheng Shu, Jianping Jiang, Benjamin W. Neuman: Description and initial characterization of metatranscriptomic nidovirus-like genomes from the proposed new family Abyssoviridae, and from a sister group to the Coronavirinae, the proposed genus Alphaletovirus. In: Virology. Band 524, Ausgabe November 2018, S. 160–171. Elsevier, 7. September 2018, 2.4. AAbV genome and subgenome sequences and their potential expression, S. 162 f., doi:10.1016/j.virol.2018.08.010, PMID 30199753, PMC 7112036 (freier Volltext) – (englisch, Volltext [PDF; 3,3 MB; abgerufen am 18. Mai 2020] „Coronavirinae“: heute „Orthocoronavirinae“).
- ↑ Khulud Bukhari, Geraldine Mulley, Anastasia A. Gulyaeva, Lanying Zhao, Guocheng Shu, Jianping Jiang, Benjamin W. Neuman: Description and initial characterization of metatranscriptomic nidovirus-like genomes from the proposed new family Abyssoviridae, and from a sister group to the Coronavirinae, the proposed genus Alphaletovirus. In: Virology. Band 524, Ausgabe November 2018, S. 160–171. Elsevier, 7. September 2018, 2.5. AAbV protein bioinformatics, S. 163 f., doi:10.1016/j.virol.2018.08.010, PMID 30199753, PMC 7112036 (freier Volltext) – (englisch, Volltext [PDF; 3,3 MB; abgerufen am 18. Mai 2020] „Coronavirinae“: heute „Orthocoronavirinae“).
- ↑ Khulud Bukhari, Geraldine Mulley, Anastasia A. Gulyaeva, Lanying Zhao, Guocheng Shu, Jianping Jiang, Benjamin W. Neuman: Description and initial characterization of metatranscriptomic nidovirus-like genomes from the proposed new family Abyssoviridae, and from a sister group to the Coronavirinae, the proposed genus Alphaletovirus. In: Virology. Band 524, Ausgabe November 2018, S. 160–171. Elsevier, 7. September 2018, 2.6. AAbV proteinases, S. 164 f., doi:10.1016/j.virol.2018.08.010, PMID 30199753, PMC 7112036 (freier Volltext) – (englisch, Volltext [PDF; 3,3 MB; abgerufen am 18. Mai 2020] „Coronavirinae“: heute „Orthocoronavirinae“).
- ↑ Khulud Bukhari, Geraldine Mulley, Anastasia A. Gulyaeva, Lanying Zhao, Guocheng Shu, Jianping Jiang, Benjamin W. Neuman: Description and initial characterization of metatranscriptomic nidovirus-like genomes from the proposed new family Abyssoviridae, and from a sister group to the Coronavirinae, the proposed genus Alphaletovirus. In: Virology. Band 524, Ausgabe November 2018, S. 160–171. Elsevier, 7. September 2018, 2.7. AAbV pp1ab expression, S. 165, doi:10.1016/j.virol.2018.08.010, PMID 30199753, PMC 7112036 (freier Volltext) – (englisch, Volltext [PDF; 3,3 MB; abgerufen am 18. Mai 2020] „Coronavirinae“: heute „Orthocoronavirinae“).
- ↑ Zitat:
“Hyla comes Herculis, ab eo in Bithinia perditus; sacris postea institutis, ut continuo clamaretur hyla! hyla! hyla! quasi ad eundem repetendum; ad cujus imitationem dixit Virgilius:
Ut littus hyla hyla omne sonaret.
Eccl. VI. 44.
Quam ob rem hæc quasi Hylæ sacerdos nomen ejusdem merita est.”„Hylas, Gefährte Herkules', von ihm in Bithinia verloren, später (Gefährte) eines Heiligtums (mit) der Sitte, wie des quasi immer gleichen Wiederholens der Fortsetzung, dass er „Hyla! Hyla! Hyla!“ gerufen würde. Zu dessen Nachahmung hat Virgil gesagt:
Wie das Meerufer, so erklänge das „Hyla! Hyla!“ aller.
Eccl. VI. 44.
Deswegen ist dies zurecht ebenfalls quasi eine Bezeichnung eines Priesters der Hyla [weiblich!].“– Josephus Nicolaus Laurenti: SPECIMEN MEDICUM, EXHIBENS SYNOPSIN REPTILIUM EMENDATAM CUM EXPERIMENTIS CIRCA VENENA ET ANTIDOTA REPTILIUM AUSTRIACORUM. Wien 1768, S. 32–33, Fußnoten (Latein, uni-goettingen.de [abgerufen am 22. Mai 2020]). (Volltext-Download: uni-goettingen.de [PDF; 17,1 MB; abgerufen am 22. Mai 2020])Dazu:
“His adjungit, Hylan nautæ quo fonte relictum
Clamâssent, ut littus, Hyla! Hyla! omne sonaret;”– Virgil: Eccl. VI. 43–44.Siehe auch: Wikipedia:Auskunft/Archiv/2020/Woche_21#Latein-Hilfe (←später raus)
- ↑ a b Antonio C. P. Wong, Xin Li, Susanna K. P. Lau and Patrick C. Y. Woo: Global Epidemiology of Bat Coronaviruses. Abstract. In: Viruses. Band 11, Nr. 2. MDPI, 20. Februar 2019, S. 174, doi:10.3390/v11020174, PMID 30791586, PMC 6409556 (freier Volltext) – (englisch, Volltext [PDF; 2,2 MB; abgerufen am 31. Mai 2020]).
- ↑ Yi Fan, Kai Zhao, Zheng-Li Shi and Peng Zhou: Bat Coronaviruses in China. In: Viruses. Band 11, Nr. 3. MDPI, 2. März 2019, S. 210, doi:10.3390/v11030210, PMID 30832341, PMC 6466186 (freier Volltext) – (englisch, Volltext [PDF; 0 kB; abgerufen am 16. Juni 2020]).
- ↑ Amir Saberi, Anastasia A. Gulyaeva, John L. Brubacher, Phillip A. Newmark, Alexander E. Gorbalenya: A planarian nidovirus expands the limits of RNA genome size. In: PLOS Pathogens. Band 14, Ausgabe 11, Artikel e1007314, 1. November 2018, Bereich „Abstract“, S. 1, doi:10.1371/journal.ppat.1007314, PMID 30383829, PMC 6211748 (freier Volltext) – (englisch, Volltext [PDF; 4,2 MB; abgerufen am 1. Juni 2020]).
- ↑ Amir Saberi, Anastasia A. Gulyaeva, John L. Brubacher, Phillip A. Newmark, Alexander E. Gorbalenya: A planarian nidovirus expands the limits of RNA genome size. In: PLOS Pathogens. Band 14, Ausgabe 11, Artikel e1007314, 1. November 2018, Bereich „Author summary“, S. 2, doi:10.1371/journal.ppat.1007314, PMID 30383829, PMC 6211748 (freier Volltext) – (englisch, Volltext [PDF; 4,2 MB; abgerufen am 1. Juni 2020]).
- ↑ Amir Saberi, Anastasia A. Gulyaeva, John L. Brubacher, Phillip A. Newmark, Alexander E. Gorbalenya: A planarian nidovirus expands the limits of RNA genome size. In: PLOS Pathogens. Band 14, Ausgabe 11, Artikel e1007314, 1. November 2018, Kapitel „Results“, S. 5–19, doi:10.1371/journal.ppat.1007314, PMID 30383829, PMC 6211748 (freier Volltext) – (englisch, Volltext [PDF; 4,2 MB; abgerufen am 1. Juni 2020]).
- ↑ Amir Saberi, Anastasia A. Gulyaeva, John L. Brubacher, Phillip A. Newmark, Alexander E. Gorbalenya: A planarian nidovirus expands the limits of RNA genome size. In: PLOS Pathogens. Band 14, Ausgabe 11, Artikel e1007314, 1. November 2018, Kapitel „Discussion“, S. 19–24, doi:10.1371/journal.ppat.1007314, PMID 30383829, PMC 6211748 (freier Volltext) – (englisch, Volltext [PDF; 4,2 MB; abgerufen am 1. Juni 2020]).
- ↑ Amir Saberi, Anastasia A. Gulyaeva, John L. Brubacher, Phillip A. Newmark, Alexander E. Gorbalenya: A planarian nidovirus expands the limits of RNA genome size. In: PLOS Pathogens. Band 14, Ausgabe 11, Artikel e1007314, 1. November 2018, Abschnitt „Identification and genomic assembly of a large RNA virus from planarians“, S. 5, doi:10.1371/journal.ppat.1007314, PMID 30383829, PMC 6211748 (freier Volltext) – (englisch, Volltext [PDF; 4,2 MB; abgerufen am 1. Juni 2020]).
- ↑ Amir Saberi, Anastasia A. Gulyaeva, John L. Brubacher, Phillip A. Newmark, Alexander E. Gorbalenya: A planarian nidovirus expands the limits of RNA genome size. In: PLOS Pathogens. Band 14, Ausgabe 11, Artikel e1007314, 1. November 2018, Abschnitt „PSCNV variants in worldwide planarian laboratories imply recent virus transmission“, S. 6, doi:10.1371/journal.ppat.1007314, PMID 30383829, PMC 6211748 (freier Volltext) – (englisch, Volltext [PDF; 4,2 MB; abgerufen am 1. Juni 2020]).
- ↑ Amir Saberi, Anastasia A. Gulyaeva, John L. Brubacher, Phillip A. Newmark, Alexander E. Gorbalenya: A planarian nidovirus expands the limits of RNA genome size. In: PLOS Pathogens. Band 14, Ausgabe 11, Artikel e1007314, 1. November 2018, Abschnitt „PSCNV infects the secretory cells of planarians“, S. 6–9, doi:10.1371/journal.ppat.1007314, PMID 30383829, PMC 6211748 (freier Volltext) – (englisch, Volltext [PDF; 4,2 MB; abgerufen am 1. Juni 2020]).
- ↑ Amir Saberi, Anastasia A. Gulyaeva, John L. Brubacher, Phillip A. Newmark, Alexander E. Gorbalenya: A planarian nidovirus expands the limits of RNA genome size. In: PLOS Pathogens. Band 14, Ausgabe 11, Artikel e1007314, 1. November 2018, Abschnitt „Overview of the PSCNV proteome reveals a unique nidovirus“, S. 9–10, doi:10.1371/journal.ppat.1007314, PMID 30383829, PMC 6211748 (freier Volltext) – (englisch, Volltext [PDF; 4,2 MB; abgerufen am 1. Juni 2020]).
- ↑ Amir Saberi, Anastasia A. Gulyaeva, John L. Brubacher, Phillip A. Newmark, Alexander E. Gorbalenya: A planarian nidovirus expands the limits of RNA genome size. In: PLOS Pathogens. Band 14, Ausgabe 11, Artikel e1007314, 1. November 2018, Abschnitt „Conserved and distinctive features in PSCNV’s replicative and regulatory proteins“, S. 10–12, doi:10.1371/journal.ppat.1007314, PMID 30383829, PMC 6211748 (freier Volltext) – (englisch, Volltext [PDF; 4,2 MB; abgerufen am 1. Juni 2020]).
- ↑ Amir Saberi, Anastasia A. Gulyaeva, John L. Brubacher, Phillip A. Newmark, Alexander E. Gorbalenya: A planarian nidovirus expands the limits of RNA genome size. In: PLOS Pathogens. Band 14, Ausgabe 11, Artikel e1007314, 1. November 2018, Abschnitt „PSCNV encodes protein domains that are novel to nidoviruses“, S. 12, doi:10.1371/journal.ppat.1007314, PMID 30383829, PMC 6211748 (freier Volltext) – (englisch, Volltext [PDF; 4,2 MB; abgerufen am 1. Juni 2020]).
- ↑ Amir Saberi, Anastasia A. Gulyaeva, John L. Brubacher, Phillip A. Newmark, Alexander E. Gorbalenya: A planarian nidovirus expands the limits of RNA genome size. In: PLOS Pathogens. Band 14, Ausgabe 11, Artikel e1007314, 1. November 2018, Abschnitt „Putative structural proteins of PSCNV“, S. 12–14, doi:10.1371/journal.ppat.1007314, PMID 30383829, PMC 6211748 (freier Volltext) – (englisch, Volltext [PDF; 4,2 MB; abgerufen am 1. Juni 2020]).
- ↑ Amir Saberi, Anastasia A. Gulyaeva, John L. Brubacher, Phillip A. Newmark, Alexander E. Gorbalenya: A planarian nidovirus expands the limits of RNA genome size. In: PLOS Pathogens. Band 14, Ausgabe 11, Artikel e1007314, 1. November 2018, Abschnitt „PSCNV clusters with invertebrate nidoviruses in phylogenetic analyses“, S. 14, doi:10.1371/journal.ppat.1007314, PMID 30383829, PMC 6211748 (freier Volltext) – (englisch, Volltext [PDF; 4,2 MB; abgerufen am 1. Juni 2020]).
- ↑ Amir Saberi, Anastasia A. Gulyaeva, John L. Brubacher, Phillip A. Newmark, Alexander E. Gorbalenya: A planarian nidovirus expands the limits of RNA genome size. In: PLOS Pathogens. Band 14, Ausgabe 11, Artikel e1007314, 1. November 2018, Abschnitt „Origin of single-ORF genome organization“, S. 14–15, doi:10.1371/journal.ppat.1007314, PMID 30383829, PMC 6211748 (freier Volltext) – (englisch, Volltext [PDF; 4,2 MB; abgerufen am 1. Juni 2020]).
- ↑ Amir Saberi, Anastasia A. Gulyaeva, John L. Brubacher, Phillip A. Newmark, Alexander E. Gorbalenya: A planarian nidovirus expands the limits of RNA genome size. In: PLOS Pathogens. Band 14, Ausgabe 11, Artikel e1007314, 1. November 2018, Abschnitt „PSCNV expanded disproportionately in the ORF1b-like region“, S. 15–16, doi:10.1371/journal.ppat.1007314, PMID 30383829, PMC 6211748 (freier Volltext) – (englisch, Volltext [PDF; 4,2 MB; abgerufen am 1. Juni 2020]).
- ↑ Amir Saberi, Anastasia A. Gulyaeva, John L. Brubacher, Phillip A. Newmark, Alexander E. Gorbalenya: A planarian nidovirus expands the limits of RNA genome size. In: PLOS Pathogens. Band 14, Ausgabe 11, Artikel e1007314, 1. November 2018, Abschnitt „PSCNV genome features suggest mechanisms to regulate the stoichiometry of proteins encoded by a single-ORF genome“, S. 16–18, doi:10.1371/journal.ppat.1007314, PMID 30383829, PMC 6211748 (freier Volltext) – (englisch, Volltext [PDF; 4,2 MB; abgerufen am 1. Juni 2020]).
- ↑ Amir Saberi, Anastasia A. Gulyaeva, John L. Brubacher, Phillip A. Newmark, Alexander E. Gorbalenya: A planarian nidovirus expands the limits of RNA genome size. In: PLOS Pathogens. Band 14, Ausgabe 11, Artikel e1007314, 1. November 2018, Abschnitt „Identification of partial genome sequences of putative planarian viruses related to PSCNV“, S. 18, doi:10.1371/journal.ppat.1007314, PMID 30383829, PMC 6211748 (freier Volltext) – (englisch, Volltext [PDF; 4,2 MB; abgerufen am 1. Juni 2020]).
- ↑ Amir Saberi, Anastasia A. Gulyaeva, John L. Brubacher, Phillip A. Newmark, Alexander E. Gorbalenya: A planarian nidovirus expands the limits of RNA genome size. In: PLOS Pathogens. Band 14, Ausgabe 11, Artikel e1007314, 1. November 2018, Abschnitt „PSCNV is distantly related to previously described nidoviruses“, S. 20–21, doi:10.1371/journal.ppat.1007314, PMID 30383829, PMC 6211748 (freier Volltext) – (englisch, Volltext [PDF; 4,2 MB; abgerufen am 1. Juni 2020]).
- ↑ Amir Saberi, Anastasia A. Gulyaeva, John L. Brubacher, Phillip A. Newmark, Alexander E. Gorbalenya: A planarian nidovirus expands the limits of RNA genome size. In: PLOS Pathogens. Band 14, Ausgabe 11, Artikel e1007314, 1. November 2018, Abschnitt „The PSCNV single-ORF genome may be expressed in a manner similar to that of multi-ORF nidoviruses“, S. 21–22, doi:10.1371/journal.ppat.1007314, PMID 30383829, PMC 6211748 (freier Volltext) – (englisch, Volltext [PDF; 4,2 MB; abgerufen am 1. Juni 2020]).
- ↑ Amir Saberi, Anastasia A. Gulyaeva, John L. Brubacher, Phillip A. Newmark, Alexander E. Gorbalenya: A planarian nidovirus expands the limits of RNA genome size. In: PLOS Pathogens. Band 14, Ausgabe 11, Artikel e1007314, 1. November 2018, Abschnitt „PSCNV has acquired novel proteins with potential functions in host-virus interactions“, S. 22–23, doi:10.1371/journal.ppat.1007314, PMID 30383829, PMC 6211748 (freier Volltext) – (englisch, Volltext [PDF; 4,2 MB; abgerufen am 1. Juni 2020]).
- ↑ Amir Saberi, Anastasia A. Gulyaeva, John L. Brubacher, Phillip A. Newmark, Alexander E. Gorbalenya: A planarian nidovirus expands the limits of RNA genome size. In: PLOS Pathogens. Band 14, Ausgabe 11, Artikel e1007314, 1. November 2018, Abschnitt „Emergence and evolution of the PSCNV genome: implications for the viability of large RNA genomes“, S. 23–24, doi:10.1371/journal.ppat.1007314, PMID 30383829, PMC 6211748 (freier Volltext) – (englisch, Volltext [PDF; 4,2 MB; abgerufen am 1. Juni 2020]).
- ↑ Amir Saberi, Anastasia A. Gulyaeva, John L. Brubacher, Phillip A. Newmark, Alexander E. Gorbalenya: A planarian nidovirus expands the limits of RNA genome size. In: PLOS Pathogens. Band 14, Ausgabe 11, Artikel e1007314, 1. November 2018, Unterabschnitt „3CL protease (main protease of polyprotein processing)“, S. 10–11, doi:10.1371/journal.ppat.1007314, PMID 30383829, PMC 6211748 (freier Volltext) – (englisch, Volltext [PDF; 4,2 MB; abgerufen am 1. Juni 2020]).
- ↑ Amir Saberi, Anastasia A. Gulyaeva, John L. Brubacher, Phillip A. Newmark, Alexander E. Gorbalenya: A planarian nidovirus expands the limits of RNA genome size. In: PLOS Pathogens. Band 14, Ausgabe 11, Artikel e1007314, 1. November 2018, Unterabschnitt „NiRAN, RdRp, ZBD, HEL1 (RNA replicative enzyme domains)“, S. 11, doi:10.1371/journal.ppat.1007314, PMID 30383829, PMC 6211748 (freier Volltext) – (englisch, Volltext [PDF; 4,2 MB; abgerufen am 1. Juni 2020]).
- ↑ Amir Saberi, Anastasia A. Gulyaeva, John L. Brubacher, Phillip A. Newmark, Alexander E. Gorbalenya: A planarian nidovirus expands the limits of RNA genome size. In: PLOS Pathogens. Band 14, Ausgabe 11, Artikel e1007314, 1. November 2018, Unterabschnitt „ExoN, N-MT, O-MT (proofreading and RNA-modifying enzyme domains)“, S. 11–12, doi:10.1371/journal.ppat.1007314, PMID 30383829, PMC 6211748 (freier Volltext) – (englisch, Volltext [PDF; 4,2 MB; abgerufen am 1. Juni 2020]).
- ↑ Amir Saberi, Anastasia A. Gulyaeva, John L. Brubacher, Phillip A. Newmark, Alexander E. Gorbalenya: A planarian nidovirus expands the limits of RNA genome size. In: PLOS Pathogens. Band 14, Ausgabe 11, Artikel e1007314, 1. November 2018, Unterabschnitt „RNase T2“, S. 12, doi:10.1371/journal.ppat.1007314, PMID 30383829, PMC 6211748 (freier Volltext) – (englisch, Volltext [PDF; 4,2 MB; abgerufen am 1. Juni 2020]).
- ↑ Amir Saberi, Anastasia A. Gulyaeva, John L. Brubacher, Phillip A. Newmark, Alexander E. Gorbalenya: A planarian nidovirus expands the limits of RNA genome size. In: PLOS Pathogens. Band 14, Ausgabe 11, Artikel e1007314, 1. November 2018, Unterabschnitt „Fibronectin type II (FN2) domains“, S. 12, doi:10.1371/journal.ppat.1007314, PMID 30383829, PMC 6211748 (freier Volltext) – (englisch, Volltext [PDF; 4,2 MB; abgerufen am 1. Juni 2020]).
- ↑ Amir Saberi, Anastasia A. Gulyaeva, John L. Brubacher, Phillip A. Newmark, Alexander E. Gorbalenya: A planarian nidovirus expands the limits of RNA genome size. In: PLOS Pathogens. Band 14, Ausgabe 11, Artikel e1007314, 1. November 2018, Unterabschnitt „Ankyrins“, S. 12, doi:10.1371/journal.ppat.1007314, PMID 30383829, PMC 6211748 (freier Volltext) – (englisch, Volltext [PDF; 4,2 MB; abgerufen am 1. Juni 2020]).
- ↑ Amir Saberi, Anastasia A. Gulyaeva, John L. Brubacher, Phillip A. Newmark, Alexander E. Gorbalenya: A planarian nidovirus expands the limits of RNA genome size. In: PLOS Pathogens. Band 14, Ausgabe 11, Artikel e1007314, 1. November 2018, Unterabschnitt „Genome translation and frameshifting“, S. 17–18, doi:10.1371/journal.ppat.1007314, PMID 30383829, PMC 6211748 (freier Volltext) – (englisch, Volltext [PDF; 4,2 MB; abgerufen am 1. Juni 2020]).
- ↑ Amir Saberi, Anastasia A. Gulyaeva, John L. Brubacher, Phillip A. Newmark, Alexander E. Gorbalenya: A planarian nidovirus expands the limits of RNA genome size. In: PLOS Pathogens. Band 14, Ausgabe 11, Artikel e1007314, 1. November 2018, Unterabschnitt „Discontinuous genome synthesis (transcription)“, S. 18, doi:10.1371/journal.ppat.1007314, PMID 30383829, PMC 6211748 (freier Volltext) – (englisch, Volltext [PDF; 4,2 MB; abgerufen am 1. Juni 2020]).
- ↑ Amir Saberi, Anastasia A. Gulyaeva, John L. Brubacher, Phillip A. Newmark, Alexander E. Gorbalenya: A planarian nidovirus expands the limits of RNA genome size. In: PLOS Pathogens. Band 14, Ausgabe 11, Artikel e1007314, 1. November 2018, Fig. 1. Genome sizes of nidoviruses, S. 3, doi:10.1371/journal.ppat.1007314, PMID 30383829, PMC 6211748 (freier Volltext) – (englisch, Volltext [PDF; 4,2 MB; abgerufen am 1. Juni 2020]).
- ↑ Amir Saberi, Anastasia A. Gulyaeva, John L. Brubacher, Phillip A. Newmark, Alexander E. Gorbalenya: A planarian nidovirus expands the limits of RNA genome size. In: PLOS Pathogens. Band 14, Ausgabe 11, Artikel e1007314, 1. November 2018, Fig. 1. Genome sizes of nidoviruses, Teilbild A, S. 3, doi:10.1371/journal.ppat.1007314, PMID 30383829, PMC 6211748 (freier Volltext) – (englisch, Volltext [PDF; 4,2 MB; abgerufen am 1. Juni 2020]).
- ↑ Amir Saberi, Anastasia A. Gulyaeva, John L. Brubacher, Phillip A. Newmark, Alexander E. Gorbalenya: A planarian nidovirus expands the limits of RNA genome size. In: PLOS Pathogens. Band 14, Ausgabe 11, Artikel e1007314, 1. November 2018, Fig. 1. Genome sizes of nidoviruses, Teilbild B, S. 3, doi:10.1371/journal.ppat.1007314, PMID 30383829, PMC 6211748 (freier Volltext) – (englisch, Volltext [PDF; 4,2 MB; abgerufen am 1. Juni 2020]).
- ↑ Amir Saberi, Anastasia A. Gulyaeva, John L. Brubacher, Phillip A. Newmark, Alexander E. Gorbalenya: A planarian nidovirus expands the limits of RNA genome size. In: PLOS Pathogens. Band 14, Ausgabe 11, Artikel e1007314, 1. November 2018, Fig. 2. Genomes and proteomes of nidoviruses, S. 4, doi:10.1371/journal.ppat.1007314, PMID 30383829, PMC 6211748 (freier Volltext) – (englisch, Volltext [PDF; 4,2 MB; abgerufen am 1. Juni 2020]).
- ↑ Amir Saberi, Anastasia A. Gulyaeva, John L. Brubacher, Phillip A. Newmark, Alexander E. Gorbalenya: A planarian nidovirus expands the limits of RNA genome size. In: PLOS Pathogens. Band 14, Ausgabe 11, Artikel e1007314, 1. November 2018, Fig. 3. Expression of PSCNV RNA in planarians, S. 7, doi:10.1371/journal.ppat.1007314, PMID 30383829, PMC 6211748 (freier Volltext) – (englisch, Volltext [PDF; 4,2 MB; abgerufen am 1. Juni 2020]).
- ↑ Amir Saberi, Anastasia A. Gulyaeva, John L. Brubacher, Phillip A. Newmark, Alexander E. Gorbalenya: A planarian nidovirus expands the limits of RNA genome size. In: PLOS Pathogens. Band 14, Ausgabe 11, Artikel e1007314, 1. November 2018, Fig. 3. Expression of PSCNV RNA in planarians, Teilbild A, S. 7, doi:10.1371/journal.ppat.1007314, PMID 30383829, PMC 6211748 (freier Volltext) – (englisch, Volltext [PDF; 4,2 MB; abgerufen am 1. Juni 2020]).
- ↑ Amir Saberi, Anastasia A. Gulyaeva, John L. Brubacher, Phillip A. Newmark, Alexander E. Gorbalenya: A planarian nidovirus expands the limits of RNA genome size. In: PLOS Pathogens. Band 14, Ausgabe 11, Artikel e1007314, 1. November 2018, Fig. 3. Expression of PSCNV RNA in planarians, Teilbild B, S. 7, doi:10.1371/journal.ppat.1007314, PMID 30383829, PMC 6211748 (freier Volltext) – (englisch, Volltext [PDF; 4,2 MB; abgerufen am 1. Juni 2020]).
- ↑ Amir Saberi, Anastasia A. Gulyaeva, John L. Brubacher, Phillip A. Newmark, Alexander E. Gorbalenya: A planarian nidovirus expands the limits of RNA genome size. In: PLOS Pathogens. Band 14, Ausgabe 11, Artikel e1007314, 1. November 2018, Fig. 3. Expression of PSCNV RNA in planarians, Teilbild C, S. 7, doi:10.1371/journal.ppat.1007314, PMID 30383829, PMC 6211748 (freier Volltext) – (englisch, Volltext [PDF; 4,2 MB; abgerufen am 1. Juni 2020]).
- ↑ Amir Saberi, Anastasia A. Gulyaeva, John L. Brubacher, Phillip A. Newmark, Alexander E. Gorbalenya: A planarian nidovirus expands the limits of RNA genome size. In: PLOS Pathogens. Band 14, Ausgabe 11, Artikel e1007314, 1. November 2018, Fig. 4. Putative PSCNV particles revealed by electron microscopy, S. 8–9, doi:10.1371/journal.ppat.1007314, PMID 30383829, PMC 6211748 (freier Volltext) – (englisch, Volltext [PDF; 4,2 MB; abgerufen am 1. Juni 2020]).
- ↑ Amir Saberi, Anastasia A. Gulyaeva, John L. Brubacher, Phillip A. Newmark, Alexander E. Gorbalenya: A planarian nidovirus expands the limits of RNA genome size. In: PLOS Pathogens. Band 14, Ausgabe 11, Artikel e1007314, 1. November 2018, Fig. 4. Putative PSCNV particles revealed by electron microscopy, Teilbild A, S. 8–9, doi:10.1371/journal.ppat.1007314, PMID 30383829, PMC 6211748 (freier Volltext) – (englisch, Volltext [PDF; 4,2 MB; abgerufen am 1. Juni 2020]).
- ↑ Amir Saberi, Anastasia A. Gulyaeva, John L. Brubacher, Phillip A. Newmark, Alexander E. Gorbalenya: A planarian nidovirus expands the limits of RNA genome size. In: PLOS Pathogens. Band 14, Ausgabe 11, Artikel e1007314, 1. November 2018, Fig. 4. Putative PSCNV particles revealed by electron microscopy, Teilbild B, S. 8–9, doi:10.1371/journal.ppat.1007314, PMID 30383829, PMC 6211748 (freier Volltext) – (englisch, Volltext [PDF; 4,2 MB; abgerufen am 1. Juni 2020]).
- ↑ Amir Saberi, Anastasia A. Gulyaeva, John L. Brubacher, Phillip A. Newmark, Alexander E. Gorbalenya: A planarian nidovirus expands the limits of RNA genome size. In: PLOS Pathogens. Band 14, Ausgabe 11, Artikel e1007314, 1. November 2018, Fig. 4. Putative PSCNV particles revealed by electron microscopy, Teilbild C, S. 8–9, doi:10.1371/journal.ppat.1007314, PMID 30383829, PMC 6211748 (freier Volltext) – (englisch, Volltext [PDF; 4,2 MB; abgerufen am 1. Juni 2020]).
- ↑ Amir Saberi, Anastasia A. Gulyaeva, John L. Brubacher, Phillip A. Newmark, Alexander E. Gorbalenya: A planarian nidovirus expands the limits of RNA genome size. In: PLOS Pathogens. Band 14, Ausgabe 11, Artikel e1007314, 1. November 2018, Fig. 4. Putative PSCNV particles revealed by electron microscopy, Teilbild D, S. 8–9, doi:10.1371/journal.ppat.1007314, PMID 30383829, PMC 6211748 (freier Volltext) – (englisch, Volltext [PDF; 4,2 MB; abgerufen am 1. Juni 2020]).
- ↑ Amir Saberi, Anastasia A. Gulyaeva, John L. Brubacher, Phillip A. Newmark, Alexander E. Gorbalenya: A planarian nidovirus expands the limits of RNA genome size. In: PLOS Pathogens. Band 14, Ausgabe 11, Artikel e1007314, 1. November 2018, Fig. 4. Putative PSCNV particles revealed by electron microscopy, Teilbild E, S. 8–9, doi:10.1371/journal.ppat.1007314, PMID 30383829, PMC 6211748 (freier Volltext) – (englisch, Volltext [PDF; 4,2 MB; abgerufen am 1. Juni 2020]).
- ↑ Amir Saberi, Anastasia A. Gulyaeva, John L. Brubacher, Phillip A. Newmark, Alexander E. Gorbalenya: A planarian nidovirus expands the limits of RNA genome size. In: PLOS Pathogens. Band 14, Ausgabe 11, Artikel e1007314, 1. November 2018, Fig. 5. Largest proteins of nidoviruses and other RNA viruses in comparison with PSCNV polyprotein, S. 10, doi:10.1371/journal.ppat.1007314, PMID 30383829, PMC 6211748 (freier Volltext) – (englisch, Volltext [PDF; 4,2 MB; abgerufen am 1. Juni 2020]).
- ↑ Amir Saberi, Anastasia A. Gulyaeva, John L. Brubacher, Phillip A. Newmark, Alexander E. Gorbalenya: A planarian nidovirus expands the limits of RNA genome size. In: PLOS Pathogens. Band 14, Ausgabe 11, Artikel e1007314, 1. November 2018, Fig. 6. ANK domain of PSCNV and its homologs, S. 13, doi:10.1371/journal.ppat.1007314, PMID 30383829, PMC 6211748 (freier Volltext) – (englisch, Volltext [PDF; 4,2 MB; abgerufen am 1. Juni 2020]).
- ↑ Amir Saberi, Anastasia A. Gulyaeva, John L. Brubacher, Phillip A. Newmark, Alexander E. Gorbalenya: A planarian nidovirus expands the limits of RNA genome size. In: PLOS Pathogens. Band 14, Ausgabe 11, Artikel e1007314, 1. November 2018, Fig. 7. Phylogeny of PSCNV, S. 15, doi:10.1371/journal.ppat.1007314, PMID 30383829, PMC 6211748 (freier Volltext) – (englisch, Volltext [PDF; 4,2 MB; abgerufen am 1. Juni 2020]).
- ↑ Amir Saberi, Anastasia A. Gulyaeva, John L. Brubacher, Phillip A. Newmark, Alexander E. Gorbalenya: A planarian nidovirus expands the limits of RNA genome size. In: PLOS Pathogens. Band 14, Ausgabe 11, Artikel e1007314, 1. November 2018, Fig. 8. Nidovirus genome and region size differences, S. 16, doi:10.1371/journal.ppat.1007314, PMID 30383829, PMC 6211748 (freier Volltext) – (englisch, Volltext [PDF; 4,2 MB; abgerufen am 1. Juni 2020]).
- ↑ Amir Saberi, Anastasia A. Gulyaeva, John L. Brubacher, Phillip A. Newmark, Alexander E. Gorbalenya: A planarian nidovirus expands the limits of RNA genome size. In: PLOS Pathogens. Band 14, Ausgabe 11, Artikel e1007314, 1. November 2018, Fig. 8. Nidovirus genome and region size differences, Teilbild A, S. 16, doi:10.1371/journal.ppat.1007314, PMID 30383829, PMC 6211748 (freier Volltext) – (englisch, Volltext [PDF; 4,2 MB; abgerufen am 1. Juni 2020]).
- ↑ Amir Saberi, Anastasia A. Gulyaeva, John L. Brubacher, Phillip A. Newmark, Alexander E. Gorbalenya: A planarian nidovirus expands the limits of RNA genome size. In: PLOS Pathogens. Band 14, Ausgabe 11, Artikel e1007314, 1. November 2018, Fig. 8. Nidovirus genome and region size differences, Teilbild B, S. 16, doi:10.1371/journal.ppat.1007314, PMID 30383829, PMC 6211748 (freier Volltext) – (englisch, Volltext [PDF; 4,2 MB; abgerufen am 1. Juni 2020]).
- ↑ Amir Saberi, Anastasia A. Gulyaeva, John L. Brubacher, Phillip A. Newmark, Alexander E. Gorbalenya: A planarian nidovirus expands the limits of RNA genome size. In: PLOS Pathogens. Band 14, Ausgabe 11, Artikel e1007314, 1. November 2018, Fig. 9. Genome translation, S. 17, doi:10.1371/journal.ppat.1007314, PMID 30383829, PMC 6211748 (freier Volltext) – (englisch, Volltext [PDF; 4,2 MB; abgerufen am 1. Juni 2020]).
- ↑ Amir Saberi, Anastasia A. Gulyaeva, John L. Brubacher, Phillip A. Newmark, Alexander E. Gorbalenya: A planarian nidovirus expands the limits of RNA genome size. In: PLOS Pathogens. Band 14, Ausgabe 11, Artikel e1007314, 1. November 2018, Fig. 10. Genome transcription, S. 19, doi:10.1371/journal.ppat.1007314, PMID 30383829, PMC 6211748 (freier Volltext) – (englisch, Volltext [PDF; 4,2 MB; abgerufen am 1. Juni 2020]).
- ↑ Amir Saberi, Anastasia A. Gulyaeva, John L. Brubacher, Phillip A. Newmark, Alexander E. Gorbalenya: A planarian nidovirus expands the limits of RNA genome size. In: PLOS Pathogens. Band 14, Ausgabe 11, Artikel e1007314, 1. November 2018, Fig. 10. Genome transcription, Teilbild A, S. 19, doi:10.1371/journal.ppat.1007314, PMID 30383829, PMC 6211748 (freier Volltext) – (englisch, Volltext [PDF; 4,2 MB; abgerufen am 1. Juni 2020]).
- ↑ Amir Saberi, Anastasia A. Gulyaeva, John L. Brubacher, Phillip A. Newmark, Alexander E. Gorbalenya: A planarian nidovirus expands the limits of RNA genome size. In: PLOS Pathogens. Band 14, Ausgabe 11, Artikel e1007314, 1. November 2018, Fig. 10. Genome transcription, Teilbild B, S. 19, doi:10.1371/journal.ppat.1007314, PMID 30383829, PMC 6211748 (freier Volltext) – (englisch, Volltext [PDF; 4,2 MB; abgerufen am 1. Juni 2020]).
- ↑ Amir Saberi, Anastasia A. Gulyaeva, John L. Brubacher, Phillip A. Newmark, Alexander E. Gorbalenya: A planarian nidovirus expands the limits of RNA genome size. In: PLOS Pathogens. Band 14, Ausgabe 11, Artikel e1007314, 1. November 2018, Fig. 10. Genome transcription, Teilbild C, S. 19, doi:10.1371/journal.ppat.1007314, PMID 30383829, PMC 6211748 (freier Volltext) – (englisch, Volltext [PDF; 4,2 MB; abgerufen am 1. Juni 2020]).
- ↑ ICTV Master Species List 2018b.v2. MSL #34, März 2019
- ↑ J. Ziebuhr, S. Baker, R.S. Baric, R.J. de Groot, C. Drosten, A. Gulyaeva, B.L. Haagmans, B.W. Neuman, S. Perlman, L.L.M. Poon, I. Sola, A.E. Gorbalenya: 2019.006G. Proposal. In: Virus Taxonomy. History. Revision 2019, Juli. International Committee on Taxonomy of Viruses (ICTV), April 2019, Proposal-Code 2019.021S (englisch, ictvonline.org [ZIP; 725 kB; abgerufen am 9. Juni 2020] ratifiziert im März 2020, noch nicht vollständig veröffentlicht).
- ↑ Datenbankeintrag und relevantes Proposal:
- Eintrag: ICTV Taxonomy history: Severe acute respiratory syndrome-related coronavirus. In: ICTV-Homepage. International Committee on Taxonomy of Viruses (ICTV), abgerufen am 7. Mai 2020 (englisch).
- Proposal: 2008.085-126V. (PDF; 175 KiB) In: ICTV-Homepage. International Committee on Taxonomy of Viruses (ICTV), S.
23 [2008.105V], 34 [2008.119V] und 36 [2008.121V], abgerufen am 7. Mai 2020 (englisch).
- ↑
Datenbankeintrag und zugehöriger Revisionsvorschlag:
- EINTRAG: ICTV Taxonomy history: Severe acute respiratory syndrome-related coronavirus. In: Virus Taxonomy. International Committee on Taxonomy of Viruses (ICTV), abgerufen am 7. Mai 2020 (englisch).
- PROPOSAL: Raoul J. de Groot, John Ziebuhr, Leo L. Poon, Patrick C.Woo, Pierre Talbot, Peter J.M. Rottier, Kathryn V. Holmes, Ralph Baric, Stanley Perlman, Luis Enjuanes, Alexander E. Gorbalenya: Revision of the family Coronaviridae. Proposal. In: Virus Taxonomy. History. Revision 2009, June. International Committee on Taxonomy of Viruses (ICTV), 2008, Proposal-Code 2008.085-126V (englisch, ictvonline.org [PDF; 175 kB; abgerufen am 5. Mai 2020]).
- ↑
Datenbankeintrag und zugehöriger Revisionsvorschlag:
- EINTRAG: ICTV Taxonomy history: Planidovirus 1. In: Virus Taxonomy. International Committee on Taxonomy of Viruses (ICTV), abgerufen am 22. Mai 2020 (englisch).
- PROPOSAL: J. Ziebuhr, R.S. Baric, S. Baker, R.J. de Groot, C. Drosten, A. Gulyaeva, B.L. Haagmans, B.W. Neuman, S. Perlman, L.L.M. Poon, I. Sola, A.E. Gorbalenya: Reorganization of the family Coronaviridae into two families, Coronaviridae (including the current subfamily Coronavirinae and the new subfamily Letovirinae) and the new family Tobaniviridae (accommodating the current subfamily Torovirinae and three other subfamilies), revision of the genus rank structure and introduction of a new subgenus rank. Proposal. In: Virus Taxonomy. History. Revision 2018a, July. International Committee on Taxonomy of Viruses (ICTV), 18. Februar 2017, Proposal-Code 2017.013S (englisch, ictvonline.org [ZIP; 5,1 MB; abgerufen am 7. Mai 2020]).
- ↑ J. Ziebuhr, R.S. Baric, S. Baker, R.J. de Groot, C. Drosten, A. Gulyaeva, B.L. Haagmans, B.W. Neuman, S. Perlman, L.L.M. Poon, I. Sola, A.E. Gorbalenya: 2017.012_015S.A.v1.Nidovirales. Proposal. In: Virus Taxonomy. History. Revision 2018a, July. International Committee on Taxonomy of Viruses (ICTV), 18. Februar 2017, Proposal-Code 2017.012_015S (englisch, ictvonline.org [ZIP; 5,1 MB; abgerufen am 7. Mai 2020]). TEST1
- ↑ Spreadsheet 2017.012-015S zu ICTV-Revision № 2018a: 2017.012-015S. (Eine XLSX-Datei in ZIP-Ordner (mit mehreren Dateien); 4,49 MiB) In: ICTV-Homepage. International Committee on Taxonomy of Viruses (ICTV), abgerufen am 9. Juni 2020 (englisch).
- ↑ Can I translate virus names into my own language? In: ICTV-Homepage. International Committee on Taxonomy of Viruses (ICTV), 11. August 2017, abgerufen am 7. Mai 2020 (englisch).
- ↑ Autoren des neunten ICTV-Reports: Virus Taxonomy – Classification and Nomenclature of Viruses. Online-Ausgabe. In: ICTV Reports. International Committee on Taxonomy of Viruses, 2011, abgerufen am 12. Juni 2020 (englisch, parallel archiviert am 2. April 2019 auf web.archive.org.).
- ↑ Autoren des neunten ICTV-Reports: Virus Taxonomy – Classification and Nomenclature of Viruses. Online-Ausgabe. Kapitel: „Introduction“. In: ICTV Reports. International Committee on Taxonomy of Viruses, 2011, abgerufen am 12. Juni 2020 (englisch, parallel archiviert am 2. April 2019 auf web.archive.org.).
- ↑ Autoren des neunten ICTV-Reports: Virus Taxonomy – Classification and Nomenclature of Viruses. Online-Ausgabe. Einleitungskapitel: „9th Report Introduction“. In: ICTV Reports. International Committee on Taxonomy of Viruses, 2011, abgerufen am 12. Juni 2020 (englisch, parallel archiviert am 2. April 2019 auf web.archive.org.).
- ↑ Autoren des neunten ICTV-Reports: Virus Taxonomy – Classification and Nomenclature of Viruses. Online-Ausgabe. Einleitungskapitel: „Preface and Acknowledgements“. In: ICTV Reports. International Committee on Taxonomy of Viruses, 2011, abgerufen am 12. Juni 2020 (englisch, parallel archiviert am 2. April 2019 auf web.archive.org.).
- ↑ Autoren des neunten ICTV-Reports: Virus Taxonomy – Classification and Nomenclature of Viruses. Online-Ausgabe. Einleitungskapitel: „Introduction to Virus Taxonomy“. In: ICTV Reports. International Committee on Taxonomy of Viruses, 2011, abgerufen am 12. Juni 2020 (englisch, parallel archiviert am 2. April 2019 auf web.archive.org.).
- ↑ Autoren des neunten ICTV-Reports: Virus Taxonomy – Classification and Nomenclature of Viruses. Online-Ausgabe. Einleitungskapitel: „Editors and Contributors“. In: ICTV Reports. International Committee on Taxonomy of Viruses, 2011, abgerufen am 12. Juni 2020 (englisch, parallel archiviert am 2. April 2019 auf web.archive.org.).
- ↑ Autoren des neunten ICTV-Reports: Virus Taxonomy – Classification and Nomenclature of Viruses. Online-Ausgabe. Einleitungskapitel: „Table of Contents“. In: ICTV Reports. International Committee on Taxonomy of Viruses, 2011, abgerufen am 12. Juni 2020 (englisch, parallel archiviert am 2. April 2019 auf web.archive.org.).
- ↑ Autoren des neunten ICTV-Reports: Virus Taxonomy – Classification and Nomenclature of Viruses. Online-Ausgabe. Einleitungskapitel: „Order of Presentation“. In: ICTV Reports. International Committee on Taxonomy of Viruses, 2011, abgerufen am 12. Juni 2020 (englisch, parallel archiviert am 2. April 2019 auf web.archive.org.).
- ↑ Autoren des neunten ICTV-Reports: Virus Taxonomy – Classification and Nomenclature of Viruses. Online-Ausgabe. Einleitungskapitel: „Virus Diagrams“. In: ICTV Reports. International Committee on Taxonomy of Viruses, 2011, abgerufen am 12. Juni 2020 (englisch, parallel archiviert am 2. April 2019 auf web.archive.org.).