Boolesches Netzwerk
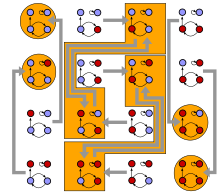
Boolesche Netzwerke bezeichnen ein Modell aus der Statistischen Physik. Sie können als Generalisierung des Ising-Modells verstanden werden, nämlich als eine Spin-Dynamik auf einem Digraph . Jedem Knoten sind dabei sowohl ein boolescher Zustand (oder Spin) als auch eine Boolesche Funktion über die Zustände der eingehenden Knoten zugeordnet.
Diese Aktualisierungsregeln definieren die Systemdynamik, die trotz einfacher Regeln nicht trivial ist. Der Arzt Stuart Kauffman war 1969 der erste Wissenschaftler, der Boolesche Netzwerke als Modell für genetische Netzwerke vorschlug[1] und zwar für den Spezialfall, dass es Knoten (oder Gene) gibt die jeweils exakt von anderen Knoten abhängen, deswegen wird diese Variation auch --Modell genannt. Obwohl Boolesche Netzwerke durch einfache Regeln definiert sind, wurde die Systemdynamik erst nach 2000 mathematisch verstanden.[2]
Obwohl Boolesche Netzwerke eine starke Vereinfachung darstellen (Gene sind nie einfach nur an oder aus), gibt es viele Beispiele, in denen ein Boolesches Modell die richtige Abfolge der Schaltereignisse in einem genetischen Netzwerk vorhersagt.[3][4]
Mathematischer Hintergrund
[Bearbeiten | Quelltext bearbeiten]Netzwerke sind eine Möglichkeit, die Eigenschaften komplexer Systeme zu untersuchen. Die Topologie eines Graphen spiegelt dabei wider, wie die verschiedenen Einheiten des Systems miteinander wechselwirken. Eine zusätzlich zu der Topologie definierte Dynamik kann dabei entweder die Kanten des Graphen im Laufe der Zeit verändern (Dynamik vom Netzwerk) oder nur die Werte der Knoten (Dynamik auf dem Netzwerk). Bei Booleschen Netzwerken handelt es sich um die zweite Variante.
Eine wichtige Fragestellung bei der Untersuchung Boolescher Netzwerke ist, die globalen Eigenschaften eines gegebenen Ensembles zu analysieren. Die Zahl und die Länge der Attraktoren sind eine Möglichkeit, die globalen Eigenschaften zu quantifizieren. Die Herausforderung ist dabei, dass der Zustandsraum extrem schnell wächst, nämlich mit . Auch mit modernen Computern sind die Speichergrenzen schnell erreicht. Das einfachste Ensemble sind die zufälligen Booleschen Netzwerke (englisch: Random Boolean Networks, RBN). Hier ist die Graph-Topologie ein Erdős-Rényi-Zufallsgraph. Für RBNS gab es viele Versuche, die Verteilung der Attraktoren mittels Mittelwerte in Abhängigkeit von der Anzahl der Knoten zu charakterisieren.
| Autoren | Jahr | Mittlere Attraktorlänge | Mittlere Attraktorzahl |
|---|---|---|---|
| Kauffman[1] | 1969 | ||
| Bastolla/Parisi[5] | 1998 | schneller als ein Potenzgesetz, | schneller als ein Potenzgesetz, |
| Bilke/Sjunnesson[6] | 2002 | linear mit der Systemgröße, | |
| Socolar/Kauffman[7] | 2003 | schneller als linear, with | |
| Samuelsson/Troein[8] | 2003 | Beweis des superpolynomiales Wachstum, | |
| Mihaljev/Drossel[9] | 2005 | analytisches Argument für superpolynomiales Wachstum, | analytisches Argument für superpolynomiales Wachstum, |
Einzelnachweise
[Bearbeiten | Quelltext bearbeiten]- ↑ a b Stuart Kauffman: Homeostasis and Differentiation in Random Genetic Control Networks. In: Nature. 224. Jahrgang, Nr. 5215, 11. Oktober 1969, S. 177–178, doi:10.1038/224177a0.
- ↑ Barbara Drossel: Chapter 3. Random Boolean Networks. In: Cornell University. Dezember 2009, doi:10.1002/9783527626359.ch3, arxiv:0706.3351v2.
- ↑ Réka Albert, Hans G Othmer: The topology of the regulatory interactions predicts the expression pattern of the segment polarity genes in Drosophila melanogaster. In: Journal of Theoretical Biology. 223. Jahrgang, Nr. 1, Juli 2003, S. 1–18, doi:10.1016/S0022-5193(03)00035-3.
- ↑ J. Li, A. J. Bench, G. S. Vassiliou, N. Fourouclas, A. C. Ferguson-Smith, A. R. Green: Imprinting of the human L3MBTL gene, a polycomb family member located in a region of chromosome 20 deleted in human myeloid malignancies. In: Proceedings of the National Academy of Sciences. 101. Jahrgang, Nr. 19, 30. April 2004, S. 7341–7346, doi:10.1073/pnas.0308195101.
- ↑ U. Bastolla, G. Parisi: The modular structure of Kauffman networks. In: Physica D: Nonlinear Phenomena. 115. Jahrgang, Nr. 3–4, Mai 1998, S. 219–233, doi:10.1016/S0167-2789(97)00242-X.
- ↑ Sven Bilke, Fredrik Sjunnesson: Stability of the Kauffman model. In: Physical Review E. 65. Jahrgang, Nr. 1, Dezember 2001, doi:10.1103/PhysRevE.65.016129.
- ↑ J. Socolar, S. Kauffman: Scaling in Ordered and Critical Random Boolean Networks. In: Physical Review Letters. 90. Jahrgang, Nr. 6, Februar 2003, doi:10.1103/PhysRevLett.90.068702, arxiv:cond-mat/0212306.
- ↑ Björn Samuelsson, Carl Troein: Superpolynomial Growth in the Number of Attractors in Kauffman Networks. In: Physical Review Letters. 90. Jahrgang, Nr. 9, März 2003, doi:10.1103/PhysRevLett.90.098701.
- ↑ Tamara Mihaljev, Barbara Drossel: Scaling in a general class of critical random Boolean networks. In: Physical Review E. 74. Jahrgang, Nr. 4, Oktober 2006, doi:10.1103/PhysRevE.74.046101.













