Elongation (Transkription)
| Übergeordnet |
|---|
| Transkription RNA-Biosynthese |
| Gene Ontology |
| QuickGO |
Die Elongation während der Transkription behandelt den Prozess, durch welchen der Großteil des den Gencode enthaltenden DNA-Stranges in eine mRNA kopiert wird. Dies ist die letzte Phase der Transkription, nachdem sich der Präinitiationskomplex teilweise aufgelöst und die mRNA eine 5'-Cap-Struktur erhalten hat. Das Transkript ist zu diesem Zeitpunkt etwa 10 bis 30 Basen lang und die Cap-Struktur bindet bereits jetzt den Cap-Binding-Komplex.
In der Elongationsphase ist RNA-Polymerase II Teil des Elongationskomplexes, der sich durch Anlagerung mehrerer Proteine bildet. Die für die Elongation benötigte Energie liefert die Abspaltung von Pyrophosphat von den NTPs. Dadurch bewegt sich der Elongationskomplex am DNA-Doppelstrang entlang, bis der Punkt der Termination erreicht ist. Bereits davor beginnt gleichzeitig das Spleißen der noch mit Introns durchsetzten mRNA.[1][2]
Der Elongationskomplex ist Ansatzpunkt für die Regulation der Elongation, und sie kann sowohl gebremst, als auch beschleunigt werden. Eine automatische Pause wird außerdem jedes Mal dann eingelegt, wenn sich der Komplex (nach Durchlaufen einer Histonschleife) wieder in der Nähe des Promotors befindet. Diese regelmäßigen Pausen scheinen wichtig für die Koordination der Elongation zu sein.[3]
Die Elongation der Prokaryoten und Eukaryoten ähneln sich sehr. Ein wesentlicher Unterschied ist, dass bei eukaryotischen Zellen der instabile Initationskomplex nach ca. 60 Nukleotiden zerfällt. Dieser Vorgang wiederholt sich mehrmals bis sich ein RNA-Transkript gebildet wird an dem die Transkription weitergeführt werden kann.[4]
Initiation der Elongation
[Bearbeiten | Quelltext bearbeiten]Der Beginn des Elongationsprozesses wird markiert durch die Dissoziation der Capping-Enzyme von der Polymerase II. Bevor jedoch die Hauptreaktion unter der vollständigen Kontrolle des Elongationskomplexes steht, muss dieser zusammengebaut werden. Zunächst wird die A-Untereinheit (RPB1) der Polymerase an den Serinen 2 und 5 dephosphoryliert; durch diese von der CTD-Phosphatase katalysierte Reaktion ist die Pol-II in der Lage, die Proteinkomplexe DSIF und NELF zu binden. Zusammen mit dem Kinasekomplex P-TEFb ist der frühe Elongationskomplex vollständig, wobei DSIF:NELF eine Pause in der Elongation bewirken kann und P-TEFb dem durch Phosphorylierung des Serin-2 entgegenspielt.
Der vollständige Elongationskomplex besteht aus dem frühen Elongationskomplex und dem TFIIS-Protein, dem FACT-Komplex, dem Elongationsfaktor ELL, dem Elongin-Komplex und dem Transkriptionsfaktor IIH.[5]
Hauptreaktion
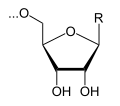
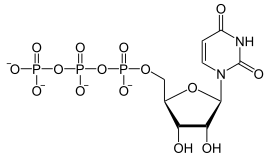
[Bearbeiten | Quelltext bearbeiten]Die zentrale Reaktion während der Elongation und auch bereits während der Initiation ist die Verlängerung der mRNA nach dem Vorbild des gebundenen DNA-Einzelstrangs, hier am Beispiel eines U:
UTP wird mit RNA verestert, es entsteht eine verlängerte RNA.
Weiterführende Literatur
[Bearbeiten | Quelltext bearbeiten]- Conaway, J./Conaway, R./reactome: RNA Polymerase II Transcription Elongation
Einzelnachweise
[Bearbeiten | Quelltext bearbeiten]- ↑ Gopinathrao/reactome: Formation of the Early Elongation Complex
- ↑ Gopinathrao/reactome: Formation of RNA Pol II elongation complex
- ↑ UniProt O00267
- ↑ P.C. Heinrich, M. Müller und L. Graeve: Löffler/Petrides Biochemie und Pathobiochemie. Hrsg.: Springer Verlag. 9. Auflage. Heidelberg 2014, ISBN 978-3-642-17972-3, S. 575.
- ↑ reactome: Recruitment of elongation factors to form elongation complex