Translation (Biologie)
Als Translation wird in der Biologie die Synthese von Proteinen in den Zellen lebender Organismen bezeichnet, die nach Vorgabe genetischer Information an den Ribosomen abläuft (siehe auch Proteinbiosynthese).
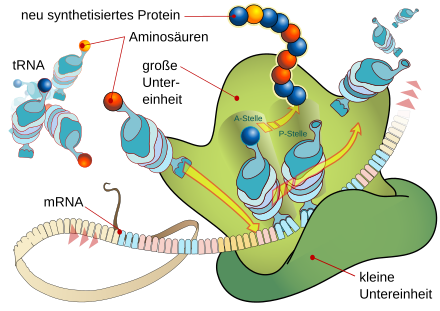
Das aus zwei Untereinheiten zusammengesetzte Ribosom synthetisiert ein Protein, indem dessen Peptidkette aus bestimmten Aminosäuren (bunte Kugeln) schrittweise aufgebaut wird. Das jeweils von einer spezifischen tRNA herangetragene Aminosäuremolekül wird nur dann angefügt, wenn das Anticodon dieser tRNA basenpaarend zum nächsten Codon des mRNA-Strangs passt. Auf diese Weise wird je an der A-Stelle die Aminosäure bestimmt, welche an der P-Stelle an das Peptid bindet. Derart kann die in der Basen-Sequenz der mRNA enthaltene Information in die codierte Formation der Aminosäure-Sequenz eines Proteins übersetzt werden.
Die Translation ist ein wesentlicher Teilprozess der Genexpression im Anschluss an die Transkription, bei der die Information eines DNA-Abschnitts auf einzelne RNA-Stränge überschrieben wurde. Nach der vorgegebenen Information findet dann an den Ribosomen im Cytoplasma einer Zelle die Translation statt. Dabei wird die Basensequenz eines mRNA-Moleküls in die codierte Aminosäuresequenz eines Polypeptids übersetzt und so ein Protein gebildet.
Allgemeiner Ablauf
[Bearbeiten | Quelltext bearbeiten]
An das aus drei Nukleinbasen bestehende Basentriplett eines Codons des mRNA-Stranges bindet basenpaarend das Anticodon auf der Anticodonschleife einer tRNA, die am Ende ihres Akzeptorarms beladen ist mit einer bestimmten Aminosäure. Diese steht damit bereit für den nächsten Schritt der ribosomalen Synthese, und wird per Peptidbindung der Aminosäurenkette angeknüpft.
Im Genom eines jeden Organismus sind Abschnitte zu finden, die als Gene nicht nur Informationen für den Bau von RNA enthalten, sondern darüber hinaus für den Aufbau von Proteinen. Die nach der Basenfolge eines solchen Abschnitts der DNA gebildete und gegebenenfalls prozessierte mRNA („m“ steht für englisch messenger ‚Bote‘) enthält in der Abfolge ihrer Basen, der Basensequenz, jeweils ausgewählte Informationen für die Biosynthese bestimmter Proteine.
Diese genetische Information wird im Verlauf der Translation als Anweisung genutzt, um das entsprechende Protein zu synthetisieren, indem nach dem genetischen Code Abschnitte der Basensequenz in die Aminosäuresequenz eines Peptids übersetzt werden. Dabei stellen je drei aufeinanderfolgende Nukleotide der mRNA ein Codon dar und codieren so als Basentriplett für eine bestimmte Aminosäure. Aus den codierten Aminosäuren wird am Ribosom in der durch die Nukleotidsequenz vorgegebenen Reihenfolge sequentiell die Polypeptidkette eines Proteins aufgebaut, mit der festgelegten Aminosäurensequenz. Die Information der mRNA wird hierbei in 5'→3'-Richtung abgelesen, also der gleichen Richtung, in der auch die RNA (durch RNA-Polymerase) transkribiert wurde.
Für den Translationsprozess sind als Aminosäuren-„Transporter“ verschiedene tRNA-Moleküle notwendig („t“ steht für englisch transfer ‚Übertragung‘). Diese können jeweils mit einer ihrer Schleifen, der Anticodonschleife, über ihr Anticodon komplementär basenpaarend an ein Codon auf der mRNA binden und sind an ihrem anderen Ende, dem Akzeptorarm, durch die unterschiedlichen Aminoacyl-tRNA-Synthetasen mit der zum Codon passenden Aminosäure beladen.
Bei der Translation legt sich das Ribosom an den mRNA-Strang und bringt diesen mit einer beladenen tRNA so zusammen, dass sich an ein Basentriplett eines Codons auf der mRNA nun als passendes Gegenstück das Basentriplett eines Anticodons der tRNA anlagern kann. Der eigentliche Translationsvorgang beginnt an jener Stelle der mRNA, wo die Basensequenz (z. B. → …, Adenin, Uracil, Guanin, …) das Startcodon darstellt (meist AUG). Eine zweite, zum folgenden Codon passende tRNA, die ebenfalls eine Aminosäure trägt, setzt sich neben der ersten tRNA an die mRNA. Die beiden nebeneinander positionierten Aminosäuren werden sodann durch die enzymatische Aktivität (Ribozym) der großen Untereinheit mit einer Peptidbindung verknüpft, und die erste tRNA verlässt ohne Aminosäure unbeladen das Ribosom. Nun lagert sich an die mRNA eine dritte, zum nächsten Codon passende beladene tRNA. Deren Aminosäure wird an die bereits bestehende Aminosäurekette geknüpft und verlängert sie so um ein weiteres Glied. Dieser Prozess setzt sich vom N- zum C-Terminus fort, so dass sich eine immer länger werdende Kette aus Aminosäuren bildet. Das Ribosom, das diesen Prozess katalysiert, wandert dabei jeweils schrittweise um ein Triplett bzw. Codon auf der mRNA weiter. Beendet wird die Translation, wenn sich in diesem Leseraster auf der mRNA ein Basentriplett findet, das ein Stopcodon darstellt (z. B. UGA). An dieses kann üblicherweise keine der vorliegenden tRNA-Molekülarten binden. Der Bereich auf einer mRNA zwischen dem Start- und dem zugeordneten Stop-Codon wird auch als offener Leserahmen (open reading frame) bezeichnet.
Mit dem Translationsende löst sich das als Verkettung von Aminosäuren synthetisierte Peptid vom Ribosom und die naszierende Polypeptidkette faltet sich im Medium zum nativen Protein, meistens so, dass eine komplexe räumliche Struktur entsteht (Sekundärstruktur und Tertiärstruktur). Eventuell verbindet es sich noch mit anderen Proteinen zu übergeordneten Quartärstrukturen.
Eine mRNA wird in der Regel mehrfach abgelesen, bis sie durch die Aktivität von Nucleasen in ihre Bausteine, die Ribonucleotide, zerlegt wird. Bei Eukaryoten ist die Haltbarkeit durch posttranskriptionelle Modifikationen im Kern erhöht.
Biochemischer Ablauf
[Bearbeiten | Quelltext bearbeiten]Obwohl es 61 Codons für die 20 kanonischen proteinogenen Aminosäuren gibt, werden im Zytoplasma einer Zelle nicht ebenso viele verschiedene Arten von tRNA gebraucht. Tatsächlich genügen in Bakterien schon 31 verschiedene Anticodons als Mittler zwischen den 20 Aminosäuren und den 61 Codons.[1] Die beim Menschen vorkommenden etwa 600 tRNA-Gene stellen nur 48 verschiedene Anticodons dar.[2] Denn manche tRNAs können mehrere verschiedene Codons für die gleiche Aminosäure erkennen. Das ist beispielsweise der Fall, wenn schon die beiden ersten Basen eines Basentripletts eine bestimmte Aminosäure festlegen, und die dritte so keine Rolle mehr spielt. Das Anticodon der mit der entsprechenden Aminosäure beladenen tRNA erkennt hier vorrangig die ersten beiden Positionen des Tripletts auf der mRNA mit der üblichen komplementären Basenpaarung – die dritte Paarung kann wackelig sein (siehe auch Wobble-Hypothese) – und somit verschiedene ähnliche Codons. Hingegen erkennt beispielsweise die mit Tryptophan beladbare tRNA (tRNATrp) normalerweise nur ein bestimmtes Codon (UGG).
Alle reifen tRNA-Moleküle bestehen aus einem RNA-Strang mit etwas weniger als 100 Nukleotiden, bilden in ihrer Sekundärstruktur infolge intramolekularer Paarungen von komplementären Nukleotidsequenzen mit Schleifen eine kleeblattähnliche Form und falten dreidimensional in eine hakenähnliche Tertiärstruktur. Im sogenannten Akzeptorarm sind das 5'- und das 3'-Ende vereint. Hier bindet dann die entsprechende Aminosäure am 3'-Ende, über ein posttranskriptional angefügtes CCA-Triplett. Die Anticodonschleife liegt in der Sekundärstruktur dem Akzeptorstamm gegenüber, auch in der Tertiärstruktur hat sie den größten Abstand. Drei zentrale Basen dieser Schleife im Anticodonarm bilden das Anticodon – meist in Position Nummer 36, 35 und 34, wobei letztere dann mit der 3. Base des Codons paart. Die D-Schleife enthält das ungewöhnliche Dihydrouridin (D), die T-Schleife neben Thymidin (T) typischerweise Pseudouridin (Ψ) und Cytosin (C). Die V-Schleife ist variabel, also bei einzelnen tRNA-Arten unterschiedlich zusammengesetzt.
Für die Beladung einer tRNA mit ihrer Aminosäure ist jeweils eine besondere Aminoacyl-tRNA-Synthetase zuständig. Meist gibt es für jede Aminosäure eine spezifische Synthetase.
Ribosomen und Protein-Synthese
[Bearbeiten | Quelltext bearbeiten]An den Ribosomen erfolgt die Paarung einer Aminoacyl-tRNA über ihr Anticodon mit dem Codon der mRNA und durch Peptidbindung der herangetragenen Aminosäuren die Synthese der Polypeptidkette von Proteinen. Diese ribosomale Peptidsynthese durch Translation der genetisch codierten Information ist der Hauptschritt der Proteinbiosynthese.
Ribosomen bestehen aus zwei Untereinheiten, die jeweils wiederum aus RNA (ribosomale RNA) und Polypeptiden (ribosomale Proteine) aufgebaut sind. Zunächst sind die beiden Untereinheiten getrennt. Bei der Translation vereinigen sie sich und bilden zwei Bereiche aus, an denen die tRNAs anlagern können: die Aminoacyl-Stelle (A-Stelle) für die tRNA mit der nächsten anzufügenden Aminosäure, die Peptidyl-Stelle (P-Stelle) für die tRNA der an die wachsende Peptidkette angefügten Aminosäure. Die entladenen tRNA-Moleküle verlassen das Ribosom dann über eine andere Region, die Exit-Stelle (E-Stelle).
Initiation der Translation bei Prokaryoten
[Bearbeiten | Quelltext bearbeiten]
Für die Initiation als den Start des Prozesses wie den Anfang der Kette benötigt die Zelle neben den beiden ribosomalen Untereinheiten und der mRNA noch eine spezielle tRNA. Diese Initiator-tRNA bindet an das Startcodon AUG und ist bei Bakterien eine tRNAifMet, die Formylmethionin (fMet) überträgt, statt des Methionins der bei Archaeen (und Eukaryoten) üblichen tRNAiMet. Darüber hinaus spielen bei Prokaryoten drei Initiationsfaktoren (IF 1, IF 2, IF 3) eine Rolle.
Die kleine Untereinheit (30S) bildet zu Beginn einen Komplex mit den Initiationsfaktoren 1 und 3.[3] Die Aufgabe des IF1 ist die Dissoziation der (in einem dynamischen Gleichgewicht liegenden) Nichtinitiator-tRNA. Der IF3 verhindert zusammen mit dem IF1 eine frühzeitige Bindung der beiden ribosomalen Untereinheiten.
Der IF2, ein G-Protein, bindet GTP, durchläuft eine Konformationsänderung und kann so die Initiator-tRNA binden. Dieser Komplex aus IF2-GTP und (beladener) fMet-tRNAifMet hat nun die Möglichkeit, sowohl an mRNA wie an 30S-Einheit zu binden.
Die kleine Untereinheit vermag durch eine Interaktion der anti-Shine-Dalgarno-Sequenz ihrer 16S-rRNA (ribosomale RNA als Teil der 30S-Einheit) mit der Shine-Dalgarno-Sequenz auf der mRNA die geeignete Bindungsstelle zu erkennen. Diese nicht-codierende Sequenz liegt wenige Nukleotide (9 nt upstream) vor einem Basentriplett, das ein AUG darstellt, und ermöglicht somit die Erkennung des Startcodons durch die Initiator-tRNA. Der Abschluss der Initiation wird durch GTP-Hydrolyse am IF2 eingeleitet. Es kommt zum Entlassen der Initiationsfaktoren und erst dann zur Bindung der 50S-Untereinheit, wodurch der 70S-Initiator-Komplex entsteht. Die fMet-tRNAifMet befindet sich zu Beginn der Translation bereits in der P-Stelle der 50S-Untereinheit. Die beiden anderen Stellen, A und E, sind leer.
Elongation der Polypeptidkette
[Bearbeiten | Quelltext bearbeiten]
Die Elongation ist der Prozess der Verlängerung der Aminosäurenkette; sie findet am Erkennungs- und am Bindungsort des Ribosoms statt. Ein einzelner Elongationsschritt enthält drei Schritte: Bindung der beladenen tRNA, Ausbildung der Peptidbindung und Vorbereitung auf den nächsten Elongationsschritt. Dies wiederholt sich so lange, bis ein terminierendes Codon erreicht ist.
Termination bei Prokaryoten
[Bearbeiten | Quelltext bearbeiten]
Das Ende der Translation ist erreicht, wenn eines der Stopp-Tripletts UAG, UAA oder UGA in der A-Stelle des Ribosoms auftaucht. Da es in der Zelle keine passende tRNA für diese Codons gibt, hält die Translation an.
Terminationsfaktoren (release factors) binden dann an das Basentriplett des Stopcodons: RF1 an UAG und UAA oder RF2 an UAA und UGA. Das veranlasst die Spaltung der Bindung zwischen der letzten Aminosäure und der letzten tRNA im Ribosom. Während der Translation kann der Ester nicht durch Hydrolyse aufgebrochen werden, da der Bereich der Peptidyl-Transferase vollkommen wasserfrei ist. So wird eine spontane Hydrolyse während der Elongation verhindert. Der RF bringt aber, vermittelt durch die Aminosäure-Sequenz Glycin-Glycin-Glutamin genau ein Molekül Wasser in das Peptidyl-Transferase-Zentrum. Dieses kann dann mit Hilfe katalytischer Aktivität des Ribosoms die Esterbindung spalten. Diese Sequenz befindet sich auch im eukaryotischen RF. Die Dissoziation von RF1/RF2 vom Ribosomen wird durch den Terminationsfaktor RF3 katalysiert.
Nun fallen das Protein und die mRNA vom Ribosom ab, das wieder in seine beiden Untereinheiten zerfällt. Der Initiationsfaktor IF3 erhält den dissoziierten Zustand aufrecht. Somit kann der Kreislauf von Neuem beginnen.
Translation in Eukaryoten
[Bearbeiten | Quelltext bearbeiten]Initiation
[Bearbeiten | Quelltext bearbeiten]
Die Translation bei Eukaryoten unterscheidet sich von der prokaryotischen Translation insbesondere in der Initiation, an der eine Reihe spezieller eukaryotischer Initiationsfaktoren (eIF) beteiligt sind. Die Initiator-tRNA ist hier eine tRNAiMet, die Methionin trägt und nicht formyliert ist. Eine Shine-Dalgarno-Sequenz ist auf der eukaryotischen mRNA nicht zu finden. Es wird gewöhnlich das vom 5'-Ende her erste Basentriplett AUG der mRNA als Startcodon gewählt. Die Bindung der 40S-Untereinheit erfolgt zumeist an der 5'-Cap-Struktur der mRNA. Nach Bildung des Präinitiatinskomplexes aus kleiner Untereinheit und Initiator-tRNA mit eIF-2 und weiteren Faktoren wird die mRNA in 3'-Richtung nach einem AUG abgesucht. Wenn diese Suche erfolgreich war, lagert sich die Initiator-Met-tRNA an das Basentriplett der mRNA. Der Translationsvorgang beginnt aber erst, wenn auch die größere Untereinheit (60S) des Ribosoms gebunden wurde (siehe nebenstehende Abbildung).
Bilden eukaryotische mRNA während der Prozessierung oder ihres Transports aus dem Kern komplexe Sekundärstrukturen, können diese durch Helikasen wieder aufgebrochen werden.
Termination
[Bearbeiten | Quelltext bearbeiten]Das Ende der Translation wird üblicherweise durch das Basentriplett eines Stopcodons markiert. Auch beim Menschen wurden aber inzwischen einige Gene entdeckt, bei denen durch das Überlesen eines Stopsignals auf der mRNA (englisch translational readthrough genannt) verlängerte Proteine und damit neue Isoformen entstehen. Dazu kann es kommen, wenn beispielsweise das Codon UGA anders interpretiert und in eine Aminosäure übersetzt wird, etwa Tryptophan.[4] Hiervon abzugrenzen sind jene Sonderfälle der Recodierung, bei denen durch Einsatz spezifischer tRNA-Moleküle der Einbau zusätzlicher proteinogener Aminosäuren wie Selenocystein und Pyrrolysin ermöglicht wird.
Regulation
[Bearbeiten | Quelltext bearbeiten]Jedes von der Zelle zum Überleben benötigte Protein ist in den Genen codiert. Die benötigte Menge allerdings ist dabei nicht direkt im Gen codiert und außerdem abhängig von Umgebungsbedingungen, Alter und Zellzyklus und vor allem von der Art der Zelle (Zelltyp). Der quantitativ weitaus wichtigste Angriffspunkt der Steuerung der Proteinherstellung (Proteinexpression) ist aber nicht die Translation, sondern die Transkription. Die Frage, ob ein bestimmtes Protein hergestellt wird, wird also nicht in erster Linie darüber entschieden, ob die mRNA, die dieses Protein kodiert, an der Translation teilnimmt, sondern darüber, ob die mRNA überhaupt hergestellt wird.
Dennoch ist die Regulation der Translation ein wichtiger Angriffspunkt der Genregulation. Dabei wird also gesteuert, wie viel bzw. welches Protein von einer bestimmten mRNA hergestellt werden. Zwei Beispiele:
- Regulation der Initiation: Durch Phosphorylierung kann der eukaryotische Initiationsfaktor eIF2 reguliert werden. Über den mTOR-Signalweg ist so die Regulation der Translation an das Zellwachstum bzw. den Zellzyklus und die Menge an verfügbaren Nährstoffen gekoppelt.
- Regulation der Termination: Durch funktionalen translationalen Readthrough können die peroxisomalen Isoformen der LDH in der Zelle hergestellt werden.
Weitere Stichworte zur Regulation der Translation sind 5'-positionierte kleine offene Leserahmen, codon optimality und der Startkodonkontext Kozak-Sequenz.
Beispiel einer Regulation der Translation ribosomaler Proteine
[Bearbeiten | Quelltext bearbeiten]Die quantitativ abgestimmte Expression ribosomaler Proteine stellt ein regulatorisch interessantes Problem auf Zellebene dar. Jedes Ribosom enthält rund 50 spezielle Proteine, die alle mit derselben Rate synthetisiert werden müssen. Des Weiteren sind die Syntheserate von Proteinen der Zelle und der Bedarf an Ribosomen eng mit dem Zellwachstum verbunden. Eine Veränderung der Wachstumsbedingungen führt daher rasch zu einem Anstieg oder Absinken der Syntheserate dieser ribosomalen Komponenten.
Die hierfür nötige Regulation der Genexpression ist auf Ebene der Transkription vereinfacht durch eine Organisation in verschiedene Operons, die jeweils Gene für bis zu 11 ribosomale Proteine enthalten. Die wesentliche Kontrolle der Syntheserate geschieht jedoch auf der Ebene der Translation wie durch das folgende Experiment nachgewiesen wurde:
Durch gentechnische Veränderung wurden zusätzliche Kopien eines solchen Operons in das Erbgut einer Zelle eingebracht; dementsprechend steigt die Menge der durch Transkription erzeugten mRNA an. Dennoch bleibt die Syntheserate des Proteins aber nahezu unverändert. Die Zelle kompensiert also regulatorisch die erhöhte mRNA-Menge. Dabei wirken ribosomale Proteine als Repressor ihrer eigenen Translation.
Bei jedem Operon kann dabei ein schon synthetisiertes ribosomales Protein an die mRNA des Operons binden. Diese Bindungsstelle liegt in der Nähe eines der ersten Gene des Operons. Dadurch werden Ribosome daran gehindert, an die mRNA zu binden und mit der Translation zu beginnen. Die Repression der Translation der ersten Gene verhindert also die Expression mindestens eines Teils der nachfolgenden Gene.
Dieser Mechanismus ist sehr empfindlich. Schon wenige nicht zur Bildung von Ribosomen verbrauchten Moleküle des Proteins L4 zum Beispiel verhindern sowohl die Synthese dieses Proteins als auch die der übrigen 10 ribosomalen Proteine im gleichen Operon. Dadurch wird also sichergestellt, dass die Proteine nicht in zu großen Mengen erzeugt werden und nahezu komplett zur Bildung von Ribosomen verbraucht werden können.
Wie ein Protein sowohl als ribosomale Komponente als auch als Regulator seiner eigenen Translation dienen kann, konnte durch Vergleich der Bindungsstellen des Proteins an der rRNA mit den Bindungsstellen mit seiner eigenen mRNA erforscht werden. Beide Bindungsstellen ähneln sich in ihrer Sequenz und ihrer Sekundärstruktur. Da die Bindung der ribosomalen Proteine an die rRNA stärker ist als die an die mRNA, wird die Translation nur unterdrückt, wenn der Bedarf an Proteinen für die Produktion von Ribosomen gedeckt ist.
Translokation in und durch Membranen
[Bearbeiten | Quelltext bearbeiten]Sowohl bei Prokaryoten als auch bei Eukaryoten findet die Proteinsynthese an den Ribosomen im Cytosol der Zelle statt. Von hier aus können Proteine in eine Membran oder durch sie hindurch transportiert werden. Diese Verlagerung an einen anderen Ort, auch Translokation genannt, kann schon bei der Synthese eines Proteins während der Translation eingeleitet werden, also cotranslational ablaufen, oder erst nach abgeschlossener Synthese, also posttranslational stattfinden.
Entscheidend für die Translokation, die bevorzugte Transportsart und den jeweiligen Bestimmungsort sind zumeist gewisse Abschnitte in der Aminosäurensequenz des gebildeten Proteins, die als Signalsequenzen von Signalerkennungspartikeln oder besonderen Proteinkomplexen (etwa des Sec-Systems) erkannt werden. Bei Prokaryoten kann ein neugebildetes Protein derart bestimmt werden für den Transport in die Zellmembran oder durch sie hindurch in den extraplasmatischen Raum, beispielsweise für den Aufbau einer Zellwand. Da Eukaryoten verschiedene Organellen als membranumhüllte Zellkompartimente besitzen, sind die möglichen Zielorte einer Translokation von Proteinen hier vielfältiger. Von dem Transport in den extrazellulären Raum oder in die Zytomembran zu unterscheiden sind die Transportwege in Zielkompartimente wie Endoplasmatisches Retikulum, Zellkern, Peroxisome und andere Vesikel sowie die in Mitochondrien, Chloroplasten oder andere Plastiden.
Cotranslationaler Proteintransport
[Bearbeiten | Quelltext bearbeiten]Bei diesem Vorgang wird das Ribosom noch während der Translation zunächst an die Membran des Endoplasmatischen Reticulums (ER) geführt, indem eine spezifische Signalsequenz am soeben gebildeten Anfang der Polypeptidkette erkannt wird, das spezifische Signalerkennungspartikel (SRP) durch Bindung an das Ribosom die Proteinsynthese verzögert, und dann an einen SRP-Rezeptor in der Membran des ER bindet. Das Ribosom kann dadurch mit einem tunnelbildenden (Sec61-)Komplex in der Membran interagieren, in dessen Tunnel das naszierende Polypeptid einfädelt. Nachdem sich das SRP gelöst hat, kann mit Fortsetzung der ribosomalen Synthese das neugebildete Protein dadurch auf die andere Seite der Membran gebracht und so transloziert werden. Hierbei wird zunächst eine Schleife des Proteins durch den Translokationskanal geschoben und danach die im Kanal fixierte Signalsequenz abgespalten.
Posttranslationaler Proteintransport
[Bearbeiten | Quelltext bearbeiten]Das in der Zelle vollständig zusammengebaute und durch ein Chaperon vor vorzeitiger Auffaltung geschützte Protein wird an seinen Bestimmungsort transportiert. Bei Bakterien wird durch einen eingebauten „Knick“ im Protein das Durchfädeln durch die Zellmembran erleichtert. Der eukaryotische posttranslationale Transport durch die ER-Membran konnte in Hefen gezeigt werden.
Literatur
[Bearbeiten | Quelltext bearbeiten]- Rolf Knippers: Molekulare Genetik. 9. komplett überarbeitete Auflage. Thieme, Stuttgart u. a. 2006, ISBN 3-13-477009-1.
Weblinks
[Bearbeiten | Quelltext bearbeiten]- Lukas Hensel – Animation zur Translation
- Universität Bern – Translation bei Prokaryonten und Eukaryonten
- Zentrale für Unterrichtsmedien im Internet – Translation
- IUBMB: Prokaryotic and eukaryotic translation factors
- MRC-LabMB: Movies and Overview Figures of the Ribosome
Einzelnachweise
[Bearbeiten | Quelltext bearbeiten]- ↑ B. Alberts, A. Johnson, J. Lewis et al.: Molecular Biology of the Cell. 4. Ausgabe. Garland Science, New York 2002, Kapitel: From RNA to Protein. Online auf dem NCBI-Bookshelf
- ↑ siehe in der Genomischen tRNA Datenbank (GtRNAdb) Einträge für Homo sapiens.
- ↑ James D. Watson u. a.: Molecular Biology of the Gene. 5. Auflage. Pearson/Cummings u. a., San Francisco CA 2004, ISBN 0-8053-4635-X, S. 435.
- ↑ F. Schueren und S. Thoms: Functional Translational Readthrough: A Systems Biology Perspective. In: PLOS Genetics. 12(8). Jahrgang, e1006196, August 2016, S. 12, doi:10.1371/journal.pgen.100619, PMID 27490485, PMC 4973966 (freier Volltext).