Strictosidin-Synthase
| Strictosidin-Synthase (Rauvolfia serpentina) | ||
|---|---|---|
| ||
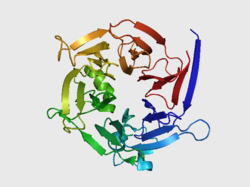
| Bändermodell nach PDB 2FP9 | ||
|
Vorhandene Strukturdaten: s. UniProt | ||
| Masse/Länge Primärstruktur | 322 Aminosäuren | |
| Sekundär- bis Quartärstruktur | Monomer | |
| Bezeichner | ||
| Externe IDs |
| |
| Enzymklassifikation | ||
| EC, Kategorie | 4.3.3.2, Lyase | |
| Reaktionsart | Pictet-Spengler Kondensation | |
| Substrat | Tryptamin und Secologanin | |
| Produkte | Strictosidin und H2O | |
Strictosidin-Synthase ist ein pflanzliches Enzym, das die Mannich-artige Kondensation von Tryptamin und Secologanin nach Strictosidin katalysiert. Dies ist ein wichtiger Schritt in der Biosynthese der Indolalkaloide. Das Enzym ist in den Vakuolen der Wurzeln lokalisiert.[1]
Der nächste Schritt im anabolischen Stoffwechselweg von Strictosidin ist die Deglykolysierung durch Strictosidin-Glucosidase und ergibt Cathenamin.[2]
Strictosidin-Synthase besitzt eine molekulare Masse von ungefähr 34 kDa und wurde erstmals 1979 aus Zellkulturen von in Catharanthus roseus isoliert und von Johannes Treimer und Meinhart Zenk an der Ruhr-Universität Bochum beschrieben.[3] Seither wurde das Enzym auch aus anderen Pflanzen der Familie der Hundsgiftgewächse isoliert und das Enzym der Arten Catharanthus roseus, Rauvolfia serpentina[4] und Ophiorrhiza pumila[5] wurde kloniert. Obwohl Strictosidin-Synthase eine hohe Substratspezifität[3] und eine hohe Substrataffinität aufweist (KM von 2,3 mM für Tryptamin und 3,4 mM für Secologanin[3]), toleriert es verschieden substituiertes Tryptophan und Secologanin ebenfalls als Substrate.[6]

Das Enzym gehört in die Klassen der Amin-Lyasen (EC 4.3), welche Kohlenstoff-Stickstoff Bindungen synthetisieren. Das Enzym spielt in der Synthese der Indolalkaloide eine wichtige Rolle, da Strictosidin der Vorläufer von etwa 3000[7] unterschiedlichen Indol-Terpen-Alkaloiden ist, darunter auch die beiden wichtigen Krebsmedikamente Vincristin und Vinblastin, das Malariamedikament Chinin, der Blutdrucksenker Reserpin und das Antiarrhythmikum Ajmalin.[8]
2006 gelang es Forschern der Johannes Gutenberg-Universität in Mainz, die Struktur des Enzymes aus Rauvolfia serpentina aufzuklären. Dabei lösten die Forscher sowohl die Struktur des nativen[9] Pflanzenproteins als auch die Struktur eines mutierten[10] Proteins.[8] Strukturell handelt es sich bei dem Enzym um einen Beta-Propeller mit einer sechsfachen pseudo-Symmetrieachse. Die sechs Untereinheiten bestehen aus vier antiparallelen Beta-Faltblättern. Das Protein kristallisiert als Dimer, ist jedoch als Monomer aktiv.[8]
Einzelnachweise
[Bearbeiten | Quelltext bearbeiten]- ↑ UniProt P68175
- ↑ McCoy E, Galan MC, O’Connor SE: Substrate specificity of strictosidine synthase. In: Bioorg Med Chem Lett. 16(9). Jahrgang, 2006, S. 2475–8, PMID 16481164.
- ↑ a b c Treimer JF, Zenk MH: Purification and properties of strictosidine synthase, the key enzyme in indole alkaloid formation. In: Eur J Biochem. 101. Jahrgang, 1979, S. 225–33, doi:10.1111/j.1432-1033.1979.tb04235.x, PMID 510306.
- ↑ Kutchan TM, Hampp N, Lottspeich F, Beyreuther K, Zenk MH: The cDNA clone for strictosidine synthase from Rauvolfia serpentina. DNA sequence determination and expression in Escherichia coli. In: FEBS Lett. 237(1-2). Jahrgang, 1988, S. 40–44, PMID 3049153.
- ↑ Yamazaki Y, Sudo H, Yamazaki M, Aimi N, Saito K: Camptothecin biosynthetic genes in hairy roots of Ophiorrhiza pumila: cloning, characterization and differential expression in tissues and by stress compounds. In: Plant Cell Physiol. 44(4). Jahrgang, 2003, S. 395–403, PMID 12721380.
- ↑ O’Connor SE, Maresh JJ: Chemistry and biology of monoterpene indole alkaloid biosynthesis. In: Nat Prod Rep. 4. Jahrgang, 2006, S. 532-47, PMID 16874388.
- ↑ van Der Heijden R, Jacobs DI, Snoeijer W, Hallard D, Verpoorte R: The Catharanthus alkaloids: pharmacognosy and biotechnology. In: Curr. Med. Chem. 11(5). Jahrgang, 2004, S. 607–28, PMID 15032608.
- ↑ a b c Ma X, Panjikar S, Koepke J, Loris E, Stöckigt J: The structure of Rauvolfia serpentina strictosidine synthase is a novel six-bladed beta-propeller fold in plant proteins. In: Plant Cell. 4. Jahrgang, 2006, S. 907-20, PMID 16531499.
- ↑ PDB 2FP8, PDB 2FP9 und PDB 2FPC
- ↑ PDB 2FPB