Affinitätschromatographie
Die Affinitätschromatographie (manchmal ungebräuchliches Synonym: Proteinaffinitätschromatographie) ist ein chromatographisches Trennverfahren zur Isolation eines Analyten aus einer Lösung verschiedener Stoffe. Voraussetzung ist, dass ein geeigneter Ligand (Bindungspartner) zu dem interessierenden Analyten (Protein) zur Verfügung steht. Sie ist eine der leistungsfähigsten Trennmethoden. Die verwendeten Säulen sind jedoch relativ kostspielig, so dass das Verfahren nur in besonderen Fällen beziehungsweise im kleinen Maßstab (Labormaßstab) zum Einsatz kommt.
Prinzip
[Bearbeiten | Quelltext bearbeiten]
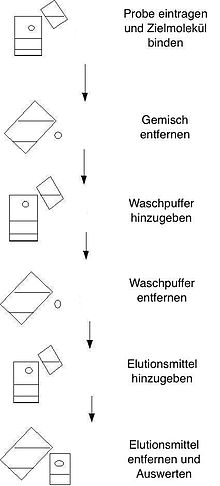
Die Trennung erfolgt meist in Säulen, kann aber auch im Batchverfahren vorgenommen werden. Der Reinigungseffekt dieser Methode basiert entweder auf der spezifischen Erkennung eines Proteins durch einen Antikörper oder bei Enzymen auf Ausnutzung der spezifischen Affinität eines Enzyms zu einem Inhibitor, Substrat oder Cofaktor.
Die stationäre Phase, oft ein Gel, z. B. aus Dextranen oder quervernetzter Agarose (Handelsname Sepharose), wird mit einem geeigneten Liganden (z. B. Antikörper bzw. Immunglobulinen) gekoppelt, der spezifisch den zu reinigenden Analyten bindet. Hierbei ist in der Praxis darauf zu achten, dass die Affinität gegen den Analyten nicht zu hoch ist, da die Elution dadurch erschwert wird. Umgekehrt kann zur präparativen Reinigung von Antikörpern auch eine stationäre Phase mit einem Protein (meist Protein A, G oder L) verwendet werden, das bestimmte Immunglobulinklassen bindet.
Zur Bindung eines Liganden auf dem Trägermaterial wird dieses vorher in eine aktivierte Form überführt. Beim Beispiel der Agarose kommt die Bromcyan-Aktivierungsmethode in Betracht, bei der reaktive Imidocarbonat-Gruppen erzeugt werden, die mit Aminogruppen des Analyten unter Ausbildung einer kovalenten Bindung reagieren. Nach der Fixierung eines niedermolekularen Liganden auf der Matrix (z. B. aktivierte Agarose) kann es zu sterischer Hinderung kommen, wenn der Analyt höhermolekular ist. In diesem Fall kann der Ligand über ein Brückenglied (Spacer) an die Matrix gekoppelt werden. Als Spacer werden normalerweise kurze Kohlenwasserstoffketten verwendet, die dann gewissermaßen aus der Matrixoberfläche herausragen.
Für die Bindung von Ligand und Zielprotein ist die Assoziationskonstante, die sich aus der Gleichgewichtskonstanten ableitet, von Bedeutung. Je größer diese ist, desto günstiger verläuft die affinitätschromatographische Trennung. Der Wert sollte mindestens bei KAss = 104 mol−1 liegen.
Anwendung
[Bearbeiten | Quelltext bearbeiten]Primär benutzt man die Affinitätschromatographie zum Reinigen und Konzentrieren einer Substanz aus einer Mischung in eine Pufferlösung oder zum Reduzieren einer Substanzmenge in einer Mischung. Auch um festzustellen, welche biologischen Verbindungen an eine bestimmte Substanz binden, oder zum Reinigen und Konzentrieren einer Enzymlösung, wird die Affinitätschromatographie häufig verwendet.
Durchführung
[Bearbeiten | Quelltext bearbeiten]Zur Ausführung affinitätschromatographischer Arbeiten werden heute meist biokompatible Anlagen der HPLC eingesetzt, die jedoch hier nur mit geringeren Drücken von bis zu 150 bar betrieben werden. Sogenannte FPLC-Anlagen (für englisch fast protein liquid chromatography) arbeiten sogar mit deutlich geringeren Drücken von oft unter 10 bar. Das zu trennende Gemisch wird in der Regel über Probenschleifen oder über Probenpumpen auf die Säule aufgegeben und der Analyt durch die Liganden gebunden. Alle anderen Stoffe eluieren schnell, da sie mit dem Liganden nicht stark wechselwirken. Nach einem Waschschritt, um unspezifisch gebundene Verunreinigungen zu entfernen, wird der am Liganden gebundene Analyt durch Veränderung der Bedingungen (z. B.: anderer Puffer) ebenfalls eluiert. Als Elutionsmittel wird oft ein saurer Puffer oder ein Lösungsmittel/Wasser-Gemisch verwendet. Alternativ können auch kompetitiv zum Zielprotein agierende Substanzen oder ein Überschuss an freien Liganden zugesetzt werden. Das Eluat enthält den gereinigten und angereicherten Analyten. Das Batchverfahren ähnelt sehr stark der Vorgehensweise des zuvor erläuterten Säulenbetriebs. Wichtiger Unterschied ist jedoch die Nutzung von Zentrifugation und weiterer Trennmethoden, welche druckunabhängig sind. Je nach Bedarf können auch beide Verfahren untereinander kombiniert werden, in dem der Säulenbetrieb nach dem Batchverfahren folgt.
Neuere Entwicklungen basieren auf der Verwendung von mehreren hintereinander geschalteten Säulen. Hierbei können die teuren Liganden bis zur maximalen Belegung genutzt werden. In Ein-Säulen-Prozessen ist dies nicht möglich, da ein Teil des Produktes nicht mehr gebunden wird und somit eluiert und verloren ginge. In Mehrsäulenprozessen wird dieses Eluat in einer weiteren Säule aufgefangen. Durch abwechselndes Beladen und Eluieren der Säulen wird ein periodischer Prozess erreicht, der oft als kontinuierliche Chromatographie bezeichnet wird. Dies kann bereits mit zwei Säulen effizient umgesetzt werden; weitere Säulen verringern die Effizienz wiederum.[1]
Spezielle Verfahren
[Bearbeiten | Quelltext bearbeiten]Die Affinitätschromatographie findet spezielle Verwendungen in der Aufreinigung von Nukleinsäuren, Proteinen oder Blut. Beispiele hierfür sind u. a. der His-Tag oder der Strep-tag[2] zur Aufreinigung rekombinanter Proteine:
| 1. Immunaffinität: Reinigung von Antikörpern aus Blutseren |
| 2. Immobilisierte-Metallionen-Affinitätschromatographie (IMAC): Beruht auf der kovalenten Bindung von Aminosäuren, insbesondere Histidin, an Metallen |
| 3. Rekombinante Proteine |
| 4. Lektine: Proteine, welche Kohlenhydrate binden, um Substanzen in der Probe zu trennen.[3] |
Beispiele
[Bearbeiten | Quelltext bearbeiten]Beispiele für Liganden zur Proteinreinigung:
| Ligand | Zielprotein |
|---|---|
| Antigen, Protein A, Protein G oder Protein L |
Antikörper |
| Substrat, Kofaktor | Enzym |
| Ligand | Rezeptor |
| Lectin | Glykoprotein |
| Nukleinsäure | Nukleinsäure bindendes Protein |
| Streptavidin, Avidin | Biotin, Protein mit Streptavidin-peptid |
| Metallionenchelat wie Ni2+-NTA |
Protein mit Poly-histidin-peptid |
Literatur
[Bearbeiten | Quelltext bearbeiten]- Heinz Bende: Affinitäts-Chromatographie. In: Chemie in unserer Zeit. Band 8, Nr. 1, 1974, S. 17–25, doi:10.1002/ciuz.19740080104.
- Jerker Porath et al. (1975): Metal chelate affinity chromatography, a new approach to protein fractionation. In: Nature. Bd. 258, Nr. 5536, S. 598–599. PMID 1678
Siehe auch
[Bearbeiten | Quelltext bearbeiten]Einzelnachweise
[Bearbeiten | Quelltext bearbeiten]- ↑ Daniel Baur, Monica Angarita, Thomas Müller-Späth, Fabian Steinebach, Massimo Morbidelli: Comparison of batch and continuous multi-column protein A capture processes by optimal design. In: Biotechnology Journal. Band 11, Nr. 7, 1. Juli 2016, S. 920–931, doi:10.1002/biot.201500481.
- ↑ Thomas GM Schmidt, Arne Skerra: The Strep-tag system for one-step purification and high-affinity detection or capturing of proteins. In: Nature Protocols. Band 2, Nr. 6, S. 1528–1535, doi:10.1038/nprot.2007.209.
- ↑ Immobilized Lectin. In: gelifesciences.com. GE Healthcare Lifesciences, archiviert vom (nicht mehr online verfügbar) am 20. Januar 2015; abgerufen am 1. Juli 2024.
Weblinks
[Bearbeiten | Quelltext bearbeiten]- Amersham: Affinity chromatography – Principles and Methods (PDF; 2,8 MB)