Benutzer:Physiosoziologicus/Epigenetik

Die Epigenetik ist ein Spezialgebiet der Biologie. Sie befasst sich mit Zelleigenschaften (dem Phänotyp), die zwar bei der Zellteilung weitergegeben werden, aber nicht im genetischen Code (dem Genotyp) festgelegt sind. Hierbei erfolgen insbesondere Veränderungen an den Chromosomen, wodurch Abschnitte oder ganze Chromosomen in ihrer Aktivität beeinflusst werden. Man spricht infolgedessen auch von epigenetischer Veränderung bzw. epigenetischer Prägung.[1] Epigenetische Veränderungen können auf natürlichen Prozessen beruhen. Daneben sind sie aber auch Thema und Ziel medizinischer und biotechnologischer Forschung, unter anderem in der Stammzellforschung.
Einführung
[Bearbeiten | Quelltext bearbeiten]Den wesentlichen Hintergrund der natürlichen Epigenetik bilden Prozesse der Zelldifferenzierung. Hierbei entstehen bei allen Vielzellern aus einer Zelle sehr unterschiedliche, auf einzelne Aufgaben spezialisierte Körperzellen, obwohl der genetische Code, die DNA, nach wie vor die Informationen für alle Körperzellen enthält. Die Epigenetik beschäftigt sich mit den Prozessen und biochemischen Strukturen, die diese Auswahl in der Verarbeitung von Informationen bewirken.
Im Rahmen der induviduellen Entwicklung teilt sich die Eizelle nach der Befruchtung und durchläuft verschiedene Stadien. Bis zum Stadium der Morula sind dabei alle Tochterzellen gleich. Danach finden sich Zellen mit einem unterschiedlichen 'inneren Programm' und jeweils eigener Funktion. Ist schließlich der Körper fertig entwickelt, sind die meisten Körperzellen für ihre Funktion fest programmiert. Während dieser Entwicklung bleibt die Information Erbgutes (bis auf zufällige genetische Mutationen) unverändert. Die funktionelle Festlegung erfolgt durch biochemische Ergänzungen an einzelnen Basen der Sequenz und der die DNA verpackenden Histone. Solche Veränderungen führen dazu, dass bestimmte Bereiche des Erbgutes ruhig gestellt, andere dafür leichter in RNA und Protein überführt werden können. Diese Modifizierungen finden sich nur in Körperzellen, nicht aber z. B. in Eizellen oder Spermien. Von Bedeutung für epigenetische Phänomene ist vor allem die Methylierung von Cytidin-Basen (sog. CpG). Außerdem spielt die Seitenketten-Methylierung und -Acetylierung von Histonen eine wichtige Rolle.
Begriffsbezeichnung
[Bearbeiten | Quelltext bearbeiten]Die griechische Vorsilbe epi in „Epigenetik“ hat mehrere Bedeutungen, wie „nach“, „hinterher“, „um herum“ oder „zusätzlich“. Epigenetisch sind danach alle Prozesse in einer Zelle, die als „zusätzlich zu“ den Inhalten und Vorgängen der Genetik gelten. Conrad Hal Waddington soll den Begriff „Epigenetik“ erstmals benutzt haben. Im Jahr 1942 definierte er Epigenetik als „the branch of biology which studies the causal interactions between genes and their products which bring the phenotype into being“.
Epigenese
[Bearbeiten | Quelltext bearbeiten]Mit dem Ausdruck Epigenese werden Prozesse der embryonalen Morphogenese, also der Bildung und Reifung von Organen beschrieben. Epigenese spricht also eine Entwicklung auf makroskopischer Ebene an. Auf mikroskopischer Ebene beruht allerdings auch die Epigenese auf epigenetischen Prozessen bei der Zellteilung der Vorläuferzellen, der Zelldifferenzierung.
Epigenetik im Vergleich zur Genetik
[Bearbeiten | Quelltext bearbeiten]
Man kann das Thema „Epigenetik“ weiterhin illustrieren, indem man sich die zentralen Vorgänge der Vererbung vor Augen führt:
- Vor einer Zellteilung wird die Erbsubstanz verdoppelt. Jeweils die Hälfte dieses verdoppelten Genoms wird dann auf eine der beiden Tochterzellen übertragen. Bei der sexuellen Vermehrung des Menschen, der Fortpflanzung werden von der Eizelle die Hälfte des mütterlichen Erbguts und vom Spermium die Hälfte des väterlichen Erbguts miteinander vereint.
- Das Standardmodell der Molekulargenetik beschreibt die Erbsubstanz als Doppelhelix aus zwei DNS-Strängen. Die genetische Information selbst ist danach durch die Reihenfolge der vier Basen Adenin (A), Cytidin (C), Guanin (G) und Thymin (T) bestimmt, die die Stränge wie Sprossen einer Strickleiter verbinden. Sie können als eine Art Buchstaben verstanden werden, mit denen der 'genetische Text' geschrieben ist.
Einige Phänomene der Vererbung lassen sich nicht mit dem gerade beschriebenen DNA-Modell erklären:
- Bei der Zelldifferenzierung entstehen im Verlauf von Zellteilungen Tochterzellen mit unterschiedlicher Funktion, obwohl das Erbgut in allen Zellen gleich ist. Die Festlegung der funktionellen Identität einer Zelle ist ein Thema der Epigenetik.
- Es gibt Eigenschaften, die nur vom Vater her (paternal) vererbt werden, so wie es Eigenschaften gibt, die nur von der Mutter (maternal) stammen und die nicht mit der Basensequenz in Zusammenhang stehen. Störungen dieses Zustandes führen zu schweren Krankheiten.
- Es gibt eine Rückumwandlung von funktionell festgelegten Zellen in undifferenzierte Zellen, die sich wieder in verschiedene Zellen entwickeln können und die z. B. für die Klonierung von "Dolly", also bei der Klonierung von Individuen eingesetzt werden. Da auch hier die Reihenfolge der vier Basen sich nicht ändert, müssen epigenetische Fixierungen aufgehoben werden, damit eine Zelle wieder alle bzw. viele Funktionen erwerben und weitergeben kann.
Epigenetik in der Natur
[Bearbeiten | Quelltext bearbeiten]Funktionen epigenetischer Zellentwicklung
[Bearbeiten | Quelltext bearbeiten]Die Hauptfunktion epigenetischer Veränderungen besteht in der Zelldifferenzierung, also der Anpassung einzelner Zellen eines vielzelligen Lebewesens an verschiedene Aufgaben. In der individuellen Entwicklung teilt sich die Eizelle nach der Befruchtung. Dabei sind bis zum Stadium der Morula alle Tochterzellen gleich. Danach finden sich Zellen mit einem unterschiedlichen inneren Programm und jeweils eigener Funktion. Wenn bei Tieren bzw. dem Menschen der Körper fertig ausgebildet ist, sind die meisten Körperzellen für ihre Funktion fest programmiert. Dabei bleibt der genetische Code des Erbgutes unverändert. Ebenso wie bei Mensch und Tieren gehen auch bei Pflanzen solche Zelldifferenzierungen damit einher, dass Teile der Erbsubstanz mit einer Ableseblockade versehen werden.
Neben dieser Hauptbedeutung können epigenetische Veränderungen auch als Anpassungen eines Lebenwesens an verschiedene Umweltbedingungen interpretiert werden. Hierzu zwei Beispiele:
- Die Maispflanze Zea mays kann durch teilweises Schließen ihrer 'Poren' auch unter besonders trockenen Bedingungen wachsen. Dabei wird das nun ebenfalls schlechter aufgenommene, aber notwendige Kohlendioxid durch besondere Vorgänge, den C4-Mechanismus, um das Kohlendioxid-verwertende Enzym herum angereichert. Die Baupläne für die am C4-Mechanismus beteiligten Enzyme liegen normalerweise epigenetisch 'blockiert' auf der DNA. Eine intensive Bestrahlung mit Licht lockert nun die DNA-Blockade, so dass die Maispflanze die Enzyme bildet und den Mechanismus in Gang setzt. [2]
- Epigenetisch basierte Veränderungen der Fellfarbe aufgrund verschiedener Umweltbedingungen finden sich bei Katzen und Nagetieren. So können zwei eineiige Zwillingskatzen (deren Erbsubstanz also identisch ist) durch verschiedene Fütterung verschiedene Fellfarben ausbilden.[3]
Solche epigenetischen Umwelteinwirkungen können also sehr funktional erscheinende Anpassungen sein (wie im Falle der Maispflanze) oder Veränderungen im Phänotyp, deren Sinnhaftigkeit weniger deutlich sind (wie im Falle der Fellfarben).
Mechanismen und Prozesse epigenetischer Veränderungen
[Bearbeiten | Quelltext bearbeiten]Epigentische Vererbung beruht auf verschiedenen molekularbiologischen Mechanismen.
Zum Einen kann die DNA durch chemische oder räumliche Veränderungen in schwer ablesbare Formen gebracht werden. Hierzu zählt
- die DNA-Methylierung, bei dem ein chemischer Rest an die Erbsubstanz gehängt wird
- die Bildung von Heterochromatin, bei der die DNA dichter gepackt und um Histon-Bausteine gewickelt wird. Hiermit ist auch die Regulation von Histonmodifikationen verbunden.
Zum anderen kann die weitere Verwertung der Erbinformation nach ihrer ersten Ablesung (der Transkription) beschränkt oder verhindert werden. Dies geschieht durch den aktiven Abbau von Ablesekopien, der m-RNA.
Diese drei (sowie weitere, noch nicht genauer erforschte) Mechanismen liegen verschiedenen epigenetischen Prozessen zugrunde. Man zählt hierzu die Paramutation, das Bookmarking, das Imprinting, das Gen-Silencing, die X-Inaktivierung, den Positionseffekt, die Reprogrammierung, die Transvection, maternale Effekte, den Prozess der Karzinogenese, viele Effekte von teratogenen Substanzen sowie technische Limitierungen beim Klonen.
Vererbung epigenetischer Prägungen
[Bearbeiten | Quelltext bearbeiten]Die weit überwiegende Zahl natürlicher epigenetischer Veränderungen an der Erbsubstanz wird nach heutigem Wissensstand (2010) nicht weiter vererbt. Dennoch gibt es inzwischen einige Beispiele für eine Weitergabe epigenetischer Prägungen durch Vererbung von einer Generation auf die nächste.
Beispielsweise unterzogen Forscher in den USA Mäuse einer Lern-Therapie, die zunächst eine genetisch bedingte Gedächnisschwäche ausgleichen konnte. Bei der Untersuchung der (direkt nach der Geburt von den Muttertieren getrennten) Nachkommen zeigten diese den selben Lerneffekt bezüglich ihrer Gedächnisschwäche, wie ihre Mütter. [4]
In einer anderen Studie zeigte sich ein Einfluss der Ernährung von Mäusen auf die Fellfarbe ihrer Nachkommen. Bekamen die Mütter eine besonders vitaminreiche Kost, so hatten die Nachkommen regelmäßig ein dukleres Fell. Parallel hierzu scheint ein besonderer Genabschnitt der mütterlichen DNA (das "Aguti-Gen") durch die vitaminreiche Kost häufiger Methyl-Moleküle zu tragen - eine bereits bekannte Form epigentischer Erbgut-Veränderungen. Sollte sich das Beispiel bestätigen, wäre ein wichtiger Schritt in der Frage erreicht, ob epigenetische Veränderungen tatsächlich vererbbar sind: So verweisen Kritiker zu recht darauf, dass bislang kein biologischer Mechanismus bekannt ist, wie Umwelteinflüsse und erworbene Eigenschaften eigentlich die nachfolgenden Generationen prägen sollen. Die umweltabhängige Methylierung beim Aguti-Gen könnte bei Beispiel eines solchen Mechanismus darstellen.[5]
So könnten Veränderungen bei Pigmenten der Maispflanze bzw. der Tomate auf Epigenetik beruhen.
Eine klassische Evolutionstheorie, nach der erlernte und erworbene Fähigkeiten von einer Generation zur anderen direkt vererbt werden könnten, der Lamarckismus, kann insgeseamt mit den heute bekannten Beispielen epigenetischer Prozesse eher nicht belegt werden.
Dennoch stellt die epigenetische Forschung eine klassische Hypothese der Genetik, das Bestehen der Weismann-Barriere zunehmend in Frage. Nach dieser Hypothese sollten erworbene Eigenschaften eines Induviduums niemals Einfluss auf die Erbinformationen der Keimzellen, und damit der Nachkommen, haben können. Bestätigen sich die heute bekannten Beispiele epigenetischer Vererbung, so muss die Weissmann-Hypothese revidiert oder umformuliert werden.
?? Paramutation, TGS/PTGS, transgenerationelle Effekte bei Pflanzen, PEV und entwicklungsgesteuerte Kontrolle von Chromatinstrukturen in Drosophila.
Epigenetische Forschung
[Bearbeiten | Quelltext bearbeiten]Zur epigenetischen Forschung können eine Reihe von Forschungsaktivitäten und -Vorhaben gezählt werden. Viele solcher Forschungsvorhaben basieren auf biochemischen Untersuchungen an Tier- oder Pflanzenzellen. Daneben gehen Generationen übergreifende Untersuchungen der möglichen Vererbbarkeit epigenetischer Veränderungen nach.
Ziele epigenetischer Eingriffe
[Bearbeiten | Quelltext bearbeiten]Ein derzeitiges Hauptziel in der eipigenetischen Forschung ist die Bildung von Stammzellen aus bereits differenzierten Zellen. Jede intakte Zelle enthält die gesamte Erbinformation des Lebenwesens. Die Ablesung ist jedoch bei allen differenzierten Zellen zu einem Teil blockiert. Gelingt es, diese Ablesehemmnisse rückgängig zu machen, so entstehen Zellen, die sich wiederum zu allen möglichen Körperzellen ausdifferenzieren können.
Hintergrund solcher Bemühungen sind insbesondere Hoffnungen auf neue medizinische Behandlungsmöglichkeiten. So könnte z.B. - so die Hoffnung in Forscherkreisen - einem Patient mit einer Lebererkrankung solche Stammzellen eingepflanzt werden. Diese könnten sich zu neuen, gesunden Leberzellen entwickeln. Da die epigenetische Forschung allerdings noch nicht viel über die genauen Regulationsmechanismen bei der Zelldifferenzierung weiß, wären solche Behandlungen heute mit erheblichen Risiken verbunden. So könnten die eingepflanzten Stammzellen sich auch in eine andere Richtung differenzieren oder zu Krebszellen werden.
Ein besonderes Feld epigentischer Forschung ist jedoch auch, die Ursachen für Krebs zu erforschen, der auf einem gerade nicht (richtig) regulierten Wachstum von Zellen basiert. So gibt es Hinweise, dass bei Krebszellen die normale epigenetische Wachstums- und Teilungs-Regulation gestört ist. So zeigte eine Arbeitsgruppe um Tryndiak, dass bei Körperzellen auf dem Weg zum Mammakarzinom ein fortschreitender globaler Verlust von DNA-Methylierung mit einer fehlgeleiteten Bildung eines bindenden Proteins und Veränderungen in den Histonen einhergeht.[6]
Hier könnten, so Ziel und Hoffnung epigenetischer und pharmazeutischer Forschung, Behandlungsmethoden bzw. Medikamente zur Entwicklung kommen, die eine solche Regulationsstörung beheben oder unterdrücken. Auch hier scheint jedoch die Forschung aufgrund der Komplexität der Regulationsmechanismen kaum baldige sichere Ergebnisse zu liefern.
Methoden epigenetischer Eingriffe
[Bearbeiten | Quelltext bearbeiten]Die epigenetische Forschung arbeitet mit verschiedenen Methoden, die auf der molekularbiologischen Ebene die betreffende DNA in erster Linie in ihrem Aktivitätsstatus untersucht oder diesen zu verändern trachtet.
Untersuchungs-Methoden zum epigenetischen Status von Zellen
[Bearbeiten | Quelltext bearbeiten]- Chromatin-Immunopräzipitation: Dieses Verfahren weist nach, ob ein spezifisches Protein an einen DNA-Abschnitt gebunden war. War das Protein z.B. eine RNA-Polymerase, so muss der Genabschnitt vorher aktiv (also nicht abgeschaltet gewesen) sein. War hingegen ein CG-bindendes Protein (sog. MeCP) angeheftet, so lag der DNA-Abschnitt in abgeschalteter Form vor.
- Bi-Sulfit-Sequenzierung und die Reaktion mit Restriktionsenzymen: Diese beiden Verfahren weisen eine ursprünglich bestehende, den DNA-Abschnitt deaktivierende, Methylierung nach.
- Gelshift-Elektrophorese: Hiermit wird allgemeiner nachgewiesen, ob ein DNA-Abschnitt mit einem (z.B. regulatorischen) Protein verbunden war, oder frei und damit aktiv vorlag. Auch über die Proteingröße gibt das Verfahren Auskunft.
Methoden der epigenetischen Veränderung von DNA
[Bearbeiten | Quelltext bearbeiten]- DNA-Methylierung: Dieses Verfahren ahmt den biologischen Haupt-Markierungsprozess, das Anheften einer Methylgruppe, nach und schaltet damit DNA-Abschnitte aus.
- De-Methylierung: Sie hebt umgekehrt Blockierungen von DNA-Abschnitten auf, indem die Methylgruppe und reaktiviert diese damit.
- Target-Screenings, meist als sog. HTS durchgeführt: Hierbei werden veschiedene Proteine daraufhin untersucht, ob sie eine biologische Aktivität besitzen, insbesondere, ob sie deaktivierend an DNA-Abschnitte binden können. Ziel kann damit sein, regulatorische Proteine zu finden.
Bi-Sulfit-Sequenzierung
[Bearbeiten | Quelltext bearbeiten]Durch Behandlung von DNA mit Natriumhydrogensulfit (alter Name Bisulfit) werden Cytidine (C) in Uracil (U) umgewandelt. Bei einer anschließenden Sequenzierung findet man daher an den Stellen, wo vorher ein C war, nun ein U/T. Da bisulfit-behandelte DNA sehr labil ist, wird daher das Gen, das man analysieren möchte, mittels PCR wieder amplifiziert. Bei der nachfolgenden Sequenzierung werden dann T bzw. TG (Thymin-Guanosin-Dimere) identifiziert, wo in der unbehandelten DNA Cytosin bzw. CG-Dimere existierten.
Für die epigenetische Analyse ist wichtig, dass nur nicht-methylierte C-Basen konvertiert werden, während meC in CG-Dimeren nicht in Thymin konvertiert werden. Man kann daher mit dieser Methode exakt analysieren, welche CG-Dimere in einer bestimmten Zelle methyliert waren. Indem man das bisulfit-behandelte Genstück, das man analysieren möchte, nach der PCR-Amplifikation kloniert und verschiedene Klone sequenziert, erhält man ein Abschätzung, ob ein bestimmtes CG-Dimer gar nicht, vollständig oder nur partiell methyliert war. Bei der Methode des Pyrosequencing ist dieses Verfahren noch verfeinert und erlaubt genauere quantitative Aussagen: Man kann zum Beispiel den Schweregrad einer Tumorentartung mit dem Methylierungsgrad von CG-Inseln einzelner sogenannter Tumor-Suppressorproteine vergleichen und stellt fest, dass in bestimmten Tumoren des blutbildenden System (Hämatopoietisches System) bestimmte meCG-Dimere mit steigenden Schweregrad immer stärker methyliert sind.
Einzelnachweise
[Bearbeiten | Quelltext bearbeiten]- ↑ Rudolf Hagemann: Epigenetik und Lamarckismus haben nichts gemeinsam! In: Laborjournal 4/2009; S. 12
- ↑ Rainer Kalamajka 2005: Die epigenetische Regulation des C4–Syndroms: Belichtungsabhängige Chromatinveränderungen am Promotor der Phosphoenolpyruvat-Carboxylase aus Mais (Zea mays L.). Dissertation an der Rheinisch–Westfälischen Technischen Hochschule Aachen
- ↑ Epigenetische Vererbung - die Rehabilitierung von Lamarck
- ↑ Mütter können Erfahrungen vererben
- ↑ Großvaters Erblast. Können erworbene Eigenschaften vererbt werden? Unsinn, sagen die Darwinisten. Aber die Wirklichkeit ist komplizierter als das Dogma.
- ↑ Tryndiak, VP. et al. (2006): Loss of DNA methylation and histone H4 lysine 20 trimethylation in human breast cancer cells is associated with aberrant expression of DNA methyltransferase 1, Suv4-20H2 histone methyltransferase and methyl-binding proteins. In: Cancer Biol Ther. 5(1), 65–70; PMID 16322686; PDF (freier Volltextzugriff, engl.)
Übersichtsartikel
[Bearbeiten | Quelltext bearbeiten]- Bradbury, J. (2003): Human Epigenome Project—Up and Running. In: PLoS Biol. 1:e82 doi:10.1371/journal.pbio.0000082; PDF (freier Volltextzugriff, engl.)
- Costa, FF. (2008): Non-coding RNAs, epigenetics and complexity. In: Gene 410(1); 9–17; PMID 18226475; doi:10.1016/j.gene.2007.12.008
- Jablonka, E. und Lamb, MJ. (2002): The Changing Concept of Epigenetics. In: Annals of the New York Academy of Sciences 981:82–96; PMID 12547675;
- Delcuve, GP. et al. (2009): Epigenetic control. In: J Cell Physiol. 219(2); 243–250; PMID 19127539; PDF (freier Volltextzugriff, engl.)
Restriktionsendonukleasen, die nur an demethylierten CG-Dimeren schneiden
[Bearbeiten | Quelltext bearbeiten]HpaII (Die zweite Restriktionsendonuklease aus Haemophilus parainfluenza) schneidet CCGG-Palindrome nur, wenn die CG-Dimere nicht methyliert sind, im Vergleich zu BsiSI (aus Bacillus), die auch methylierte CmeCGG-Palindrome schneidet. Tryndiak und Mitarbeiter zeigen damit, dass bei Zellen auf dem Weg zum Mammakarzinom ein fortschreitender globaler Verlust von DNA-Methylierung mit einer fehlgeleiteten Bildung der DNMT1, meCG-bindender Protein und Veränderungen in den Histonen einhergeht.[1]
Bei dieser Methode kann man bestimmen, ob ein bestimmtes Proteine an ein gegebenes DNA-Stück bindet: Durch Behandlung mit Formaldehyd werden die bindenden Proteine mit der DNA chemisch verknüpft. Durch Behandlung mit Ultraschall wird die DNA dann in Bruchstücke von 50 bis 1000 Basenpaare fragmentiert mit den gebundenen Proteinen an der DNA hängend. In einem nächsten Schritt werden mit Antikörpern gegen das bindende Proteine die an ein Protein gebundenen Gene gefischt. Nach der Auftrennung der chemischen Verknüpfung durch Hitzebehandlung in 300 mM Kochsalz-Lösung kann man mit einer PCR das interessierende Genstück amplifizieren und schließlich bestimmen, ob eine Amplifikation stattgefunden hat oder nicht. Im ersteren Fall war das Protein an das Gen assoziiert, im zweiten Fall nicht. Je nach dem, welches Protein man mit Antikörpern versucht zu fischen, kann man z.B. sagen:
- die RNA-Polymerase hat an dem Gen gebunden, daher wurde es transkribiert, das Gen war aktiv
- das meCG-bindene Protein (MeCP) war an das Gen gebunden, daher wurde dieses nicht transkribiert und war ruhig gestellt (silencing)
(noch Baustelle)
Die funktionelle Festlegung erfolgt durch biochemische Ergänzungen an einzelnen Basen der Sequenz und der die DNA verpackenden Histone. Solche Veränderungen führen dazu, dass bestimmte Bjx0ereiche des Erbgutes ruhig gestellt, andere dafür leichter in RNA und Protein überführt werden können. Diese Modifizierungen finden sich nur in Körperzellen, nicht aber z. B. in Eizellen oder Spermien. Von Bedeutung ist vor allem die Methylierung von Cytidin-Basen in Cytosin-Guanosin-Nukleotid-Dimeren (CpG) für epigenetische Phänomene. Außerdem spielt die Seitenketten-Methylierung und -Acetylierung von Histonen eine wichtige Rolle.
Die Methylierung der DNA
[Bearbeiten | Quelltext bearbeiten]
Die wichtigste epigenetische Veränderung ist die Methylierung von Cytidin-Basen der DNA.[2] Dabei werden nur solche Cytidine überhaupt methyliert, die innerhalb von Cytosin-Guanosin-Dinukleotiden angetroffen werden. Andere Cytidine werden durch die bekannten menschlichen DNA-Methyltransferasen (DNMT)) nicht verändert.[3]
Während der DNA-Verdopplung vor jeder Zellteilung gibt es den alten DNA-Strang, an dem bestimmte Cytidine methyliert sind, während der neugebildete DNA-Strang noch nicht methyliert ist. Das Enzym DNMT3 methyliert jedes Cytidin in einem halbmethylierten CG/CG-Paar. Eine solche CG-Methylierung führt dazu, dass Methyl-CG-erkennende Proteine an solche meCG-Paare binden. Diese Bindung führt zur Anlagerung weiterer Proteine und zur Verdichtung der Nukleosomen (siehe weiter unten). Dadurch ist die DNA an solchen meCG-Paaren für die RNA-Polymerase nicht ablesbar und das darunterliegende Gen ist inaktiv.
Methylierte Cytidin sind anfällig für Desaminierung, dabei verlieren die Cytidine die Aminogruppe an Position 6 des Ringes. Ein desaminiertes Cytidin ist ein Uracil. Dieses ist keiner der vier DNA-Bausteine Adenosin, Cytosin, Guanosin oder Thymin. Daher wird ein Uracil in der DNA als Fehler erkannt und schnellstens ausgetauscht. Wird aber ein 5-Methylcytidin desaminiert, entsteht daraus ein Thymin, das ein DNA-Baustein ist. Hier kann der DNA-Reparaturapparat nicht erkennen, ob das Thymin oder das gegenüberliegende Guanosin falsch eingebaut ist. Dass diese natürliche Mutation tatsächlich von statten gegangen ist, erkennt man daran, dass die Häufigkeit von CG-Dinukleotiden gegenüber den anderen Dinukleotid-Kombinationen stark reduziert ist.
CG-Dinukleotide treten gehäuft vor allem in den Genbereichen auf, die für die Steuerung von Genen zuständig sind, den Promotoren. Man spricht von CG-Inseln. Der Methylierungsgrad in solchen CG-Inseln ist häufig ein Maß für die Aktivität, mit der das zugehörige Gen transkribiert wird.
DNA-Methylierung bei Bakterien
[Bearbeiten | Quelltext bearbeiten]
Besonders bei Bakterien hat die Adenin-Methylierung eine wichtige Rolle bei der Fehlerkorrektur der frisch replizierten DNA.[4][5] Innerhalb von GATC-Tetrameren wird das Adenin an der 6-Aminogruppe methyliert (vgl. Bild rechts). Manchmal paart ein Thymin mit einem Cytidin anstelle eines Guanins und wird bei der DNA-Verdopplung irrtümlich eingebaut. Diese und andere Fehlpaarungen können von einem Komplex gefunden werden, der den DNA-Strang absucht und eine Fehlerkorrektur (proof-reading) einleitet. Hierbei wird der DNA-Strang um den Fehler herausgeschnitten, der keine methylierten Adenine aufweist. Das ausgeschnittene Stück wird schließlich neu synthetisiert.
Ist das proof-reading abgeschlossen, werden die Adenine im neuen Strang methyliert.
DNA-Methylierung bei Eukaryoten
[Bearbeiten | Quelltext bearbeiten]Anders als bei Bakterien benutzt der DNA-Reparatur-Apparat der Eukaryoten nicht hemimethylierte GATC-Palindrome, um den neuen von dem alten Strang zu unterscheiden, sondern methylierte CG-Dimere. Bei C→T-Punktmutationen außerhalb von CG-Inseln kann es aber zu Irrtümern kommen, wenn das nächste CG zu weit entfernt ist. Wahrscheinlich sind deshalb die methylierten CG-Dimere nur innerhalb von CG-Inseln stabil.
CG-Methylierung spielt bei der Promotor-Inaktivierung, der Chromatin-Kondensierung, dem genomischen Imprinting und der X-Chromosom-Inaktivierung eine wichtige Rolle [6] Im Gegensatz zu Hefen, Fliegen wie Drosophila oder Nematoden wie Caenorhabditis elegans finden sich bei Pflanzen und bei den Vertebraten näher verwandten Echinodermata CH3CG-Methylierungen. .
Bislang sind drei menschliche DNA-Methyltransferasen bekannt: DNMT1, DNMT3a und DNMT3b (DNMT2 methyliert RNA). Für die Erhaltungs-Methylierung (Maintenance-Methylierung) bei der Zellteilung sind DNMT3a und DNMT3b zuständig. DNMT1 methyliert die CG-Dimere, die aufgrund von Zelldifferenzierungen neu methyliert werden (de-novo-Methylierung). Mutationen im DNMT3b-Gen auf Chromosom 20 führen zum Immunschwäche/zentromere Instabilität/Gesichtsausdrucks-Anomalie-Syndrom (ICF). An methylierte DNA kann sich das Methyl-bindende Protein (MeCP) anlagern. Dieses wiederum ist Keim für weitere Proteinanlagerungen, die schließlich auch zur Modifizierung von Histonen führen. Kondensiertes Histon in Zusammenarbeit mit dem durch MeCP ausgelösten Proteinkomplex führt zur Inaktivierung eines Chromosomenabschnittes.
Auch das Methyl-abspaltende Enzym DNA-Methylase wurde identifiziert.[7] Es war als Methyl-CpG-Domäne-bindendes Protein 2 (MBD2) schon früher beschrieben worden. Damit ist die Methylierung von DNA keine Einbahnstraße, sondern der Methylierungszustand kann Zellfunktions-abhängig geregelt werden. Eine solche Situation nennt man plastisch.
Regulation der DNA-Methylierung in Tumoren
[Bearbeiten | Quelltext bearbeiten]- Die Analyse der DNA-Methylierung von Tumorzellen hat ergeben, dass in Tumorzellen häufig die Gene für sogenannte Tumorsuppressorproteine im Vergleich zu Normalzellen methyliert sind.
- So ist in der akuten myeloischen Leukämie (AML) häufig die CG-Insel des P15-Proteins (auch CDKN2B oder ink4b genannt) methyliert.
- P15 ist ein hemmender Regulator des Zellzyklus.
- Nach Bildung von meCG in der CG-Insel von P15 wird dessen Transkription und die Biosynthese des P15-Proteins eingestellt.
- Beim Zellzyklus-Regulator P53 ist in 50 % aller menschlichen Tumoren das P53-Gen hypermethyliert und damit inaktiviert.[2]
- Da P53 das proof-reading kontrolliert, wird durch Ausschalten von P53 die Fehlerkontrolle aufgegeben und Mutationen können sich anhäufen, die zur Ausschaltung weiterer Tumorsuppressor-Gene oder zur Aktivierung zellwachstums-fördernder Proteine führen können.
Andererseits ist in Tumorzellen die globale DNA-Methylierung geringer als in Normalzellen. Das führt man darauf zurück, dass das in Normalzellen hochmethylierte Heterochromatin (vor allem die Zentromer-Region) in Tumorzellen geringer methyliert ist.
Seitdem man den Einfluss der Hypermethylierung auf das Tumorwachstum identifiziert hat, hat man nach Wegen gesucht, um durch Demethylierung die im Entstehen begriffenen bzw. auch schon existierende Tumore wieder der Zellzykluskontrolle zu unterwerfen:[8]
- Cytosin-ähnliche Substanzen wie Azacytosin oder Aza-Desoxy-Cytosin werden in Patienten mit Akuter Myeloischer Leukämie infundiert.
- Diese Stoffe werden in Zellen aufgenommen, deren DNA verdoppelt wird.
- Azacytosin kann in der Zelle in Aza-Desoxy-Cytosin umgewandelt werden.
- Aza-Desoxy-Cytosin wird anstelle von Cytosin in DNA eingebaut.
- Die DNA-Methylase, die die hemimethylierten CGs methylieren will, bindet an das Aza-Analog.
- Der Austausch von Kohlenstoff gegen Stickstoff bewirkt, dass das Enzym bei dem enzymatischen Methyltransfer an der DNA hängenbleibt und keine weiteren Reaktionen durchführen kann.
- Mit diesem Verfahren werden die DNA-Methylasen inaktiviert und eliminiert. Eine Methylierung findet nicht mehr statt.
- Nach der nächsten Zellteilung ist die DNA weniger methyliert. Wenn von dieser De-Methylierung z.B. das P53- oder das P15-Gen betroffen sind, findet wieder Zellzykluskontrolle statt.
- Das Tumorwachstum ist damit unterbunden.
Es wurden klinische Studien veröffentlicht, in denen bei menschlichen Patienten ein hemmender Effekt von Aza-Desoxy-Cytosin auf Tumorentwicklung gezeigt werden konnte.[9] Die Forscher nennen ihr Verfahren Epigenetische Therapie.
Für die Behandlung des Myelo-Dysplastischen Syndroms, das sich häufig zu einer Akuten Myeloischen Leukämie entwickelt, wurde 5-Aza-2'-Dexoxy-Cytosin unter dem Namen Dacogen von der FDA im Jahre 2006 als Medikament freigegeben Dacogen-Freigabe durch die FDA. Ein anderer Name für diese Substanz ist Decitabine.
Histone und ihre Rolle bei der Epigenetischen Fixierung
[Bearbeiten | Quelltext bearbeiten]
DNA liegt im Zellkern nicht nackt vor, sondern ist an Histone gebunden. Acht verschiedene Histonproteine, jeweils zwei Moleküle von Histon 2a, Histon 2b, Histon 3 und Histon 4 bilden den Kern eines Nukleosoms, auf das etwa 170 Basenpaare eines DNA-Stranges aufgespult sind. Die Enden der Histonstränge ragen aus dem Nukleosom heraus und sind Ziel von Histon-modifizierenden Enzymen. Vor allem Methylierungen und Acetylierungen an Lysin, Histidin oder Arginin, außerdem Phosphorylierungen an Serinen sind die bekannten Modifizierungen. Außerdem spielt eine Rolle, ob die Lysin-Seitenkette mit ein, zwei oder drei Methyl-Gruppen belegt ist. Durch vergleichende Analyse hat man einen Histon-Code entziffert, der in direktem Zusammenhang mit der Aktivität des von den Histonen jeweils gebundenen Gens steht.
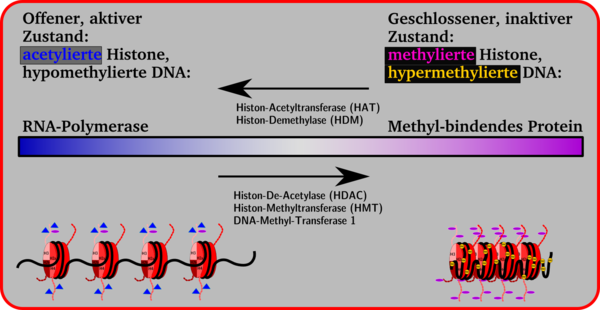
Generell kann man sagen, dass Anheftung von Acetyl-Gruppen an die Lysin-Seitenketten der Histone zur Öffnung der Nukleosomen-Konformation führt, wodurch das Gen für die Transkription durch die RNA-Polymerase verfügbar wird. Durch eine verstärkte Anheftung von Methyl-Gruppen an Lysin-Seitenketten werden Proteine angeheftet wie z. B. das Methyl-bindende Protein MeCB, die die Genexpression unterdrücken, reprimieren, daher auch Repressorproteine genannt, wodurch die Histon-Konformation geschlossen wird und keine Transkription möglich ist.
Einzelnachweise
[Bearbeiten | Quelltext bearbeiten]- ↑ Tryndiak, VP. et al. (2006): Loss of DNA methylation and histone H4 lysine 20 trimethylation in human breast cancer cells is associated with aberrant expression of DNA methyltransferase 1, Suv4-20H2 histone methyltransferase and methyl-binding proteins. In: Cancer Biol Ther. 5(1), 65–70; PMID 16322686; PDF (freier Volltextzugriff, engl.)
- ↑ a b Jeltsch, A. (2002): Beyond Watson and Crick: DNA methylation and molecular enzymology of DNA methyltransferases. In: ChemBioChem 3(4); 274–293; PMID 11933228
- ↑ Jeltsch, A. (2006): Molecular enzymology of mammalian DNA methyltransferases. In: Curr Top Microbiol Immunol. 301; 203–225; PMID 16570849
- ↑ Barras, F. und Marinus, MG. (1989): The great GATC: DNA methylation in E. coli. In: Trends Genet. 5(5); 139–143; PMID 2667217
- ↑ Marinus, MG. und Casadesus, J. (2009): Roles of DNA adenine methylation in host-pathogen interactions: mismatch repair, transcriptional regulation, and more. In: FEMS Microbiol Rev. 33(3); 488–503; PMID 19175412; doi:10.1111/j.1574-6976.2008.00159.x
- ↑ Mathieu, O. et al. (2002): Methylation of a euchromatin-heterochromatin transition region in Arabidopsis thaliana chromosome 5 left arm. In: Chromosome Research 10(6); 455–466; PMID 12489828; doi:10.1023/A:1020936229771
- ↑ Cervoni, N. et al. (1999): DNA demethylase is a processive enzyme. In: J. Biol Chem. 274(13); 8363–8366; PMID 10085064; PDF (freier Volltextzugriff, engl)
- ↑ Momparler, RL. und Bovenzi, V. (2000): DNA methylation and cancer. In: J. Cell Physiol. 183(2); 145–154; PMID 10737890
- ↑ Wijermans, PV. et al. (2005): An epigenetic approach to the treatment of advanced MDS; the experience with the DNA demethylating agent 5-aza-2'-deoxycytidine (decitabine) in 177 patients. In: Ann. Hematol. 84(Suppl1); 9–17; PMID 16211386; doi:10.1007/s00277-005-0012-1
- ↑ Clapier, CR. et al. (2007): Structure of the Drosophila nucleosome core particle highlights evolutionary constraints on the H2A-H2B histone dimer. In: Proteins 71(1); 1–7]; PMID 17957772; PDF (freier Volltextzugriff, engl.)
Übersichtsartikel
[Bearbeiten | Quelltext bearbeiten]- Bradbury, J. (2003): Human Epigenome Project—Up and Running. In: PLoS Biol. 1:e82 doi:10.1371/journal.pbio.0000082; PDF (freier Volltextzugriff, engl.)
- Costa, FF. (2008): Non-coding RNAs, epigenetics and complexity. In: Gene 410(1); 9–17; PMID 18226475; doi:10.1016/j.gene.2007.12.008
- Jablonka, E. und Lamb, MJ. (2002): The Changing Concept of Epigenetics. In: Annals of the New York Academy of Sciences 981:82–96; PMID 12547675;
- Delcuve, GP. et al. (2009): Epigenetic control. In: J Cell Physiol. 219(2); 243–250; PMID 19127539; PDF (freier Volltextzugriff, engl.)
- Marmorstein, R. und Trievel, RC. (2009): Histone modifying enzymes: structures, mechanisms, and specificities. In: Biochim Biophys Acta 1789(1); 58–68; PMID 1872256; doi:10.1016/j.bbagrm.2008.07.009
- Morgan, HD. et al. (2005): Epigenetic reprogramming in mammals. In: Hum Mol Genet. 14 Spec No 1; R47–58; PMID 15809273; PDF (freier Volltextzugriff, engl.)
Nachschlagewerke
[Bearbeiten | Quelltext bearbeiten]- Oscar Hertwig (1849-1922): Biological problem of today: preformation or epigenesis? The basis of a theory of organic development. W. Heinemann: London, 1896.
- Joachim Bauer: Das Gedächtnis des Körpers: wie Beziehungen und Lebensstile unsere Gene steuern. Frankfurt a.M.: Eichborn, 2002. Erweiterte Taschenbuchausgabe München: Piper, 2004 (10. Aufl. 2007). ISBN 978-3-492-24179-3
- Wolfgang Wieser: Gehirn und Genom: ein neues Drehbuch für die Evolution. München: C.H. Beck, 2007 ISBN 3406556345
Weblinks
[Bearbeiten | Quelltext bearbeiten]- The Epigenome Network of Excellence (enthält Verweise auf aktuelle Veröffentlichungen) (engl.)
- Epigenetische Vererbung - die Rehabilitierung von Lamarck
- wissenschaft.de: Epigenetik - Vererbung ist mehr als die Summe der Gene
- Deutschlandfunk - Wie die Epigenetik die Biologie revolutioniert
- Spiegel Online: Bruch des bösen Zaubers - Wie Kindheitserfahrungen Spuren im Erbgut hinterlassen (Epigenetik)
Kategorie:Biologie Kategorie:Epigenetik Kategorie:Evolution Kategorie:Genetik