DPANN
| DPANN | ||||||
|---|---|---|---|---|---|---|
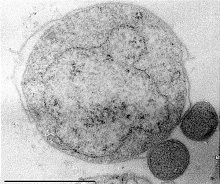
Nanoarchaeum equitans (die kleinen Symbionten auf der rechten Seite) | ||||||
| Systematik | ||||||
| ||||||
| Wissenschaftlicher Name | ||||||
| DPANN | ||||||
| Rinke et al., 2013 |

DPANN ist ein Superphylum extremophiler Archaeen („Urbakterien“).[1][2]
Das Taxon wurde 2013 von Christian Rinke et al. vorgeschlagen.[3] DPANN ist ein Akronym, gebildet aus den Anfangsbuchstaben der ersten fünf gefundenen Stämme (Phyla), Diapherotrites, Parvarchaeota, Aenigmarchaeota, Nanoarchaeota und Nanohaloarchaeota. Später entdeckten Phyla wie Woesearchaeota, Pacearchaeota[4] sowie Altiarchaeota[5] wurden nachträglich als weitere Mitglieder dieses Superphylums vorgeschlagen.
Das Superphylum DPANN vereinigt verschiedene Archaeenstämme mit einer großen Verbreitung und unterschiedlichem Stoffwechsel, welche von symbiotischen und thermophilen Formen wie den Nanoarchaeota, über acidophile (säureliebende) wie Parvarchaeota bis hin zu nicht-extremophilen wie Aenigmarchaeota und Diapherotrites reichen. Das Phylum beherbergt auch eine Reihe früher in die Euryarchaeota gestellte Gruppen.[6] Viele Mitglieder zeigen neuartige Anzeichen eines horizontalen Gentransfers aus anderen Domänen (Bakterien, Eukaryonten) des Lebens.[3]
DPANN-Archaeen wurden 2022 in der mikrobiellen Gemeinschaft der bis dato weltweit kältesten und salzigsten Quelle, Lost Hammer Spring (Axel Heiberg Island, Nunavut, Kanada) gefunden. Die Verhältnisse dort sind ähnlich, wie man sie an einigen Stellen des Mars, sowie auf den Monden Europa und Enceladus vermutet.[7]
Systematik
[Bearbeiten | Quelltext bearbeiten]Das Superphylum DPANN gliedert sich nach Dombrowski et al. (2020)[8], sowie Castelle und Banfield (2018),[9] ergänzt nach National Center for Biotechnology Information (NCBI)[10] und GTDB:[11]
- Cluster 1
- Phylum Altiarchaeota mit Spezies Altiarchaeum hamiconexum[12]
- Micrarchaeota-Diapherotrites-Gruppe, ehemals als MEG-Cluster zu den Euryarchaeota[6][8]
- Phylum Diapherotrites (Rinke et al. 2013, alias Iainarchaeota) mit Spezies Forterrea multitransposorum und Iainarchaeum andersonii[12][13]
- Phylum Micrarchaeota (Baker et al.) 2010 mit Spezies Mancarchaeum acidiphilum[12] und Micrarchaeum acidiphilum
- Cluster 2
- Phylum Aenigmarchaeota (Rinke et al. 2013), ehemals DSEG-Cluster und zu den Euryarchaeota,[6] mit Spezies Aenigmarchaeum subterraneum[14]
- Phylum Huberarchaeota (alias Huberarchaea[15]) mit Spezies Huberarchaeum crystalense[12]
- Gruppe ARMAN[4]
Entdeckt 2006 in stark sauren Abwässern einer US-Mine. Sie sind von sehr geringer Größe.[16][17][18]
- Phylum Parvarchaeota (Rinke et al. 2013) mit Spezies Parvarchaeum acidiphilum und P. paracidiphilum (früher P. acidophilus)[12]
- Phylum Nanoarchaeota (Huber et al. 2002) mit Spezies Nanoarchaeum equitans, Nanopusillus acidilobi und Nanoclepta minutus
- Phylum Nanohaloarchaeota (Rinke et al. 2013) mit Gattung Nanopetraeus sowie den Spezies Nanohalobium constans und Nanohalarchaeum antarcticum[6][12]
- Woesearchaeota-Pacearchaeota-Gruppe, ehemals als DHVEG-6-Cluster zu den Euryarchaeota[6][8]
In Sedimenten und Oberflächengewässern von Aquiferen und Seen und bevorzugen salzhaltige Bedingungen:[4][6]
- Phylum Pacearchaeota (Castelle et al. 2015)
- Phylum Woesearchaeota (Castelle et al. 2015)[19]
- Phylum Undinarchaeota, ehemals UAP2 (Uncultured Archaeal Phylum 2) / MHVG (Marine Hydrothermal Vent Group)[8][20][22][23]
- Ordnung Undinarchaeales mit Spezies Undinarchaeum marinum
(Salzwasser-Vertreter, englisch marine MAGs) - Ordnung Naiadarchaeales mit Spezies Naiadarchaeum limnaeum
(Süßwasser-Vertreter, englisch aquifer MAGs)
- Ordnung Undinarchaeales mit Spezies Undinarchaeum marinum
- Phylum Mamarchaeota
Die obigen Taxa sind in der Regel nur durch Metagenomanalyse belegt (Metagenome-Assembled Genome, MAG) und tragen daher dann den Präfix Candidatus, der hier der Übersichtlichkeit halber weggelassen wurde. Kladogramme der DPANN-Archaeen finden sich bei Parks et al. (2017),[20] Castelle und Banfield (2018)[9] sowie Dombrowski et al. (2020).[8] Letztere Autoren sehen die Woesearchaeota-Pacearchaeota-Gruppe innerhalb der durch Altiarchaeota und Diapherotrites aufgespannten Klade (genannt Cluster 2).[8] Schon länger war bekannt, dass Micrarchaeota und Parvarchaeota nahe verwandt und sind, sie wurde daher ursprünglich in einer Gruppe ARMAN (Archaeal Richmond Mine Acidophilic Nanoorganisms) zusammengefasst.[4] Das Cluster 2 stellt nur eine Erweiterung der in ihrem ursprünglichen Umfang als paraphyletisch angesehenen ARMAN-Gruppe dar.
Weblinks
[Bearbeiten | Quelltext bearbeiten]- Wikispecies:DPANN group
- Two Major Microbial Groups Discovered That Can’t Breathe – May Predate the Evolution of Respiration, auf: SciTechDaily vom 31. August 2020, Quelle: BIGELOW LABORATORY FOR OCEAN SCIENCES.
- Nina Dombrowski, Jun-Hoe Lee, Tom A Williams, Pierre Offre, Anja Spang: Genomic diversity, lifestyles and evolutionary origins of DPANN archaea, in: FEMS Microbiol Lett. 366(2), 9. Januar 2019, fnz008, doi:10.1093/femsle/fnz008, PMC 6349945 (freier Volltext), PMID 30629179 (englisch).
- Bram Henneman: Histone-DNA assemblies in archaea: shaping the genome on the edge of life. Leiden University Repository, 4. Dezember 2019; hdl:1887/81191. Hier: Kapitel 1: Introduction (englisch). Siehe Fig. 1.1.
- Bernhard Tschitschko et al.: Genomic variation and biogeography of Antarctic haloarchaea, in: Microbiome, Band 6, Nr. 113, 2018; doi:10.1186/s40168-018-0495-3 (englisch). Dazu:
- Ricardo Cavicchioli: Behind the paper: Antarctic haloarchaea – a unique Microbiome. In: Microbiology, 19. Juni 2018 (englisch).
Einzelnachweise
[Bearbeiten | Quelltext bearbeiten]- ↑ Cindy J. Castelle, Kelly C. Wrighton, Kenneth H. Williams, Jillian F. Banfield et al.: Genomic Expansion of Domain Archaea Highlights Roles for Organisms from New Phyla in Anaerobic Carbon Cycling. Current Biology, 16. März 2015, abgerufen am 14. Januar 2017 (englisch).
- ↑ Jacob P. Beam, Eric D. Becraft, Julia M. Brown, Frederik Schulz, Jessica K. Jarett, Oliver Bezuidt, Nicole J. Poulton, Kayla Clark, Peter F. Dunfield, Nikolai V. Ravin, John R. Spear, Brian P. Hedlund, Konstantinos A. Kormas, Stefan M. Sievert, Mostafa S. Elshahed, Hazel A. Barton, Matthew B. Stott, Jonathan A. Eisen, Duane P. Moser, Tullis C. Onstott, Tanja Woyke, Ramunas Stepanauskas: Ancestral Absence of Electron Transport Chains in Patescibacteria and DPANN, in: Frontiers in Microbiology, Band 11, 2020, doi:10.3389/fmicb.2020.01848
- ↑ a b Christian Rinke, P. Schwientek, A. Sczyrba, N. N. Ivanova, I. J. Anderson, J. F. Cheng, J. A. Dodsworth, B. P. Hedlund, G. Tsiamis, S. M. Sievert, W. T. Liu, J. A. Eisen, S. J. Hallam, N. C. Kyrpides, R. Stepanauskas, E. M. Rubin, Phil Hugenholtz, Tanja Woyke: Insights into the phylogeny and coding potential of microbial dark matter. In: Nature. 499. Jahrgang, Nr. 7459, Juli 2013, S. 431–437, doi:10.1038/nature12352, PMID 23851394 (englisch, escholarship.org [PDF]).
- ↑ a b c d Cindy J. Castelle, K. C. Wrighton, B. C. Thomas, L. A. Hug, C. T. Brown, M. J Wilkins, K. R. Frischkorn, S. G Tringe, A. Singh, L. M. Markillie, R. C. Taylor, K. H. Williams, Jillian F. Banfield: Genomic Expansion of Domain Archaea Highlights Roles for Organisms from New Phyla in Anaerobic Carbon Cycling. In: Current Biology. 25. Jahrgang, 19. Februar 2015, S. 690–701, doi:10.1016/j.cub.2015.01.014, PMID 25702576 (englisch).
- ↑ Anja Spang, Eva F. Caceres, Thijs J. G. Ettema: Genomic exploration of the diversity, ecology, and evolution of the archaeal domain of life. In: Science. 357. Jahrgang, Nr. 6351, 11. August 2017, S. eaaf3883, doi:10.1126/science.aaf3883, PMID 28798101 (englisch).
- ↑ a b c d e f Rüdiger Ortiz Álvarez, Emilio O. Casamayor: High occurrence of Pacearchaeota and Woesearchaeota (Archaea superphylum DPANN) in the surface waters of oligotrophic high-altitude lakes. In: Environmental Microbiology Reports. 8. Jahrgang, Nr. 2, April 2016, S. 210–217, doi:10.1111/1758-2229.12370, PMID 26711582 (englisch).
- ↑
Elisse Magnuson, Ianina Altshuler, Miguel Á. Fernández-Martínez, Ya-Jou Chen, Catherine Maggiori, Jacqueline Goordial, Lyle G. Whyte: Active lithoautotrophic and methane-oxidizing microbial community in an anoxic, sub-zero, and hypersaline High Arctic spring. In: Nature: The ISME Journal, Band 16, Juli 2022, S. 1798–1808; doi:10.1038/s41396-022-01233-8, Abstract, Epub 8. April 2022. Siehe dazu:
- Michelle Starr: Life Has Been Found in a Low-Oxygen, Super-Salty, Sub-Zero Arctic Spring . Auf: sciencealert vom 24. Juni 2022.
- A blueprint for life forms on Mars?. Auf: ScienceDaily vom 21. Juni 2022. Quelle: McGill University, Montreal.
- Scientists Discover Blueprint for Life Forms on Mars. Auf: SciTechDaily vom 19. Juli 2022. Quelle: McGill University, Montreal.
- ↑ a b c d e f g Nina Dombrowski, Tom A. Williams, Jiarui Sun, Benjamin J. Woodcroft, Jun-Hoe Lee, Bui Quang Minh, Christian Rinke, Anja Spang: Undinarchaeota illuminate DPANN phylogeny and the impact of gene transfer on archaeal evolution. In: Nature Communications, Band 11, Nr. 3939, 7. August 2020; doi:10.1038/s41467-020-17408-w.
- ↑ a b Cindy J. Castelle, Jillian F. Banfield: Major New Microbial Groups Expand Diversity and Alter our Understanding of the Tree of Life. In: Cell. 172. Jahrgang, Nr. 6, 2018, S. 1181–1197, doi:10.1016/j.cell.2018.02.016, PMID 29522741 (englisch, cell.com). Siehe insbes. Fig. 1 B
- ↑ NCBI: DPANN group (archaea); graphisch: DPANN group, auf: Lifemap, NCBI Version.
- ↑ GTDB
- ↑ a b c d e f Joshua N. Hamm, Susanne Erdmann, Emiley A. Eloe-Fadrosh, Allegra Angeloni, Ling Zhong, Christopher Brownlee, Timothy J. Williams, Kirston Barton, Shaun Carswell, Martin A. Smith, Sarah Brazendale, Alyce M. Hancock, Michelle A. Allen, Mark J. Raftery; Norman R. Pace (Hrsg.): Unexpected host dependency of Antarctic Nanohaloarchaeota, in: PNAS 116 (29), Juni/Juli 2019, S. 14661-14670, doi:10.1073/pnas.1905179116, PMID 31253704
- ↑ L. R. Comolli, B. J. Baker, K. H. Downing, C. E. Siegerist, Jillian F. Banfield: Three-dimensional analysis of the structure and ecology of a novel, ultra-small archaeon. In: The ISME Journal. 3. Jahrgang, Nr. 2, Februar 2009, S. 159–167, doi:10.1038/ismej.2008.99, PMID 18946497 (englisch).
- ↑ K. Takai, D. P. Moser, M. DeFlaun, T. C. Onstott, J. K. Fredrickson: Archaeal diversity in waters from deep South African gold mines. In: Applied and Environmental Microbiology. 67. Jahrgang, Nr. 12, Dezember 2001, S. 5750–5760, doi:10.1128/AEM.67.21.5750-5760.2001, PMID 11722932, PMC 93369 (freier Volltext) – (englisch).
- ↑ Uniprot: Huberarchaea
- ↑ B. J. Baker, Gene W. Tyson, R. I. Webb, J. Flanagan, Philip Hugenholtz, E. E. Allen, Jillian F. Banfield: Lineages of acidophilic archaea revealed by community genomic analysis. In: Science. 314. Jahrgang, Nr. 5807, Dezember 2006, S. 1933–1935, doi:10.1126/science.1132690, PMID 17185602 (englisch).
- ↑ S. Murakami, K. Fujishima, M. Tomita, A. Kanai: Metatranscriptomic analysis of microbes in an Oceanfront deep-subsurface hot spring reveals novel small RNAs and type-specific tRNA degradation. In: Applied and Environmental Microbiology. 78. Jahrgang, Nr. 4, Februar 2012, S. 1015–1022, doi:10.1128/AEM.06811-11, PMID 22156430, PMC 3272989 (freier Volltext) – (englisch).
- ↑ B. J. Baker, L. R. Comolli, G. J. Dick, L. J. Hauser, D. Hyatt, B. D. Dill, M. L. Land, N. C. Verberkmoes, R. L. Hettich, Jillian F. Banfield: Enigmatic, ultrasmall, uncultivated Archaea. In: Proceedings of the National Academy of Sciences of the United States of America. 107. Jahrgang, Nr. 19, Mai 2010, S. 8806–8811, doi:10.1073/pnas.0914470107, PMID 20421484, PMC 2889320 (freier Volltext) – (englisch).
- ↑ Charlotte D. Vavourakis, Maliheh Mehrshad, Cherel Balkema, Rutger van Hall, Adrian-Ştefan Andrei, Rohit Ghai, Dimitry Y. Sorokin, Gerard Muyzer: Metagenomes and metatranscriptomes shed new light on the microbial-mediated sulfur cycle in a Siberian soda lake, in: BMC Biology, Band 17, Nr. 69, 22. August 2019, doi:10.1186/s12915-019-0688-7. Anm.: englisch Cock Soda Lake alias Petukhov Soda Lake, ist deutsch Petuchow-Sodasee, russisch Петуховское Содовое озеро, siehe Kulundasteppe
- ↑ a b c Donovan H. Parks, Christian Rinke, Maria Chuvochina, Pierre-Alain Chaumeil, Ben J. Woodcroft, Paul N. Evans, Philip Hugenholtz, Gene W. Tyson: Recovery of nearly 8,000 metagenome-assembled genomes substantially expands the tree of life, in: Nature Microbiology, Band 2, S. 1533–1542, 11. September 2017, doi:10.1038/s41564-017-0012-7, mit Korrektur vom 12. Dezember 2017, doi:10.1038/s41564-017-0083-5
- ↑ NCBI: „UAP1“
- ↑ Taxonomicon: Taxon: Candidate phylum "Uncultured Archaeal Phylum 2 (UAP2/MHVG)"
- ↑ Previously undescribed lineage of Archaea illuminates microbial evolution:
- Royal Netherlands Institute for Sea Research (NIOZ), 10. August 2020
- phys.org, 10. August 2020
- ScienceDaily, 10. August 2020
- EurekAlert!, 10. August 2020