Eukaryogenese
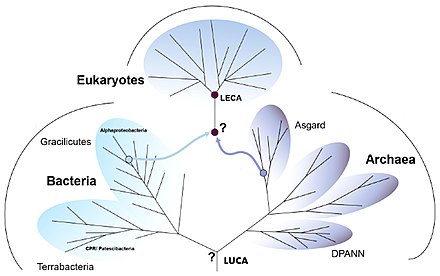
Eukaryogenese bezeichnet den Prozess, der zur Entstehung der eukaryotischen Zelle an der Wurzel des Stammbaums der Eukaryoten als komplex-zellulären Organismen geführt hat. Da die Eukaryoten alle komplex aufgebauten Einzeller und (fast) alle mehrzelligen Organismen umfassen, ist die Eukaryogenese ein Meilenstein in der Evolution. Es herrscht weitgehend Einigkeit darüber, dass es sich bei diesem Prozess um eine Symbiogenese handelte, bei der bestimmte Archaeen und Bakterien zusammenkamen, um den ersten gemeinsamen eukaryotischen Vorfahren (englisch first eukarotic common ancestor, FECA) zu schaffen. Diese Zelle verfügte nach gängigen Vorstellungen über ein neues Maß an Komplexität und Fähigkeiten, unter anderem mit einem Zellkern, mindestens einer Zentriole, fakultativ aeroben Mitochondrien, Sexualität und einer Zellwand. Sie entwickelte sich zunächst zu einer Population von Einzellern, zu der auch der letzte gemeinsame Vorfahre der Eukaryoten (engl. last eukaryotic common ancestor, LECA) gehörte, und erlangte im Laufe der Entwicklung weitere Fähigkeiten. Die Reihenfolge der an diesem Prozess beteiligten Schritte ist allerdings noch umstritten, möglicherweise hat dieser auch nicht mit der Symbiogenese begonnen. Aus dem LECA wiederum ging die Kronengruppe der Eukaryoten hervor, die die Vorfahren von Tieren, Pilzen, Pflanzen und einer Vielzahl von Einzellern umfasst.
Hintergrund
[Bearbeiten | Quelltext bearbeiten]Nachdem sich die Erde so weit abgekühlt hatte, dass sich Ozeane gebildet hatten, konnte das Leben entstehen (siehe Chemische Evolution). Der letzte gemeinsame Vorfahr oder Ahn aller heutigen Lebewesen (LUGA, engl. last universal common ancestor, LUCA) war ein Organismus, der einige allen Lebewesen gemeinsame Eigenschaften vereinigte, insbesondere Ribosomen und den genetischen Code zur Erzeugung von Proteinen nach Muster von Nukleinsäuren (RNA); er lebte vermutlich vor etwa 4 Milliarden Jahren. Aus ihm entstanden zwei Hauptzweige (Domänen) des prokaryotischen Lebens, die Bakterien und die Archaeen. Aus diesen kleinzelligen, sich oft schnell teilenden Vorfahren entwickelten sich mit der Zeit die Eukaryoten mit viel größeren Zellen, Zellkernen und einer ausgeprägten Biochemie (Stoffwechsel).[1][2] Nach einer Auffassung bilden die aus dem Zusammenschluss von Archaeen und Bakterien hervorgegangenen „chimären“ Eukaryoten eine neue, dritte Domäne.[3][4] Andere Vorschläge betonen, dass die Eukaryoten mitten aus den Asgard-Archaeen hervorgegangen sind. Damit diese und die Archaeen insgesamt nicht zu paraphyletische Gruppen werden, sollten die Eukaryoten den Asgard-Archaeen zugeordnet werden, genauso, wie die Vögel den Dinosauriern, von denen sie abstammen.[5] Siehe dazu Urvorfahr: Zwei oder drei Domänen.
Symbiogenese
[Bearbeiten | Quelltext bearbeiten]

MAnc: gemeinsamer Vorfahre der Mitochondrien und ihrer nächsten Verwandtschaft (Alphaproteobakterien).
AAnc: gemeinsamer Vorfahre der eukaryotischen Wirtslinie und ihrer nahen Verwandtschaft (Asgard-Archaeen bzw. ihrer Untergruppe Hodarchaeales).
PAnc: gemeinsamer Vorfahre der Plastiden (Chloroplasten) und ihrer nächsten Verwandtschaft (Cyanobakterien: Oxyphotobacteria).
Nach der von Lynn Margulis zum Durchbruch verholfenen Theorie der Symbiogenese (auch bekannt als Endosymbiontentheorie) verleibte sich ein Mitglied der Archaeen eine Bakterienzelle als Bestandteil ein, ohne diese zu verdauen (siehe Eozyten-Hypothese). Das Bakterium gehörte zu Alphaproteobakterien (unter diesen früher favorisierte Ordnung: Rickettsiales, seit 2023: Iodidimonadales) mit der Fähigkeit Sauerstoff für ihre Atmung zu nutzen. Dies ermöglichte ihm – und damit den Archaeen, die es aufgenommen hatten – das Überleben in Gegenwart von Sauerstoff, denn dieser ist für andere, an reduzierende Bedingungen angepasste (gärende) Organismen giftig. Die endosymbiotischen Bakterien wurden zu den Mitochondrien der eukaryotischen Zelle und lieferten den größten Teil der Energie dieser Zellen.[1][6][8][9] Einen ähnlichen Verschmelzungsprozess, aber mit anderen Stoffwechseldetails, haben William F. Martin und Miklós Müller 1998 vorgeschlagen (Wasserstoff-Hypothese, engl. hydrogen hypothesis).[10] Die Syntrophie-Hypothese betont den Austausch von Stoffwechselprodukten zwischen den Verschmelzungspartnern; ein zusätzlicher Beitrag von Deltaproteobakterien könnte zur Zellmembran der Eukaryoten geführt haben.[11] Nach heutiger Ansicht war die Archaeen-Zelle ein Mitglied der inzwischen entdeckten Asgard-Archaeen und in jüngerer Zeit wurde unter diesen wiederum die Heimdallarchaeen mit ihrer Ordnung Hodarchaeales als die nächste derzeit (Stand 2023) bekannten Verwandten der Eukaryoten identifiziert.[12][5]
Eine zweite Fusion vor 1,6 Milliarden Jahren fügte aus Cyanobakterien hervorgegangene Chloroplasten hinzu und schuf so die grünen Pflanzen.[6][13]
Lynn Margulis und Kollegen vermuteten darüber hinaus, dass die Zelle auch ein Spirochäten-Bakterium als Symbionten erwarb, das ihr das Zellskelett aus Mikrotubuli und die Fähigkeit zur Bewegung verlieh, einschließlich der Fähigkeit, die Chromosomen während der Mitose, der Zellteilung, in zwei Sätze zu zerlegen.[13] Im Gegensatz zu den beiden anderen Teilen der Endosymbiosetheorie konnte diese Vermutung (bislang) jedoch nicht bestätigt werden.
Letzter eukaryotischer gemeinsamer Ahn (LECA)
[Bearbeiten | Quelltext bearbeiten]
Der letzte gemeinsame Vorfahr oder Ahn aller heutigen Eukaryoten wird in Analogie zum LUCA als Last Eukaryotic Common Ancestor (LECA) bezeichnet.[15] Er lebte vor etwa 2 Milliarden Jahren und umfasste sehr wahrscheinlich eine ganze Population.[16]
Es wird angenommen, dass es sich um einen Protisten mit einem Zellkern, mindestens einer Zentriole und einem Cilium, fakultativ aeroben Mitochondrien, Sexualität (Meiose und Syngamie), einer Ruhezyste mit einer Zellwand aus Chitin und/oder Zellulose und Peroxisomen gehandelt hat.[17][18]
Falls der Erwerb des Zellkerns dem der Mitochondrien vorausging, muss es zeitweilig amitochondriale Organismen mit Zellkern gegeben haben, die im Laufe ihrer Evolution dann die Mitochondrien durch Endosymbiose erworben haben (siehe Endosymbiontentheorie). Carl Woese und George Fox führten 1977 für diese hypothetischen Formen von „primitiven“ amöboiden prädatorischen Einzellern den Begriff Urkaryoten (engl. urkaryotes) ein.[19] Diese Urkaryoten galten dann als Begründer der dritten Domäne der Eukaryoten, zu der alle komplexen Zellen (Protisten) und die meisten Arten von mehrzelligen Organismen, einschließlich der Tiere, Pflanzen und Pilze gehören. Thomas Cavalier-Smith führt dann 1989 rezente (heutige) primitive einzellige Eukaryoten ohne Mitochondrien – unter der Annahme, diese seien Abkömmlinge dieser Urkaryoten – die Gruppenbezeichnung Archezoa ein.[20] Allerdings mehrten sich nachfolgend Hinweise darauf, dass es sich bei den Archezoen um sekundär amitochondriale echte Eukaryoten handelt.[21] Zudem werden die Vorfahren der Eukaryoten heute mitten unter den Archaeen verortet und nicht mehr als primitive Urkaryoten abseits von diesen.
Alle bis heute untersuchten Eukaryoten besitzen einen vom Zytoplasma durch eine Kernmembran abgegrenzten Zellkern. Darüber hinaus findet man bei fast allen Eukaryoten Mitochondrien oder Organellen (Hydrogenosom, Mitosom, MLO etc.), die offensichtlich von Mitochondrien abstammen oder mit diesen einen gemeinsamen Vorfahren haben (engl. mitochondrion-related organelles, MROs). Diese Organellen besitzen meist ihre eigene, mitochondriale DNA (mtDNA). Zumindest ließen sich überall – auch bei den wenigen amitochondrialen Eukaryoten – in der Zellkern-DNA Gene mitochondrialen Ursprungs nachweisen. Diese wurden im Lauf der Evolution durch „endosymbiotischen Gentransfer“ (eine Sonderform des lateralen Gentransfers) von heute reduzierten oder ganz verschwundenen Mitochondrien zum Kern transferiert.[22][23] Es konnten somit – bisher – keine „primär amitochondrialen“ Eukaryoten gefunden werden. Daher sollte LECA bereits einen Zellkern und mitochondrienartige Organellen (mit eigener mtDNA) besessen haben.
Brian S. Leander sowie Jürgen F. H. Strassert et al. hatten 2020/2021 vorgeschlagen, dass sich der LECA durch Phagozytose ernährte und andere Mikroorganismen verschlang.[17][18] Nico Bremer et al. konnten jedoch 2022 Belege dafür finden, dass LECA Mitochondrien besaß. Sie vermuteten, dass LECA als Coenocyt über mehrere Kerne verfügte, glaubten jedoch nicht, dass er phagotroph war. Die bei vielen Eukaryoten vorhandene Fähigkeit, (organisches) Material zu verschlingen, hätte sich nach dieser Ansicht erst später entwickelt. Phagocytose hätte daher nicht zum Erwerb der Alphaproteobakterien (als Vorläufer der Mitochondrien) genutzt werden können.[24]
Für LECA wird eine „spektakulären zellulären Komplexität“ vermutet, und diese Zellen waren in Kompartimente unterteilt.[25] Es scheint eine Reihe von Proteinen der Endosomalen Sortierkomplexe für den Transport (englisch endosomal sorting complexes required for transport), ESCRT, geerbt zu haben, die es ermöglichen, Membranen umzugestalten, einschließlich der Abtrennung von Vesikeln zur Bildung von Endosomen.[26] Sein Apparat für die Transkription von DNA in RNA und die anschließende Übersetzung (Translation) der RNA in Proteine war räumlich voneinander getrennt, so dass die Expression von Genen komplexer werden konnte. Es verfügte über Mechanismen, mit denen es sein genetisches Material umordnen und möglicherweise seine eigene Evolutionsfähigkeit manipulieren konnte. All dies verschaffte LECA „ein umfassendes Bündel an Selektionsvorteilen“.[25]
Szenarien
[Bearbeiten | Quelltext bearbeiten]Für die Entstehung der Eukaryoten wurden mehrere Szenarien vorgeschlagen. Es besteht zwar weitgehend Einigkeit darüber, dass der LECA einen Zellkern, Mitochondrien und innere Membranen gehabt haben muss, doch die Reihenfolge, in der diese erworben wurden, wird diskutiert.
Nach dem syntrophen Modell erhielt der erste gemeinsame eukaryotische Vorfahr (FECA, vor etwa 2,2 Milliarden Jahren) Mitochondrien, dann Membranen und schließlich einen Zellkern. Nach dem phagotrophen Modell erhielt er zuerst einen Zellkern, dann Membranen und schließlich Mitochondrien. Das phagotrophe Modell setzt die Fähigkeit voraus, Nahrung zu verschlingen, was die Zelle in die Lage versetzte, das aerobe Bakterium zu verschlingen, aus dem das Mitochondrium wurde.[25] In einem komplexeren Prozess erlangte LECA alle drei Komponenten innerhalb vergleichsweise kurzer Zeit, und erst danach weitere Fähigkeiten. Daneben wurden auch noch andere Modelle vorgeschlagen.
Was auch immer geschah, es dürften zunächst viele Abstammungslinien entstanden sein, so dass LECA zunächst mit diesen konkurrierte – oder verschiedene Linien schlossen sich zusammen, um einen einzigen Ausgangspunkt für die heutigen Eukaryoten zu bilden.[25] Nick Lane und William F. Martin argumentierten mit der Begründung, dass Energie ein begrenzender Faktor für die Größe der prokaryotischen Zelle war, dafür, dass die Mitochondrien zuerst erworben wurden.[27]
Eugene V. Koonin et al. (2005), sowie Joran Martijn und Thijs J. G. Ettema (2013) stellten fest, dass die Archaeen (insbesondere aus der Asgard-Gruppe) viele gemeinsame Merkmale mit Eukaryoten haben. Daher schlugen sie vor, dass rudimentäre eukaryotische Merkmale wie membranartige Kompartimente bereits erworben wurden, bevor die Endosymbiose der frühen eukaryotischen Zelle Mitochondrien hinzufügte, während die Zellwand verloren ging. Der Erwerb der Mitochondrien darf aber so oder so nicht als das Ende des Eukaryogenese-Prozesses angesehen werden, denn nach oder während des endosymbiotischen Verschmelzens mussten noch etliche neue komplexe Genfamilien entwickelt werden. Die Organismen dem Weg vom FECA zum LECA können daher als Protoeukaryoten betrachtet werden. Am Ende des Prozesses war LECA bereits ein komplexer Organismus mit den zellulären Kompartimentierung beteiligten Proteinfamilien.[28][29]
E3-Modell der Eukaryogenese
[Bearbeiten | Quelltext bearbeiten]Der Stamm (en. strain) MK-D1 von Prometheoarchaeum syntrophicum hat ebenso wie der 2022 kultivierte Stamm B-35 von Lokiarchaeum ossiferum[30] lange „Tentakel“, in denen Partnermikroben (Bakterien) nisten und die ihm womöglich als „Protomitochondrien“ verbesserte Überlebenschancen bei steigendem Sauerstoff ermöglichen. Nach dem E3-Modell der Eukaryogenese konnten so Bakterien als Vorfahren der Mitochondrien umschlossen und endogenisiert wurden.[31][32][33][34]
Virale Eukaryogenese
[Bearbeiten | Quelltext bearbeiten]Virale Eukaryogenese ist die Bezeichnung einer Hypothese, nach der sich der Zellkern eukaryotischer Lebensformen aus einem großen DNA-Virus in im Zuge einer Endosymbiose in einem methanogenen Archaeon oder einem Bakterium entwickelt hat. Die Hypothese wurde mit dieser Bezeichnung erstmal von Philip John Livingstone Bell 2001 vorgeschlagen[35] Um 2005/2006 schlugen Forscher um Jean-Michel Claverie und Patrick Forterre darüber hinausgehend vor, dass der Übergang von RNA- zu DNA-Genomen zuerst in der Welt der Viren (Virosphäre) stattgefunden hat.[36][37][38] Im Einklang mit der RNA-Welt-Hypothese hätten zelluläre Organismen danach zunächst als sog. Ribozyten (oder Ribozellen) ihre Erbinformation in einem RNA-Genom gespeichert. Ein DNA-basiertes Virus könnte dann von einem Ribozyten-Wirt dazu befähigt haben, seine Erbinformation künftig in der Form eines DNA-Genoms zu speichern, so wie dies heute bei allen bekannten zellulären Organismen der Fall ist.[39] Damit könnten Bakterien und Archaeen sich unabhängig voneinander aus Ribozellen entwickelt haben.[40] In der Originalarbeit war allerdings auch von einer Ribozelle als Vorfahr der Eukaryoten vermutet, wenn auch eine komplexere, fähig zur RNA-Verarbeitung. Da sie eine Entstehung der Eukaryoten unabhängig von den Archaeen postulierte, war sie noch im Einklang mit ursprünglichen Überlegungen von Carl Woese und George Fox aus dem Jahr 1977 (Urkaryoten).[19]
Aber auch in dem in neuerer Zeit favorisierten Szenario, bei dem die Eukaryoten aus einer Gruppe der Asgard-Archaeen hervorgingen (Eozyten-Hypothese) könnten Viren einen entscheidenden Beitrag zur Eukaryogenese beigetragen zu haben (englisch out of virus hypothesis). Beispielsweise wurde vorgeschlagen, dass neben dem Zellkern auch die Telomerase und Telomere als Schlüsselaspekte der eukaryotischen Zellreplikation, viralen Ursprungs sind. Der virale Ursprung des modernen eukaryotischen Zellkerns könnte auf einer mehrfachen Infektion von Archaeen-Zellen, die bereits Bakterien als Mitochondrien-Vorläufer aufgenommen hatten, mit lysogenen Viren beruhen.[41] Um diese Hypothese zu belegen und den genauen Vorgang zu erforschen, sind allerdings noch weitere Studien an den Viren der Asgard-Archaeen nötig (Stand Mitte 2023).
Ein Schlüssel zur Erforschung der Eukaryogenese konnte im Entwurf eines Stammbaums Chromatins und der eukaryotischen, archaealen und viralen[42] Histone liegen.[43]
Syntrophie-Hypothese
[Bearbeiten | Quelltext bearbeiten]Die Syntrophie-Hypothese (zuerst vorgeschlagen von Purificación (Puri) López-García und David Moreira um das Jahr 2000), besagt, dass Eukaryoten durch die Kombination der Stoffwechselfähigkeiten eines Archaeons, eines fermentierenden Deltaproteobakteriums und eines methanotrophen Alphaproteobakteriums entstanden sind, wobei sich aus letzterem Mitochondrium entwickelte. Im April 2020 aktualisierte und konkretisierte dieses Team seinen Vorschlag auf ein Wasserstoff-produzierendes Asgard-Archäon und ein Schwefel-oxidierendes Deltaproteobakterium. Der dritte Organismus, das Alphaproteobakterium, soll in der Lage gewesen sein, sowohl aerob als auch anaerob zu atmen und Schwefel zu oxidieren. Dieses entwickelte sich wieder zum Mitochondrium, aber es war möglicherweise auch zur Photosynthese fähig.[44]
Fossiler Beleg
[Bearbeiten | Quelltext bearbeiten]Der LECA begründet die Kronengruppe der Eukaryoten (engl. crown eukaryotes, crown eukaryota). Zu ihnen gehören die Vorfahren von Tieren, Pilzen, Pflanzen und einer Vielzahl von Einzellern (Protisten) mit den neuen Fähigkeiten und der Komplexität der eukaryotischen Zelle.[45][46] Einzeller ohne Zellwände sind jedoch zerbrechlich und haben daher eine geringe Wahrscheinlichkeit, fossilisiert zu werden. Selbst wenn sie fossilisiert werden, weisen sie nur wenige Merkmale auf, die sie eindeutig von Prokaryoten unterscheiden: Größe, morphologische Komplexität und (schließlich) Vielzelligkeit (Zellkolonien). Zu den frühen Eukaryotenfossilien aus dem späten Paläoproterozoikum gehören Acritarcha-Mikrofossilien mit relativ robusten, verschnörkelten Kohlenstoffbläschen wie die Gattung Tappania vor 1,63 Milliarden Jahren oder Shuiyousphaeridium vor 1,8 Milliarden Jahren.[46] Etwas jünger sind die in einer ukrainischen Quarzmine bei Schytomyr gefundenen mutmaßlich eukaryotischen Volyn-Biota, sie sind etwa 1,5 Milliarden Jahre alt.[47]
Weblinks
[Bearbeiten | Quelltext bearbeiten]- Axel Brennicke: Archaea und Eukaryoten sind miteinander verwandt. Auf: spektrum.de vom 1. Oktober 1994.
- Jochen J. Brocks, Benjamin J. Nettersheim, Pierre Adam, Philippe Schaeffer, Amber J. M. Jarrett, Nur Güneli, Tharika Liyanage, Lennart M. van Maldegem, Christian Hallmann, Janet M. Hope: Lost world of complex life and the late rise of the eukaryotic crown. In: Nature, Band 618, Nr. 7966, S. 767–773, Juni 2023; doi:10.1038/s41586-023-06170-w, PMID 3728661, Epub 7. Juni 2023 (englisch). Protosterol-Biota. Dazu:
- Scientists discover ‘lost world’ of early ancestors in billion-year-old rocks. Auf: EurekAlert! vom 7. Juni 2023
- Remains of an Extinct World of Primordial Organisms Discovered. Auf: SciTechDaily vom 15. Juni 2023
- Enrico de Lazaro: Molecular Remains of Ancient Complex Life Found in 1.6-Billion-Year-Old Rocks. Auf: Sci.News vom 8. Juni 2023
- Prakash Chandra Arya, Claude Nambaje, Madhusoodhan Satish-Kumar, Sasidharan Kiran, Krishnan Sajeev: Himalayan magnesite records abrupt cyanobacterial growth that plausibly triggered the Neoproterozoic Oxygenation Event. In: Precambrian Research, Band 395, 1. September 2023, S. 107129; doi:10.1016/j.precamres.2023.107129, ResearchGate, Epub 17. Juli 2023 (englisch). Dazu:
- Chadlin M. Ostrander, Christian J. Bjerrum, Anne-Sofie C. Ahm, Simon R. Stenger, Kristin D. Bergmann, Mohamed A. K. El-Ghali, Abdul R. Harthi, Zayana Aisri, Sune G. Nielsen: Widespread seafloor anoxia during generation of the Ediacaran Shuram carbon isotope excursion. In: gebiology, 8. Mai 2023, doi:10.1111/gbi.12557 (englisch). Dazu:
- Avalon Explosion: Life on Earth Didn’t Arise As Described in Textbooks. Auf: SciTechDaily vom 29. Juli 2023. Die Avalon-Explosion als Vorgänger der Kambrischen Explosion der Artenvielfalt.
Einzelnachweise
[Bearbeiten | Quelltext bearbeiten]- ↑ a b c Casey McGrath: Highlight: Unraveling the Origins of LUCA and LECA on the Tree of Life. In: Genome Biology and Evolution. 14. Jahrgang, Nr. 6, 31. Mai 2022, doi:10.1093/gbe/evac072 (englisch).
- ↑ Madeline C. Weiss, Filipa L. Sousa, Natalia Mrnjavac, Sinje Neukirchen, Mayo Roettger, Shijulal Nelson-Sathi, William F. Martin: The physiology and habitat of the last universal common ancestor. In: Nature Microbiology. 1. Jahrgang, Nr. 9, 25. Juli 2016, S. 16116, doi:10.1038/nmicrobiol.2016.116, PMID 27562259 (englisch, amazonaws.com [PDF]).
- ↑ Toni Gabaldón: Origin and Early Evolution of the Eukaryotic Cell. In: Annual Review of Microbiology. 75. Jahrgang, Nr. 1, Oktober 2021, S. 631–647, doi:10.1146/annurev-micro-090817-062213, PMID 34343017 (englisch).
- ↑ Carl R. Woese, Otto Kandler, Mark L. Wheelis: Towards a natural system of organisms: proposal for the domains Archaea, Bacteria, and Eucarya. In: Proceedings of the National Academy of Sciences of the United States of America. 87. Jahrgang, Nr. 12, Juni 1990, S. 4576–4579, doi:10.1073/pnas.87.12.4576, PMID 2112744, PMC 54159 (freier Volltext), bibcode:1990PNAS...87.4576W (englisch).
- ↑ a b c
Laura Eme, Daniel Tamarit, Eva F. Cáceres, Courtney W. Stairs, Valerie De Anda, Max E. Schön, Kiley W. Seitz, Nina Dombrowski, William H. Lewis, Felix Homa, Jimmy H. Saw, Jonathan Lombard, Takuro Nunoura, Wen-Jun Li, Zheng-Shuang Hua, Lin-Xing Chen, Jillian F. Banfield, Emily St. John, Anna-Louise Reysenbach, Matthew B. Stott, Andreas Schramm, Kasper U. Kjeldsen, Andreas P. Teske, Brett J. Baker, Thijs J. G. Ettema: Inference and reconstruction of the heimdallarchaeial ancestry of eukaryotes. In: Nature, Band 618, S. 992–999; doi:10.1038/s41586-023-06186-2. Dazu:
- Nadja Podbregar: Wir sind alle „Asgardianer“ – Eukaryoten entstanden aus einer Untergruppe der Asgard-Archaeen. Auf: scinexx.de vom 26. Juni 2023.
- Asgard Archaea and All Eukaryotes Share Common Ancestor, Study Says. Auf Sci.News vom 26. Juni 2023.
- ”We’re All Asgardians”: Scientists Discover New Clues About the Origin of Complex Life. Auf: SciTechDaily vom 28. Juli 2023.
- Mysterious Microorganisms Unveiled as Key to the Origin of Complex Life. Auf: SciTechDaily vom 6. August 2023.
- ↑ a b c Amparo Latorre, Ana Durbán, Andrés Moya, Juli Peretó: Origins and Evolution of Life: An astrobiological perspective. Hrsg.: Muriel Gargaud, Purificacion López-Garcìa, Hervé Martin. Cambridge University Press, Cambridge 2011, ISBN 978-0-521-76131-4, Part V: Mechanisms for life evolution, Chapter 21: The role of symbiosis in eukaryotic evolution, S. 326–339, doi:10.1017/CBO9780511933875.023 (englisch, google.com). Buch: doi:10.1017/CBO9780511933875.
- ↑
Tara A. Mahendrarajah, Edmund R. R. Moody, Dominik Schrempf, Lénárd L. Szánthó, Nina Dombrowski, Adrián A. Davín, Davide Pisani, Philip C. J. Donoghue, Gergely J. Szöllősi, Tom A. Williams, Anja Spang: ATP synthase evolution on a cross-braced dated tree of life. In: Nature Communications, Band 14, Nr. 7456; doi:10.1038/s41467-023-42924-w (englisch). Dazu:
- Tara A. Mahendrarajah, Anja Spang: Looking for ‘LUCA’ and the timing of cellular evolution. Auf: Royal Netherlands Institute for Sea Research (NIOZ) vom 21. November 2023; Looking for ‘LUCA’ and the timing of cellular evolution. Auf: EurekAlert vom 21. November 2023.
- Anna Manz: Wann lebte der Urahn allen Lebens? Auf: scinexx.de vom 29. November 2023.
- ↑ Anthony M. Poole, David Penny: Evaluating hypotheses for the origin of eukaryotes. In: BioEssays, Band 29, Nr. 1, Januar 2007, S. 74–84, ISSN 0265-9247; doi:10.1002/bies.20516 (englisch).
- ↑ Victor V. Emelyanov: Mitochondrial connection to the origin of the eukaryotic cell. In: European Journal of Biochemistry, Band 270, Nr. 8, April 2003, S. 1599–1618; doi:10.1046/j.1432-1033.2003.03499.x, PMID 12694174 (englisch).
- ↑
William F. Martin, Miklós Müller: The hydrogen hypothesis for the first eukaryote. In: Nature. Band 392, März 1998, S. 37–41, doi:10.1038/32096, PMID 9510246 (englisch). Dazu:
- W. Ford Doolittle: A paradigm gets shifty. In: Nature. Band 392, 5. März 1998, S. 15–16, doi:10.1038/32033, PMID 9510239 (englisch).
- ↑ David Moreira, Purificación López-García: Symbiosis Between Methanogenic Archaea and δ-Proteobacteria as the Origin of Eukaryotes: The Syntrophic Hypothesis. In: Journal of Molecular Evolution, Band 47, Nr. 5, November 1998, S. 517–30, ISSN 0022-2844; doi:10.1007/pl00006408, PMID 9797402.
- ↑ Tom A. Williams, Cymon J. Cox, Peter G. Foster, Gergely J. Szöllősi& T. Martin Embley: Phylogenomics provides robust support for a two-domains tree of life. In: Nature Ecology & Evolution. 4. Jahrgang, Nr. 1, 9. Dezember 2019, S. 138–147, doi:10.1038/s41559-019-1040-x, PMID 31819234, PMC 6942926 (freier Volltext) – (englisch).
- ↑ a b Lynn Margulis, Michael Chapman, Ricardo Guerrero, John Hall: The last eukaryotic common ancestor (LECA): Acquisition of cytoskeletal motility from aerotolerant spirochetes in the Proterozoic Eon. In: Proceedings of the National Academy of Sciences. 103. Jahrgang, Nr. 35, 29. August 2006, S. 13080–13085, doi:10.1073/pnas.0604985103, PMID 16938841, PMC 1559756 (freier Volltext), bibcode:2006PNAS..10313080M (englisch).
- ↑ C. J. Cox, P. G. Foster, R. P. Hirt, S. R. Harris, T. M. Embley: The archaebacterial origin of eukaryotes. In: Proc Natl Acad Sci USA. 105. Jahrgang, Nr. 51, 2008, S. 20356–20361, doi:10.1073/pnas.0810647105, PMID 19073919, PMC 2629343 (freier Volltext), bibcode:2008PNAS..10520356C (englisch).
- ↑ Eugene V. Koonin: The origin and early evolution of eukaryotes in the light of phylogenomics, in: Genome Biology. Band 11, Nr. 209, 5. Mai 2010; doi:10.1186/gb-2010-11-5-209, PMID 20441612, PMC 2898073 (freier Volltext).
- ↑ Maureen A. O’Malley, Michelle M. Leger, Jeremy G. Wideman, Iñaki Ruiz-Trillo: Concepts of the last eukaryotic common ancestor. In: Nature Ecology & Evolution. 3. Jahrgang, Nr. 3, 18. Februar 2019, S. 338–344, doi:10.1038/s41559-019-0796-3, PMID 30778187 (englisch). hdl:10261/201794.
- ↑ a b Brian S. Leander: Predatory protists. In: Current Biology. 30. Jahrgang, Nr. 10, Mai 2020, S. R510–R516, doi:10.1016/j.cub.2020.03.052, PMID 32428491 (englisch).
- ↑ a b Jürgen F. H. Strassert, Iker Irisarri, Tom A. Williams, Fabien Burki: A molecular timescale for eukaryote evolution with implications for the origin of red algal-derived plastids. In: Nature Communications. 12. Jahrgang, Nr. 1, 25. März 2021, S. 1879, doi:10.1038/s41467-021-22044-z, PMID 33767194, PMC 7994803 (freier Volltext), bibcode:2021NatCo..12.1879S (englisch).
- ↑ a b Carl R. Woese, George E. Fox: Phylogenetic structure of the prokaryotic domain: the primary kingdoms. In: Proceedings of the National Academy of Sciences. Band 74, Nummer 11, November 1977, S. 5088–5090; doi:10.1073/pnas.74.11.5088, PMID 270744, PMC 432104 (freier Volltext).
- ↑ Tom Cavalier-Smith: Archaebacteria and Archezoa. In: Nature. 339. Jahrgang, Nr. 6220, Mai 1989, S. 100–101, doi:10.1038/339100a0, PMID 2497352 (englisch).
- ↑ Patrick J. Keeling: A kingdom’s progress: Archezoa and the origin of eukaryotes. In: BioEssays. Band 20, Nummer 1, Januar 1998, S. 87–95; doi:10.1002/(SICI)1521-1878(199801)20:1<87::AID-BIES12>3.0.CO;2-4 (alternativer Volltextzugriff: SemanticScholar).
- ↑ Brigitte Boxma, Rob M. de Graaf, Georg W. M. van der Staay, Theo A. van Alen, Guenola Ricard, Toni Gabaldon, Angela H. A. M. van Hoek, Seung Yeo Moon-van der Staay, Werner J. H. Koopman, Jaap J. van Hellemond, Aloysius G. M. Tielens, Thorsten Friedrich, Marten Veenhuis, Martijn A. Huynen, Johannes H. P. Hackstein: An anaerobic mitochondrion that produces hydrogen. In: Nature. Band 434, Nr. 7029, 3. Februar 2005, S. 74–79, doi:10.1038/nature03343.
- ↑ Anna Akhmanova, Frank Voncken, Theo van Alen, Angela van Hoek, Brigitte Boxma, Godfried Vogels, Marten Veenhuis, Johannes H. P. Hackstein: A hydrogenosome with a genome. In: Nature. Band 396, Nr. 6711, 10. Dezember 1998, S. 527–528, doi:10.1038/25023 (englisch).
- ↑ Nico Bremer, Fernando D. K. Tria, Josip Skejo, Sriram G. Garg, William F. Martin: Ancestral State Reconstructions Trace Mitochondria But Not Phagocytosis to the Last Eukaryotic Common Ancestor. In: Genome Biology and Evolution. 14. Jahrgang, Nr. 6, 31. Mai 2022, doi:10.1093/gbe/evac079, PMID 35642316, PMC 9185374 (freier Volltext) – (englisch).
- ↑ a b c d V. Lila Koumandou, Bill Wickstead, Michael L. Ginger, Mark van der Giezen, Joel B. Dacks, Mark C. Field: Molecular paleontology and complexity in the last eukaryotic common ancestor. In: Critical Reviews in Biochemistry and Molecular Biology. 48. Jahrgang, Nr. 4, 2013, S. 373–396, doi:10.3109/10409238.2013.821444, PMID 23895660, PMC 3791482 (freier Volltext) – (englisch).
- ↑ Kira S. Makarova, Natalya Yutin, Stephen D. Bell, Eugene V. Koonin: Evolution of diverse cell division and vesicle formation systems in Archaea. In: Nature Reviews Microbiology. 8. Jahrgang, Nr. 10, 6. September 2010, S. 731–741, doi:10.1038/nrmicro2406, PMID 20818414, PMC 3293450 (freier Volltext) – (englisch).
- ↑ Nick Lane, William F. Martin: The energetics of genome complexity. In: Nature. 467. Jahrgang, Nr. 7318, 20. Oktober 2010, S. 929–934, doi:10.1038/nature09486, PMID 20962839, bibcode:2010Natur.467..929L (englisch).
- ↑ Eugene V. Koonin: The incredible expanding ancestor of eukaryotes. In: Cell. 140. Jahrgang, Nr. 5, März 2005, S. 606–608, doi:10.1016/j.cell.2010.02.022, PMID 20211127, PMC 3293451 (freier Volltext) – (englisch).
- ↑ Joran Martijn, Thijs J. G. Ettema: From archaeon to eukaryote: the evolutionary dark ages of the eukaryotic cell. In: Biochemical Society Transactions. 41. Jahrgang, Nr. 1, Februar 2013, S. 451–7, doi:10.1042/BST20120292, PMID 23356327 (englisch, biochemsoctrans.org).
- ↑ Elizabeth Pennisi: Strange, tentacled microbe may resemble ancestor of complex life. In: Science, 21. Dezember 2022; Nadja Podbregar: Bindeglied der frühen Evolution kultiviert, scinexx.de vom 22. Dezember 2022. Quelle: doi:10.1038/s41586-022-05550-y Actin cytoskeleton and complex cell architecture in an Asgard archaeon.
- ↑ Carl Zimmer: Under the Sea, a Missing Link in the Evolution of Complex Cells. In: The New York Times. 6. Mai 2015, ISSN 0362-4331 (nytimes.com).
- ↑
Hiroyuki Imachi, Masaru K. Nobu, Nozomi Nakahara, Yuki Morono, Miyuki Ogawara, Yoshihiro Takaki, Yoshinori Takano, Katsuyuki Uematsu, Tetsuro Ikuta, Motoo Ito, Yohei Matsui, Masayuki Miyazaki, Kazuyoshi Murata, Yumi Saito, Sanae Sakai, Chihong Song, Eiji Tasumi, Yuko Yamanaka, Takashi Yamaguchi, Yoichi Kamagata, Hideyuki Tamaki, Ken Takai: Isolation of an archaeon at the prokaryote–eukaryote interface. In: Nature. Band 577, Nr. 7791, 23. Januar 2020, ISSN 1476-4687, S. 519–525, doi:10.1038/s41586-019-1916-6, PMID 31942073, PMC 7015854 (freier Volltext), bibcode:2020Natur.577..519I (englisch, nature.com). Dazu:
- Yasemin Saplakoglu: Out of Deep-Sea Mud, a Strange Blob May Hold Secrets to the Origins of Complex Life. Auf: LiveScience vom 17. Januar 2020.
- ↑ Jonathan Lambert: Scientists glimpse oddball microbe that could help explain rise of complex life. In: Nature. Band 572, 2019, S. 294 (nature.com).
- ↑
Daniel B. Mills, Richard A. Boyle, Stuart J. Daines, Erik A. Sperling, Davide Pisani, Philip C. J. Donoghue, Timothy M. Lenton: Eukaryogenesis and oxygen in Earth history. In: Nature Ecology & Evolution, 21. April 2022; doi:10.1038/s41559-022-01733-y. Dazu:
- Neue Studie legt nahe, dass eukaryotische Zellen in anoxischer Umgebung entstanden sind. Auf: Aus Der Welt (Max) vom 27. April 2022.
- Eukaryotic Cells Emerged in Anoxic Environment, New Study Suggests. Auf: sci-news vom 27. April 2022. Im Bild: Prometheoarchaeum syntrophicum MK-D1.
- ↑ Philip John Livingstone Bell: Viral eukaryogenesis: was the ancestor of the nucleus a complex DNA virus? In: Journal of Molecular Evolution. 53. Jahrgang, Nr. 3, September 2001, S. 251–6, doi:10.1007/s002390010215, PMID 11523012, bibcode:2001JMolE..53..251L (englisch).
- ↑ Patrick Forterre: The two ages of the RNA world, and the transition to the DNA world: a story of viruses and cells. In: Biochimie, Band 87, Nr. 9–10, September–Oktober 2005, S. 793–803; doi:10.1016/j.biochi.2005.03.015, PMID 16164990.
- ↑ Patrick Forterre: Three RNA cells for ribosomal lineages and three DNA viruses to replicate their genomes: a hypothesis for the origin of cellular domain. In: Proceedings of the National Academy of Sciences of the United States of America. 103. Jahrgang, Nr. 10, März 2006, S. 3669–74, doi:10.1073/pnas.0510333103, PMID 16505372, PMC 1450140 (freier Volltext), bibcode:2006PNAS..103.3669F, JSTOR:30048645 (englisch).
- ↑ Patrick Forterre: Evolution – Die wahre Natur der Viren. In: Spektrum der Wissenschaft. Heft 8/2017, S. 37 (Online-Artikel veröffentlicht am 19. Juli 2017).
- ↑ Jean-Michel Claverie: Viruses take center stage in cellular evolution. In: Genome Biology. 7. Jahrgang, Nr. 6, 16. Juni 2006, S. 110, doi:10.1186/gb-2006-7-6-110, PMID 16787527, PMC 1779534 (freier Volltext) – (englisch).
- ↑
Nick Lane: Der Funke des Lebens: Energie und Evolution, Konrad Theiss Verlag, Stuttgart 2017, ISBN 978-3-8062-3484-8. Originaltitel: The Vital Question: Why Is Life The Way It Is?, Profile Books, 2015; PDF ( vom 10. September 2017 im Internet Archive). Übersetzt von Martina Wiese, Monika Niehaus, Jorunn Wissmann. Dazu:
- Björn Lohmann: Aus energetischen Zwängen entstanden. spektrum.de, 24. Juni 2017, abgerufen am 8. Dezember 2022.
- Christmas 2015: The 6 best books in science. 27. November 2015, abgerufen am 8. Dezember 2022 (englisch).
- ↑ Guenther Witzany: The viral origins of telomeres and telomerases and their important role in eukaryogenesis and genome maintenance. In: Biosemiotics. 1. Jahrgang, Nr. 2, 23. Juli 2008, S. 191–206, doi:10.1007/s12304-008-9018-0 (englisch, mitdenker.at [PDF; abgerufen am 28. Juli 2023]). Info: Der Archivlink wurde automatisch eingesetzt und noch nicht geprüft. Bitte prüfe Original- und Archivlink gemäß Anleitung und entferne dann diesen Hinweis.
- ↑ Paul B. Talbert, Karim-Jean Armache, Steven Henikoff: Viral histones: pickpocket’s prize or primordial progenitor? In: BMC Epigenetics & Chromatin, Band 15, Nr. 21, 28. Mai 2022; doi:10.1186/s13072-022-00454-7, PMID 35624484, PMC 9145170 (freier Volltext).
- ↑
Xavier Grau-Bové, Cristina Navarrete, Cristina Chiva, Thomas Pribasnig, Meritxell Antó, Guifré Torruella, Luis Javier Galindo, Bernd Franz Lang, David Moreira, Purificación López-Garcia, Iñaki Ruiz-Trillo, Christa Schleper, Eduard Sabidó, Arnau Sebé-Pedrós: A phylogenetic and proteomic reconstruction of eukaryotic chromatin evolution. In: Nature Ecology & Evolution, Band 6, S. 1007–1023, 9. Juni 2022; doi:10.1038/s41559-022-01771-6, PMID 35680998 PMC 7613034 (freier Volltext). Dazu:
- Fig. 1: Diversity of post-translational modifications in eukaryotic canonical and variant histones.
- Fig. 3: Taxonomic distribution of chromatin-associated gene classes.
- Fig. 6: Chromatin evolution and eukaryogenesis.
- Xavier Grau-Bové: Repository: Chromatin evolution. Auf: GitHub.
- Chromatin First Evolved in Ancient Microbes 1-2 Billion Years Ago, New Research Suggests. Auf: sci-news vom 13. Juni 2022.
- Chromatin originated in ancient microbes one to two billion years ago. Genomic and proteomic analysis reveals that the regulatory role of chromatin is a eukaryotic innovation. Auf: ScienceDaily vom 9. Juni 2022. Quelle: Center for Genomic Regulation (CRG)
- Chromatin originated in ancient microbes one to two billion years ago. Center for Genomic Regulation (CRG) vom 9. Juni 2022.
- ↑
Purificación López-García, David Moreira: The Syntrophy hypothesis for the origin of eukaryotes revisited. In: Nature Microbiology. 5. Jahrgang, Nr. 5, 27. April 2020, ISSN 2058-5276, S. 655–667, doi:10.1038/s41564-020-0710-4, PMID 32341569 (englisch). Dazu:
- Puri López-García: Digging into the syntrophic origin of eukaryotes. In: Springer Nature, Behind the Paper, 27. April 2020
- ↑ Yves Van de Peer, Sandra L. Baldaufrid, W. Ford Doolittle, Axel Meyerid: An Updated and Comprehensive rRNA Phylogeny of (Crown) Eukaryotes Based on Rate-Calibrated Evolutionary Distances. In: Journal of Molecular Evolution. 51. Jahrgang, Nr. 6, 2000, S. 565–576, doi:10.1007/s002390010120, PMID 11116330, bibcode:2000JMolE..51..565V (englisch, uni-konstanz.de).
- ↑ a b Nicholas J. Butterfield: Early evolution of the Eukaryota. In: Palaeontology. 58. Jahrgang, Nr. 1, 2015, S. 5–17, doi:10.1111/pala.12139, bibcode:2015Palgy..58....5B (englisch).
- ↑
Gerhard Franz, Vladimir Khomenko, Peter Lyckberg, Vsevolod Chournousenko, Ulrich Struck, Ulrich Gernert, Jörg Nissen: The Volyn biota (Ukraine) – indications of 1.5 Gyr old eukaryotes in 3D preservation, a spotlight on the “boring billion”. In: Biogeosciences, Band 20, Nr. 10, S. 1901–1924, 24. Mai 2023. Dazu:
- Jan Dönges: Gesteine: Milliarden Jahre alte Mikroorganismen in 3-D erhalten. Auf: spektrum.de vom 24. Juli 2023.
- Nadja Podbregar: Älteste echte Mikrofossilien entdeckt. Auf: scinexx.de vom 25. Juli 2023.