Chloroplast

| Übergeordnet |
|---|
| Organell Plastid |
| Untergeordnet |
| Membran Intermembranraum Thylakoid |
| Gene Ontology |
| QuickGO |
Die Chloroplasten (von altgriechisch χλωρός chlōrós „grün“ und πλαστός plastós „geformt“) sind Organellen der Zellen von Grünalgen und Landpflanzen, die Photosynthese betreiben. Bei höheren Pflanzen können aus den photosynthetisch aktiven Chloroplasten durch Differenzierung Chromoplasten, Leukoplasten (Amyloplasten, Elaioplasten), Etioplasten und Gerontoplasten entstehen (zusammengefasst als Plastiden).
Aufbau von Chloroplasten
[Bearbeiten | Quelltext bearbeiten]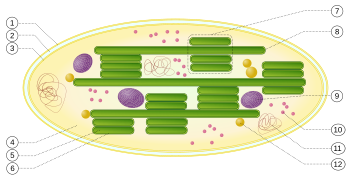
1: äußere Hüllmembran
2: Intermembranraum
3: innere Hüllmembran (1+2+3: Hülle)
4: Stroma (Matrix)
5: Thylakoidlumen (im Inneren des Thylakoids)
6: Thylakoidmembran
7: Granum (Granalamelle)
8: Thylakoid (Stromalamelle)
9: Stärkekörper
10: plastidäres Ribosom (Plastoribosom)
11: plastidäre DNA (cpDNA syn. ctDNA)
12: Plastoglobulus (kugelförmige Struktur aus Lipiden; pl.: Plastoglobuli)
Die Thylakoidmembranen liegen entweder in gestapelter (7) oder in ungestapelter (8) Form vor.

A: Der Proplastid aus unbelichteten Pflanzen besteht lediglich aus einer Innen- und einer Außenmembran
B: Licht induziert die Synthese von Chlorophyll, Phospholipiden und Thylakoid-Proteinen. Von der Innenmembran schnüren sich Vesikel in den Matrixraum ab
C: Während der Vergrößerung der Proplastiden bilden sich einzelne Thylakoide durch Fusion von Vesikeln. Durch ein Protein der Lichtsammelkomplexe kommt es dann zur Verklebung (Stapelung) von Thylakoiden.
D: Durch Ausbau der Membransysteme, wobei die Grana-Thylakoidstapel durch (ungestapelte) Stroma-Thylakoide miteinander verbunden werden, entsteht der Chloroplast
Die Chloroplasten der Landpflanzen haben Durchmesser von etwa 4 bis 8 µm. Bei vielen Algen mit nur einem einzigen Chloroplasten pro Zelle kann dieser jedoch einen großen Teil der Zelle einnehmen.
Der strukturelle Aufbau der Chloroplasten gleicht dem der Cyanobakterien (Blaugrünbakterien, früher Blaualgen genannt). Chloroplasten besitzen (fast immer) eine eigene, plastidäre, DNA (Chloroplasten-DNA, abgekürzt cpDNA oder ctDNA) zusammen mit eigenen Ribosomen (plastidären Ribosomen oder Plastoribosomen) und ähneln darin Mitochondrien.[1][2] Das Genom der Chloroplasten und der anderen Plastiden wird auch als Plastom bezeichnet.
Chloroplasten sind umhüllt von zwei Biomembranen, in deren Inneren sich das Stroma als plasmatische Phase befindet. Das Stroma wiederum wird von Thylakoidmembranen durchzogen, Abkömmlingen der Innenmembran. Mit Ausnahme vieler phototropher Protisten sind in den Chloroplasten der höheren Phototrophen an etlichen Stellen flache, runde Ausstülpungen dieser Membranen „geldrollenartig“ übereinandergelagert – einen solchen Thylakoidstapel nennt man Granum (pl. Grana). In den Membranen der Thylakoide sind verschiedene Pigmente eingelagert, vor allem der grüne Farbstoff Chlorophyll. Besonders viel davon findet sich in den Membranen der Grana, weshalb diese intensiv grün gefärbt erscheinen.
Die Pigmente können Licht bestimmter Wellenlängen absorbieren und die aufgenommene Energie wird zur Produktion von ATP aus ADP und Phosphat genutzt (siehe Phototrophie). ATP dient als Energieüberträger zum Aufbau von Glucose bzw. Stärke aus CO2 und Wasser.
Aufbau eines chemiosmotischen Membranpotenzials
[Bearbeiten | Quelltext bearbeiten]Die Biogenese dieser drei Membransysteme erklärt die Tatsache, dass das Membranpotenzial durch einen Protonengradienten bei Chloroplasten über die Thylakoidmembran aufgebaut wird (das Thylakoid-Innere weist ein saures Milieu auf), während bei Mitochondrien der Intermembranraum (Bereich zwischen Innen- und Außenmembran) chemiosmotisch mit H+-Ionen beladen wird. Analog ist die ATP-Synthase (alias FoF1-ATPase) bei Chloroplasten ein in die Thylakoidmembran eingebettetes Enzym (CF1-Teil ragt ins Stroma), bei Mitochondrien ein Bestandteil der Innenmembran (F1-Teil der Matrix zugewandt). Bei beiden Systemen wird ATP also an die Matrix/das Stroma abgegeben. Im Gegentausch zu ADP kann es in das Cytosol der Zelle gelangen.
Plastoskelett
[Bearbeiten | Quelltext bearbeiten]Heute weiß man, dass auch Bakterien ein Zytoskelett besitzen, dessen Proteine evolutionäre Verwandtschaft zu denen des eukaryotischen Zytoskeletts zeigen. Aus Versuchen am Laubmoos Physcomitrella patens (unter anderem mit Knockout-Moosen) ist bekannt, dass die FtsZ-Proteine, die Tubulin-Homologen, nicht nur die Teilung der Chloroplasten bewirken,[3] sondern auch ein komplexes Netzwerk in den Chloroplasten ausbilden können. Da diese Netzwerke stark an das Zytoskelett erinnern, prägte Ralf Reski 2000 den Begriff „Plastoskelett“ für diese komplexen Strukturen und postulierte, dass sie in den Plastiden ähnliche Funktionen erfüllen wie das Zytoskelett für die gesamte Zelle.[4][5]
Chloroplasten-DNA
[Bearbeiten | Quelltext bearbeiten]Im Jahr 1962 wurde erstmals Chloroplasten-DNA nachgewiesen,[6] 1986 wurde erstmals ein Plastom sequenziert.[7] Seitdem wurden Hunderte von Chloroplasten-DNAs aus verschiedenen Spezies sequenziert. Meist handelt es sich dabei jedoch um Chloroplastida, d. h. Landpflanzen oder Grünalgen. Glaucophyten (Glaucophyta), Rotalgen (Rhodophyta syn. Rhodophyceae) und andere Algengruppen sind stark unterrepräsentiert.[8] Die DNA der Chloroplasten ist – zumindest bei den Chloroplastida (Grünalgen und Landpflanzen) – gewöhnlich wie bei den meisten Bakterien ringförmig strukturiert. Das Chloroplasten-Genom (Plastom) weist Abschnitte auf, die als gegenläufige Kopien (englisch inverted repeats) vorliegen, ähnlich wie im Genom von Cyanobakterien. Bei Mitochondrien und Plastiden wie Chloroplasten ist die DNA wie bei Bakterien normalerweise in Nucleoiden (Kernäquivalenten) verdichtet. Dafür sorgen so genannte histonähnliche Proteine (HLPs, nach englisch histone like proteins). Diese sind untereinander homolog, zu den echten Histonen im Zellkern der eukaryotischen Zellen (Euzyten) aber nur funktionell ähnlich (analog). Die Protein-codierenden Gene werden in Messenger-RNA transkribiert, die als Vorlage für die Proteinsynthese (Translation) an den eigenen Ribosomen (Plastoribosomen) dient. Diese plastidären Ribosomen ähneln in Aufbau und Struktur denen der Bakterien und Mitochondrien, sind aber signifikant kleiner und einfacher aufgebaut als die der sie umgebenden Euzyte.
Herkunft der Chloroplasten – Endosymbiontentheorie
[Bearbeiten | Quelltext bearbeiten]Der deutsche Botaniker Andreas Franz Wilhelm Schimper hatte bereits 1883 festgestellt, dass Chloroplasten den Cyanobakterien sehr ähnlich sind und damit bereits eine symbiotische Entstehung der Chloroplasten angedeutet, so dass er als Wegbereiter der später formulierten Endosymbiontentheorie gelten darf.[9] Ähnliche Ideen vertraten zu dieser Zeit Sachs und Altmann.[10] Diese Idee hatte der russische Biologe Konstantin Sergejewitsch Mereschkowski 1905[11] und Ivan Wallin 1922[12][13] aufgegriffen und weiter konkretisiert.

Lange Zeit war unbekannt, wie Chloroplasten sich teilen und ihre Form verändern. Da es nicht gelang isolierte Chloroplasten in vitro (im Labor auf Nährmedien) zu kultivieren, konnte sich die Theorie daher erst Anfang der 1970er Jahre etablieren (Lynn Margulis), als in beiden Organelltypen DNA nachgewiesen wurde.[14][15] Die eigenständige „Vermehrung“ der Chloroplasten ähnlich wie bei Bakterien, ohne eine strukturelle Koppelung an die Zellteilung der umgebenden Eukaryotenzelle, gilt als ein weiteres Argument für die Endosymbiontentheorie.
Primäre Endosymbiose
[Bearbeiten | Quelltext bearbeiten]
Ein Eukaryot mit Mitochondrien umschloss einst ein Cyanobakterium. Aus diesem entwickelten sich im Lauf der Evolution dann Chloroplasten, so dass dieses Ereignis den Ausgangspunkt einer Abstammungslinie von Zellen mit beiden Organellen bildete.[16][17] Man beachte, dass das zum Endosymbiont werdende Cyanobakterium bereits zuvor eine Doppelmembran hatte – die Membran der phagosomalen Vakuole (Nahrungsvakuole) ging in diesem Fall verloren.[18]
Die genaue Untersuchung der Chloroplasten und ihrer „Wirtszellen“ hat ergeben, dass dieser Vorgang mehrstufig (verschachtelt) abgelaufen ist (Übersicht siehe unten). Am Beginn trat vor etwa 600 Millionen bis 2 Milliarden Jahren ein frei lebendes photosynthetisierenden Cyanobakterium in eine frühe eukaryotische Zelle (Euzyte) ein,[19][20][21] entweder als Nahrung (Phagocytose) oder als innerer Parasit (Endoparasit).[16] Es konnte jedoch der phagozytischen (phagosomalen) Vakuole, in der es zunächst war, entkommen, ohne von der Euzyte aufgelöst zu werden,[22] und wurde zu einem permanenten Bewohner dieser „Wirtszelle“.
Endosymbiotischer Gentransfer
[Bearbeiten | Quelltext bearbeiten]Im Zuge der Evolution und Eingliederung des Endosymbionten kam es zu diversen Anpassungen. Hierzu zählt unter anderem die Anpassung des Chloroplastengenoms. Die Größe des Genoms sank von ca. 3,5 Mio. Basen auf 120–160 Tausend. Das entspricht einer Reduktion von oft mehr als 1500 Genen bei Cyanobakterien auf etwa 60–100 Gene in Chloroplasten.[23] Die Verkleinerung des Genoms ging mit dem Verlust genetischer Informationen und Transfers in den Zellkern einher,[6][24][25] ein Vorgang, der als ‚endosymbiotischer Gentransfer‘ (EGT) bezeichnet wird. Diese Art des Gentransfers kann auch heute noch beobachtet werden.[26][27]
Import von Proteinen in den Chloroplasten
[Bearbeiten | Quelltext bearbeiten]Gleichzeitig entwickelte sich eine komplexe Maschinerie zum Import von Proteinen aus dem Zytosol in den Chloroplasten. So findet man ca. 2000 Proteine im Chloroplasten trotz der nur knapp 100 verbliebenen Gene. Diese übrig gebliebenen Protein-codierenden Gene lassen sich grob in zwei Kategorien einteilen: Aufrechterhaltung des genetischen Apparats (DNA-Polymerase, tRNAs und rRNAs) und Aufrechterhaltung der photosynthetischen Kapazität (Photosystemkomponenten und weitere Proteine). Bisher ist nicht vollständig geklärt, wie die Synchronisation der Expression zwischen Kern und Chloroplasten erfolgt. Dies ist notwendig, da in allen Proteinkomplexen im Chloroplasten plastidär und nukleär codierte Produkte zusammengesetzt werden.
DNA-lose Chloroplasten
[Bearbeiten | Quelltext bearbeiten]Im Jahr 2014 wurde sogar in der nicht photosynthetisch aktiven Grünalge Polytomella (Chlamydomonadales, syn. Volvocales) ein Plastid ohne Genom gefunden. Dies zeigt, dass Chloroplasten durch endosymbiotischen Gentransfer ihr gesamtes Genom verlieren können.[28] Der Sachverhalt ist damit analog zu dem bei Mitochondrien, wo mit einem Verlust der ursprünglichen Funktion ebenfalls eine starke Reduktion bzw. der komplette Verlust des Genoms einhergeht (siehe Hydrogenosom und Mitosom).
Eine Genomanalyse von Sapria himalayana (einer parasitischen Pflanze aus der Familie der Rafflesiengewächse) ergab 2021, dass die Pflanze nicht nur ihre Fähigkeit zur Chlorophyllbildung verloren hat, sondern offenbar das komplette Chloroplasten-Genom (Plastom), einschließlich der Gene im Zellkern, die das Plastom regulieren.[29][30] Im Unterschied zu Polytomella sind die Plastiden-Gene hier nicht auf den Zellkern übergegangen.
Die erste primäre Endosymbiose (Hauptlinie)
[Bearbeiten | Quelltext bearbeiten]Es wurde lange diskutiert, ob so entstandene primäre Chloroplasten aus einem einzigen endosymbiotischen Ereignis oder aus mehreren unabhängigen Ereignissen in verschiedenen eukaryotischen Abstammungslinien stamme. Es wird heute allgemein angenommen, dass praktisch alle Organismen mit primären Chloroplasten einen einzigen gemeinsamen Vorfahren haben.[19][20] Das damals aufgenommene Cyanobakterium stand offenbar nahe der heutigen Spezies Gloeomargarita lithophora, diese befindet sich basal im Stammbaum der Cyanobakterien nahe der Gattung Synechococcus.[31][32][33] Die Alge Cyanophora, ein Glaucophyt, gilt als einer der ursprünglichsten Organismen, die einen Chloroplasten enthalten.[34][24]
Eine weitere primäre Endosymbiose (Paulinella)
[Bearbeiten | Quelltext bearbeiten]Die Ausnahme ist die erwähnte amöboide Paulinella chromatophora (Euglyphida, s. u.). Diese stammt offenbar von einem Vorfahren ab, der unabhängig davon und viel später – vor etwa 90 bis 500 Millionen Jahren – ein Cyanobakterium der Gattung Prochlorococcus (oder Synechococcus) aufgenommen hatte.[35][33][20][36]
Abstammungslinien und Grundtypen der Chloroplasten
[Bearbeiten | Quelltext bearbeiten]
Insgesamt gehören alle primären Chloroplasten zu einer der folgenden vier Linien (die ersten drei mit gemeinsamem Ursprung):[37][10]
- die Glaucophyten-Linie:
die einzelligen Algen der Glaucophyta (syn. Glaucocystaceae) besitzen Plastiden, die dem Cyanobakterium in vielerlei Hinsicht noch sehr ähnlich sind und daher oft als „Cyanellen“ (cyanobkterielle Organellen) oder „Cyanoplasten“, gelegentlich auch als „Muroplasten“[38] bezeichnet werden.[10] - die Rhodophyten-Linie:
Rotalgen (wiss. Rhodophyta) besitzen „Rhodoplasten“ genannte Plastiden, die noch den Antennenaufbau (Phycobilisomen) der Cyanobakterien tragen. - die Chloroplastiden-Linie:
Die Plastiden der Viridiplantae (syn. Chloroplastida, Grünalgen und höhere Pflanzen)[39] stellen die am stärksten entwickelten Plastiden dar und tragen eine große Vielfalt an Antennenkomplexen. Die grünen Plastiden der Algen und höheren Pflanzen werden Chloroplasten genannt. - die Paulinella-Linie:
Die Plastiden der amöboiden Paulinella chromatophora (Euglyphida) nennt man „Chromatophoren“, manchmal wegen ihrer Ursprünglichkeit ebenfalls „Cyanellen“.
Sekundäre und weitere Endosymbiosen
[Bearbeiten | Quelltext bearbeiten]
Auch wenn von der einen Ausnahme abgesehen wahrscheinlich alle Chloroplasten auf ein einziges derartiges primäres Endosymbiose-Ereignis zurückgeführt werden können, finden sich Chloroplasten heute dennoch in einer extrem breiten Reihe von Organismen, von denen einige nicht einmal direkt miteinander verwandt sind. Dies wird als eine Folge vieler sekundärer und sogar tertiärer endosymbiotischer Ereignisse erklärt, bei denen anstelle von Cyanobakterien photosynthetisch aktive Algen (inklusive ihres oder ihrer Chloroplasten) aufgenommen wurden. Diese ursprünglich eukaryotischen komplexen Plastiden bzw. Chloroplasten werden als sekundäre Plastiden (Chloroplasten) bezeichnet.[38]

Eine eukaryotische Alge wird einverleibt von einem anderen Eukaryoten und damit zu einem Chloroplasten mit drei oder vier Membranen.
Anzahl der Membranen und Nucleomorph
[Bearbeiten | Quelltext bearbeiten]Während primäre Chloroplasten lediglich eine Doppelmembran haben, die von ihrem cyanobakteriellen Vorfahren abstammt, finden sich bei sekundären Chloroplasten zusätzliche Membranen außerhalb dieser beiden ursprünglichen. Dies wird als eine Folge des sekundären endosymbiotischen Ereignisse gedeutet.[18][40] Infolge des meist sehr weit gehenden Abbaus der einverleibten Alge blieben oft nur ihr (primärer) Chloroplast und manchmal ihre Zellmembran und/oder gar ein Rest ihres Zellkerns, Nucleomorph genannt, übrig. Die so entstandenen komplexen (sekundären) Chloroplasten können daher drei oder vier Membranen aufweisen:[41] die beiden Cyanobakterienmembranen, manchmal die Zellmembran der inkorporierten Algen und die Phagosomenvakuole aus der Zellmembran des Wirts.[18]
Die komplexen (sekundären und tertiären) Chloroplasten wurden oft sehr vielgestaltig umgewandelt. Auch in anderen Fällen – wie etwa bei den Apicomplexa (s. u.) – machen derartige weiteren Funktionen selbst photosynthetisch nicht mehr aktive Plastiden für die Zelle weiterhin unverzichtbar.
Apicomplexa
[Bearbeiten | Quelltext bearbeiten]Die Apicomplexa sind eine Untergruppe der Alveolata.[42] Zu den Apicomplexa gehört der Malariaparasit Plasmodium. Viele Apicomplexa haben einen von Rotalgen abstammenden Chloroplasten,[43][42] Apicoplast genannt, den sie von ihren Vorfahren geerbt haben. Die Apicoplasten haben alle photosynthetischen Funktionen verloren und enthalten keine photosynthetischen Pigmente oder echte Thylakoide.
Die Tatsache, dass Apicomplexa ihren nicht photosynthetischen Chloroplasten immer noch behalten haben, bezeugt, dass die Chloroplasten neben der Photosynthese wie bereits oben erwähnt weitere wichtige Funktionen ausüben – pflanzliche Chloroplasten versorgen Pflanzenzellen neben Zucker mit vielen wichtigen Stoffen und Apicoplasten unterscheiden sich darin nicht – sie synthetisieren Fettsäuren, Isopentenylpyrophosphat und Eisen-Schwefel-Cluster. Außerdem und führen sie einen Teil der Häm-Synthese (englisch heme pathway) aus.[43]
Dinoflagellaten
[Bearbeiten | Quelltext bearbeiten]In einigen Fällen wurden vorhandene Chloroplasten durch neue (komplexe) ersetzt, in anderen Fällen kamen zu bestehenden Plastiden neue hinzu, wenn sich die Aufgaben sehr stark unterschieden. Bei den Dinoflagellaten stammen die Chloroplasten meistens von Rotalgen ab und sind vom Peridinin-Typ, charakterisiert durch das Carotinoid-Pigment Peridinin zusammen mit Chlorophyll a und Chlorophyll c2.[18][44] Peridinin kommt in keiner anderen Gruppe von Chloroplasten vor[44] und haben drei (gelegentlich nur zwei) Membranen,[22] d. h. sie haben die ursprüngliche Zellmembran des Rotalgen-Endosymbionten verloren.[18][42]
Einige Dinoflagellaten wie Kryptoperidinium und Durinskia (beide Kryptoperidiniaceae, auch englisch dinotoms)[42] haben jedoch einen von Kieselalgen (syn. Diatomeen, Heterokontophyta) abstammenden tertiären Chloroplasten.[18][45][46][47] Diese Chloroplasten sind von bis zu fünf Membranen umgeben,[18] je nachdem, ob man den gesamten Diatomeen-Endosymbionten als den Chloroplasten ansieht oder nur die darin enthaltenene Rotalge als Chloroplast zählt. Der Diatomeenendosymbiont ist relativ wenig reduziert – er beinhaltet immer noch seine ursprünglichen Mitochondrien[42] und verfügt über Endoplasmatisches Retikulum, eukaryotische Ribosomen, einen Zellkern und natürlich den von Rotalgen abstammenden sekundären Chloroplasten – praktisch eine vollständige Zelle[48] – alles im Innern des Wirts.[42] Der ursprüngliche Drei-Membran-Peridinin-Chloroplast ging entweder verloren oder wurde zu einem Augenfleck umgebildet,[18][42] wie vermutlich bei Durinskia.[49]
Eine andere Darstellung über diese komplexe Evolution findet man bei Sven B. Gould et al. (2008).[10]
Kleptoplastidie
[Bearbeiten | Quelltext bearbeiten]Trotz Reduktion des Endosymbionten-Genoms durch Gentransfer auf den Wirt, einschließlich Reduktion von Nucleomorph (bis hin zum Verschwinden) und von Membranhüllen bei komplexen Plastiden, liegt immer noch eine Symbiose vor, solange der Endosymbiont vermehrungsfähig bleibt (was allerdings im Extremfall – dem kompletten Verlust des Organell-Genoms – in völliger Assimilation enden kann). Im Gegensatz dazu bezeichnet Kleptoplastidie den ‚Raub` von Chloroplasten, d. h. das Entfernen von Hülle und Kern der aufgenommenen Phototrophen (`grünen` Euzyten), so dass nur die Chloroplasten übrig bleiben, aber nicht mehr vermehrungsfähig sind. Diese so genannten Kleptoplasten müssen dann durch Neuaufnahme immer wieder ersetzt werden. Es kommt sogar vor, dass solche Räuber selbst wieder zum Opfer werden. Kleptoplastidie wird bei bestimmten Dinoflagellaten, Wimperntierchen und einigen Meeresschnecken beobachtet,[50][51][52][53] allerdings werden bei Vertretern der Gattung Elysia einige der Gene aus den Zellkernen der Nahrung auf die Schnecken übertragen, weshalb die Chloroplasten mit für sie lebenswichtigen Proteinen versorgt werden können.[54] Dies erlaubt es einigen Arten von Elysia, sich selbst zu enthaupten und aus dem Kopfteil wieder einen vollständigen Körper zu regenerieren (beobachtet 2021 bei Elysia cf. marginata und E. atroviridis).[55] Bei der Grünen Hydra wird aber eher Endosymbiose oder eine Zwischenform angenommen.
Siehe auch
[Bearbeiten | Quelltext bearbeiten]- Chemiosmotische Kopplung
- Photosynthese
- Calvin-Zyklus
- C3-Pflanze, C4-Pflanze (mit „dimorphogenen“ Chloroplasten) und CAM-Pflanze
- Endosymbiontentheorie (zum Ursprung der Chloroplasten)
- Komplexe Plastiden
- Augenfleck (insbesondere bei Chlamydomonas) und Ocelloid
- Zooxanthelle
- Kleptoplastid
- Zellkompartiment
- Chloroamyloplast
Literatur
[Bearbeiten | Quelltext bearbeiten]- Diter von Wettstein: The effect of genetic factors on the submicroscopic structures of the chloroplast. In: Journal of Ultrastructure Research, Band 3, 1959, S. 235–239 (englisch). Dazu:
- Diter von Wettstein: Genetics And The Submicroscopic Cotology of Plastids. In: Hereditas, Band 43, Nr. 2, Juli 1957, S. 303–317; doi:10.1111/j.1601-5223.1957.tb03440.x, PDF (englisch)
- Bidhan Chandra De, Jason E. Cournoyer, Yang-le Gao, Catherine L. Wallace, Stanley Bram, Angad P. Mehta: Photosynthetic directed endosymbiosis to investigate the role of bioenergetics in chloroplast function and evolution. In: Nature Communications, Band 15, Nr. 10622, 10. Dezember 2024; doi:10.1038/s41467-024-54051-1 (englisch). Dazu:
- Claudia Krapp: Urzeit-Chloroplasten dienten noch nicht der Zuckerproduktion. Chloroplasten halfen Pflanzen ursprünglich bei der Energieproduktion. Auf: scinexx.de vom 11. Dezember 2024.
Weblinks
[Bearbeiten | Quelltext bearbeiten]- Aufbau von Chloroplasten – Schaubild
- Chloroplast und Photosynthese / Energiegewinnung bei Pflanzen – Grafik / Animation
- Botanik online (Uni Hamburg): Zellen und Gewebe – Pflanzenzelle – Abschnitt „Cloroplasten“
- Chloroplasten schmuggeln Gene über Artgrenzen
- Wilfried Probst: Frühe Evolution und Symbiose, Europa-Universität Flensburg, Institut für Biologie und Sachunterricht und ihre Didaktik: §Plastiden, abgerufen am 19. April 2019
- Nadja Podbregar: Pflanzen tauschen untereinander Chloroplasten aus: Forscher entdecken neue Form des horizontalen Gentransfers bei Gefäßpflanzen, auf: scinexx.de vom 20. Januar 2021. Quelle: Max-Planck-Institut für molekulare Pflanzenphysiologie
- Ryota Aoki, Yayoi Inui, Yoji Okabe, Mayuko Sato, Noriko Takeda-Kamiya, Kiminori Toyooka, Koki Sawada, Hayato Morita, Baptiste Genot, Shinichiro Maruyama, Tatsuya Tomo, Kintake Sonoike, Sachihiro Matsunag: Incorporation of photosynthetically active algal chloroplasts in cultured mammalian cells towards photosynthesis in animals. In: Proceedings of the Japan Academy, Series B, ISSN 0386-2208, pjab.100.035; doi:10.2183/pjab.100.035, Online-ISSN 1349-2896 (englisch). Dazu:
- Nadja Podbregar: Forscher erzeugen Tierzellen mit Chloroplasten. Eingeschleuste Photosynthese-Organellen verhelfen Hamsterzellen zu schnellerem Wachstum. Auf: scinexx.de vom 4. November 2024.
Einzelnachweise
[Bearbeiten | Quelltext bearbeiten]- ↑ C. Cleveland (Hrsg.); C. Michael Hogan, S. Draggan: DNA ( vom 25. Dezember 2012 im Internet Archive), The Encyclopedia of Earth, National Council for Science and the Environment ( vom 25. Dezember 2012 im Internet Archive). Washington DC, 18. Juli 2012 (via WebArchiv)
- ↑ The Oxford Dictionary of Abbreviations. ctDNA—Dictionary definition 1998 (encyclopedia.com).
- ↑ R. Strepp u. a.: Plant nuclear gene knockout reveals a role in plastid division for the homolog of the bacterial cell division protein FtsZ, an ancestral tubulin. In: Proc. Natl. Acad. Sci. USA Band 95, Nr. 8, 1998, S. 4368–4373. PMID 9539743, PMC 22495 (freier Volltext).
- ↑ R. Reski: Rings and networks: the amazing complexity of FtsZ in chloroplasts. In: Trends in Plant Science. Band 7, Nr. 3, 2002, S. 103–105. PMID 11906832; doi:10.1016/S1360-1385(02)02232-X.
- ↑ J. Kiessling u. a.: Visualization of a cytoskeleton-like FtsZ network in chloroplasts. In: Journal of Cell Biology. Band 151, Nr. 4, 2000, S. 945–950. PMID 11076976, PMC 2169431 (freier Volltext).
- ↑ a b Leighton Dann: Bioscience—Explained: Green DNA - Simple isolation, restriction and electrophoresis of chloroplast DNA. BIOSCIENCE EXPLAINED, Science and Plants for Schools, Homerton College, Cambridge 2002 (archive.org [PDF]). (via WebArchiv)
- ↑ Chloroplasts and Other Plastids. University of Hamburg, archiviert vom (nicht mehr online verfügbar) am 25. September 2012; abgerufen am 16. März 2019. (via WebArchiv)
- ↑ Anna Stina Sandelius, Henrik Aronsson: The Chloroplast: Interactions with the Environment. Springer, 2009, ISBN 978-3-540-68696-5, S. 18 (google.com).
- ↑ Andreas Franz Wilhelm Schimper: Über die Entwicklung der Chlorophyllkörner und Farbkörper. In: Botanische Zeitung. Band 41, Nr. 7, 1883, S. 105–114, 121–131, 137–146, 153–162 (archive.org). (via WebArchiv)
- ↑ a b c d Sven B. Gould, Ross F. Waller, Geoffrey I. McFadden: Plastid Evolution ( vom 21. Januar 2022 im Internet Archive), in: Annu. Rev. Plant Biol., Band 59, 2008, S. 491–517, doi:10.1146/annurev.arplant.59.032607.092915. Siehe insbes. Fig. 1
- ↑ Constantin S. Mereschkowsky: Über Natur und Ursprung der Chromatophoren im Pflanzenreiche. In: Biologisches Centralblatt. Band 25, 15. September 1905, S. 593–604 (archive.org). (via WebArchiv)
- ↑ Ivan E. Wallin: On the nature of mitochondria. I. Observations on mitochondria staining methods applied to bacteria. II. Reactions of bacteria to chemical treatment. In: American Journal of Anatomy. Band 30, Nr. 2, 1922, S. 203–229, doi:10.1002/aja.1000300203.
- ↑ Ivan E. Wallin: On the nature of mitochondria. III. The demonstration of mitochondria by bacteriological methods. IV. A comparative study of the morphogenesis of root-nodule bacteria and chloroplasts. In: American Journal of Anatomy. Band 30, Nr. 4, 1922, S. 451–471, doi:10.1002/aja.1000300404.
- ↑ Lynn Sagan: On the origin of mitosing cells. In: J. Theoretical Biology. Bd. 14, Nr. 3, 1967, S. 255–274. PMID 11541392 doi:10.1016/0022-5193(67)90079-3
- ↑ Bernhard Kegel: Die Herrscher der Welt: Wie Mikroben unser Leben bestimmen. DuMont, Köln 2015, ISBN 978-3-8321-9773-5.
- ↑ a b Biology, 8th Edition, Campbell & Reece. Benjamin Cummings (Pearson Education), 2009, ISBN 978-0-321-54325-7, S. 516.
- ↑ John M. Archibald: The Puzzle of Plastid Evolution. In: Current Biology. Band 19, Nr. 2, 2009, S. R81–R88, doi:10.1016/j.cub.2008.11.067, PMID 19174147.
- ↑ a b c d e f g h Patrick J. Keeling: Diversity and evolutionary history of plastids and their hosts. In: American Journal of Botany. Band 91, Nr. 10, 2004, S. 1481–1493, doi:10.3732/ajb.91.10.1481, PMID 21652304.
- ↑ a b Geoffrey I. McFadden, Giel G. Van Dooren: Evolution: Red Algal Genome Affirms a Common Origin of All Plastids. In: Current Biology. Band 14, Nr. 13, 2004, S. R514–R516, doi:10.1016/j.cub.2004.06.041, PMID 15242632.
- ↑ a b c Patricia Sánchez-Baracaldo, John A. Raven, Davide Pisani, Andrew H. Knoll: Early photosynthetic eukaryotes inhabited low-salinity habitats. In: Proceedings of the National Academy of Sciences. Band 114, Nr. 37, 12. September 2017, S. E7737–E7745, doi:10.1073/pnas.1620089114 (pnas.org).
- ↑ Ron Milo, Rob Philips: Cell Biology by the Numbers: How large are chloroplasts? In: book.bionumbers.org. Abgerufen am 16. März 2019.
- ↑ a b Anna Stina Sandelius, Henrik Aronsson (Hrsg.): The Chloroplast (= Plant Cell Monographs. Band 13). 2009, ISBN 978-3-540-68692-7, Diversity and Evolution of Plastids and Their Genomes, S. 1–39, doi:10.1007/978-3-540-68696-5_1.
- ↑ William Martin, Tamas Rujan, Erik Richly, Andrea Hansen, Sabine Cornelsen, Thomas Lins, Dario Leister, Bettina Stoebe, Masami Hasegawa, David Penny: Evolutionary analysis of Arabidopsis, cyanobacterial, and chloroplast genomes reveals plastid phylogeny and thousands of cyanobacterial genes in the nucleus. In: Proceedings of the National Academy of Sciences. Band 99, Nr. 19, 2002, S. 12246–12251, doi:10.1073/pnas.182432999, PMID 12218172, PMC 129430 (freier Volltext), bibcode:2002PNAS...9912246M.
- ↑ a b M. T. Clegg, B. S. Gaut, G. H. Learn Jr, B. R. Morton: Rates and Patterns of Chloroplast DNA Evolution. In: Proceedings of the National Academy of Sciences. Band 91, Nr. 15, 1994, S. 6795–6801, doi:10.1073/pnas.91.15.6795, PMID 8041699, PMC 44285 (freier Volltext), bibcode:1994PNAS...91.6795C.
- ↑ Chun Y. Huang, Michael A. Ayliffe, Jeremy N. Timmis: Direct measurement of the transfer rate of chloroplast DNA into the nucleus. In: Nature. Band 422, Nr. 6927, 2003, S. 72–76, doi:10.1038/nature01435, PMID 12594458, bibcode:2003Natur.422...72H.
- ↑ DNA Jumps Directly from the Cell’s Chloroplasts into its Nucleus, auf: SciTechDaily vom 16. April 2012.
- ↑ Direkter Transfer von Pflanzen-Genen aus Chloroplasten in den Zellkern, Pressemitteilung der MPG (MPI für molekulare Pflanzenphysiologie) vom 13. April 2012.
- ↑ David Roy Smith, Robert W. Lee: A Plastid without a Genome: Evidence from the Nonphotosynthetic Green Algal Genus Polytomella. In: Plant Physiology. Band 164, Nr. 4, 1. April 2014, S. 1812–1819, doi:10.1104/pp.113.233718, PMID 24563281, PMC 3982744 (freier Volltext).
- ↑ Jake Buehler: A reeking, parasitic plant lost its body and much of its genetic blueprint, auf: ScienceNews vom 10. Februar 2021.
- ↑ Christie Wilcox: Das Geheimnis der parasitischen Riesenblumen. Auf. spektrum.de vom 19. Juli 2022. Quelle: DNA of Giant ‘Corpse Flower’ Parasite Surprises Biologists. Auf: Quanta Magazine vom 21. April 2021.
- ↑ Rafael I. Ponce-Toledo, Philippe Deschamps, Purificación López-García, Yvan Zivanovic, Karim Benzerara, David Moreira: An Early-Branching Freshwater Cyanobacterium at the Origin of Plastids. In: Current Biology. Band 27, Nr. 3, 2017, S. 386–391, doi:10.1016/j.cub.2016.11.056 (elsevier.com).
- ↑ Jan de Vries, John M. Archibald: Endosymbiosis: Did Plastids Evolve from a Freshwater Cyanobacterium? In: Current Biology. Band 27, Nr. 3, 2017, S. R103–R105, doi:10.1016/j.cub.2016.12.006 (elsevier.com).
- ↑ a b Purificación López-García, Laura Eme, David Moreira: Symbiosis in eukaryotic evolution. In: Journal of Theoretical Biology. Band 434, 2017, S. 20–33, doi:10.1016/j.jtbi.2017.02.031 (elsevier.com).
- ↑ Geoffrey I. McFadden: Chloroplast Origin and Integration. In: Plant Physiology. Band 125, Nr. 1, 2001, S. 50–53, doi:10.1104/pp.125.1.50, PMID 11154294, PMC 1539323 (freier Volltext).
- ↑ Luis Delaye, Cecilio Valadez-Cano, Bernardo Pérez-Zamorano: How Really Ancient Is Paulinella Chromatophora? In: PLoS Currents. 2016, doi:10.1371/currents.tol.e68a099364bb1a1e129a17b4e06b0c6b (plos.org).
- ↑ W. Probst: Auf dem Weg zur Chloroplastenbildung. Europa-Universität Flensburg.
- ↑ S. Ball, C. Colleoni, U. Cenci, J. N. Raj, C. Tirtiaux: The evolution of glycogen and starch metabolism in eukaryotes gives molecular clues to understand the establishment of plastid endosymbiosis. In: Journal of Experimental Botany. Band 62, Nr. 6, 2011, S. 1775–1801, doi:10.1093/jxb/erq411, PMID 21220783.
- ↑ a b Robert R. Wise, J. Kenneth Hoober (Hrsg.): The structure and function of plastids. Springer, Dordrecht 2006, ISBN 1-4020-4061-X, S. 3–21 (uwosh.edu).
- ↑ genauer: Chlorophyta und Streptophyta/Charophyta, letztere mit den Landpflanzen, wissenschaftlich Embryophyta
- ↑ Jacques Joyard, Maryse A. Block, Roland Douce: Molecular aspects of plastid envelope biochemistry. In: Eur. J. Biochem. Band 199, Nr. 3, 1991, S. 489–509, doi:10.1111/j.1432-1033.1991.tb16148.x, PMID 1868841.
- ↑ Balbir K. Chaal, Beverley R. Green: Protein import pathways in 'complex' chloroplasts derived from secondary endosymbiosis involving a red algal ancestor. In: Plant Molecular Biology. Band 57, Nr. 3, Februar 2005, S. 333–342, doi:10.1007/s11103-004-7848-y, PMID 15830125.
- ↑ a b c d e f g Patrick J. Keeling: The endosymbiotic origin, diversification and fate of plastids. In: Philosophical Transactions of the Royal Society B: Biological Sciences. Band 365, Nr. 1541, 2010, S. 729–748, doi:10.1098/rstb.2009.0103, PMID 20124341, PMC 2817223 (freier Volltext).
- ↑ a b Sethu C. Nair, Boris Striepen: What Do Human Parasites Do with a Chloroplast Anyway? In: PLoS Biology. Band 9, Nr. 8, 2011, Artikel e1001137, doi:10.1371/journal.pbio.1001137, PMID 21912515, PMC 3166169 (freier Volltext).
- ↑ a b Jeremiah D. Hackett, Donald M. Anderson, Deana L. Erdner, Debashish Bhattacharya: Dinoflagellates: A remarkable evolutionary experiment. In: American Journal of Botany. Band 91, Nr. 10, 2004, S. 1523–1534, doi:10.3732/ajb.91.10.1523, PMID 21652307.
- ↑ A. Žerdoner Čalasan, J. Kretschmann, M. Gottschling: Absence of co-phylogeny indicates repeated diatom capture in dinophytes hosting a tertiary endosymbiont. In: Organisms Diversity & Evolution. Band 18, Nr. 1, März 2018, S. 29–38, doi:10.1007/s13127-017-0348-0.
- ↑ Jacques Joyard, Maryse A. Block, Roland Douce: Molecular aspects of plastid envelope biochemistry. In: Eur. J. Biochem. Band 199, Nr. 3, 1991, S. 489–509, doi:10.1111/j.1432-1033.1991.tb16148.x, PMID 1868841.
- ↑ Chloroplast. In: Encyclopedia of Science. Abgerufen am 20. März 2019.
- ↑ Eberhard Schnepf, Malte Elbrächter: Dinophyte chloroplasts and phylogeny – A review. In: Grana. Band 38, Nr. 2–3, 1999, S. 81–97, doi:10.1080/00173139908559217.
- ↑ E. Hehenberger, B. Imanian, F. Burki, P. J. Keeling: Evidence for the retention of two evolutionary distinct plastids in dinoflagellates with diatom endosymbionts. In: Genome Biology and Evolution. Band 6, Nr. 9, 2014, S. 2321–2334. doi:10.1093/gbe/evu182
- ↑ Alf Skovgaard: Role of chloroplast retention in a marine dinoflagellate. In: Aquatic Microbial Ecology. Band 15, 1998, S. 293–301, doi:10.3354/ame015293.
- ↑ Richard G. Dorrell, Christopher J. Howe: Integration of plastids with their hosts: Lessons learned from dinoflagellates. In: Proceedings of the National Academy of Sciences. Band 112, Nr. 33, 18. August 2015, ISSN 0027-8424, S. 10247–10254, doi:10.1073/pnas.1421380112, PMID 25995366, PMC 4547248 (freier Volltext), bibcode:2015PNAS..11210247D (englisch, pnas.org).
- ↑ Matthew D. Johnson, David Oldach, Charles F. Delwiche, Diane K. Stoecker: Retention of transcriptionally active cryptophyte nuclei by the ciliate Myrionecta rubra. In: Nature. Band 445, Nr. 7126, Januar 2007, S. 426–428, doi:10.1038/nature05496, PMID 17251979.
- ↑ Aditee Mitra: Meeresbiologie – Das Beste aus zwei Welten. In: Spektrum der Wissenschaft. April 2019, S. 54–60.
- ↑ M. E. Rumpho, J. M. Worful, J. Lee, K. Kannan, M. S. Tyler, D. Bhattacharya, A. Moustafa, J. R. Manhart: Horizontal gene transfer of the algal nuclear gene psbO to the photosynthetic sea slug Elysia chlorotica. In: Proceedings of the National Academy of Sciences of the United States of America. Band 105, Nr. 46, November 2008, S. 17867–17871, doi:10.1073/pnas.0804968105, PMID 19004808, PMC 2584685 (freier Volltext), bibcode:2008PNAS..10517867R.
- ↑ Sayaka Mitoh, Yoichi Yusa: Extreme autotomy and whole-body regeneration in photosynthetic sea slugs. In: CellPress: Current Biology. Band 31, Nr. 5, 8. März 2021, S. PR233-R234, doi:10.1016/j.cub.2021.01.014. Dazu:
- Michelle Starr: These Self-Decapitating Sea Slugs Can Grow an Entire New Body on The Old Head, auf: sciencealert, 8. März 2021 (englisch)
- Martin Vieweg: Skurril: Körper-Regeneration nach Selbst-Enthauptung, auf: wissenschaft.de vom 8. März 2021 (deutsch)